Jedním z přístupů k analýze genové exprese je měření koncentrace mRNA genu. Taková analýza má několik úskalí, jako jsou rozdíly v poločasu rozpadu různých transkriptů, časové vzorce transkripce a nedostatečná korelace mezi mRNA a proteinem. K analýze RNA pomocí metody založené na PCR je třeba vytvořit cDNA pomocí reverzní transkripce (RT). Tento proces využívá enzym reverzní transkriptázu a dNTP.
Krok RT může být proveden na celkové RNA, takže se vytvoří globální cDNA reprezentující mnoho transkriptů (obvykle pomocí dvoukrokového protokolu), nebo v přístupu specifickém pro daný gen, kdy se na cDNA převede pouze RNA, která je předmětem zájmu (obvykle podle jednokrokového protokolu).
Protože bylo prokázáno, že dvoukrokové reakce RT nejsou vždy lineární s ohledem na výtěžnost vstupní RNA a cDNA1, je důležité stanovit a kontrolovat celkové množství RNA extrahované a zahrnuté do reakcí RT. Měření koncentrace RNA představuje nejistotu a absolutní hodnota závisí na přístroji nebo systému použitém k měření. Jak je vidět na obrázku 8.1, existuje velká variabilita mezi měřeními koncentrace 5 vzorků RNA (A-E) (viz Purifikace vzorků a hodnocení kvality) při použití Nanodropu, konvenční UV spektrofotometrie, Ribogreenu, bioanalyzátoru Agilent 2100 nebo přístroje Bio-Rad Experion. Všimněte si, že přítomnost EDTA (vzorky D a E), která by inhibovala následnou RT a PCR, a degradace (vzorek C) mají různý vliv na měření koncentrace v závislosti na tom, který systém je použit. Toto pozorování ilustruje důležitost provedení dalších kroků kontroly kvality před použitím vzorků v navazujících reakcích (viz Purifikace vzorků a hodnocení kvality).

Obrázek 8.1.Koncentrace pěti vzorků celkové RNA (A-E) byla měřena pomocí Nanodropu, konvenční UV spektrofotometrie, Bio-Rad Experion, Agilent 2100 BioAnalyzer (všechna měření duplicitně) nebo Ribogreen (jedno měření). Jak je vidět, absolutní koncentrace i relativní koncentrace se u jednotlivých vzorků lišily. Důvodem bylo, že vzorek C byl degradovaný a vzorky D a E obsahovaly EDTA, což způsobilo nepřesná měření v systémech Experion a BioAnalyzer.
Linearita zpětného přepisu
Relativní koncentrace celkové RNA může ovlivnit účinnost RT a koncentraci cDNA vytvořené z daného transkriptu. Proto je žádoucí do všech reakcí dvoukrokové syntézy cDNA zahrnout stejnou nebo velmi podobnou koncentraci RNA, pokud není ověřeno, že systém RT má lineární odezvu. Jak je vidět na obrázku 8.2, při použití konvenčního protokolu RT nevedou stonásobná ředění vstupní RNA k odpovídajícímu stonásobnému rozdílu ve výtěžku cDNA pro testované templáty. Zajímavé je, že prezentované údaje jsou duplicitní qPCR provedené na duplicitních reakcích RT. Jak je vidět, nedostatek linearity je reprodukovatelný mezi oběma reakcemi RT.

Obrázek 8.2.Celková RNA byla naředěna 100krát a reverzně přepsána pomocí dvoukrokového náhodného primingu; byly provedeny dvě nezávislé RT reakce. β-aktin byl detekován v duplicitních qPCR pro každou RT reakci. RT je reprodukovatelná, ale výtěžek cDNA není úměrný koncentraci vstupní RNA. Pokud tedy experimentální omezení vyžadují, aby byla do RT zahrnuta proměnlivá koncentrace RNA, je rozhodující ověřit, zda protokol a kombinace činidel vedou k lineární odezvě.
V příkladu uvedeném na obrázku 8.3 bylo použito ReadyScript® RT činidlo (RDRT) k reverzní transkripci celkové RNA z 2-násobného a 10-násobného sériového ředění templátu pomocí dvoukrokového protokolu a kombinace oligo-dT (O4387) a náhodného primingu (popsáno níže). Gen CANX byl detekován v obou sériích ředění s přímou úměrností ke koncentraci vstupní RNA.

Obrázek 8.3.K reverzní transkripci celkové RNA z 2násobného a 10násobného sériového ředění bylo použito činidlo ReadyScript® RT (RDRT). Gen CANX byl detekován v obou sériích ředění, což vedlo k přímé úměrnosti s koncentrací vstupní RNA (údaje od skupin studentů, kteří se zúčastnili semináře EMBL Advanced qPCR Workshop).
Priming pro reverzní transkripci
Výběr primerů, které se použijí pro zahájení reverzní transkripce, může výrazně ovlivnit výsledky RT-qPCR. Pro jednokrokovou
RT-qPCR se používají primery specifické pro daný gen. Při provádění dvoukrokového testu lze použít reverzní primer specifický pro gen, oligo-dT (O4387), náhodné hexamery, nonamery, dekamery, dodekamery nebo pentadekamery2 nebo kombinaci oligo-dT (O4387) a náhodných primerů. Primování specifické pro daný gen se obvykle provádí v samostatných reakcích pro každou cílovou RNA. Tyto oddělené reakce mohou mít velmi rozdílnou účinnost, což komplikuje porovnávání koncentrací RNA. Na druhou stranu, při použití genově specifických primerů bude celý produkt RT kódovat gen zájmu a může umožnit kvantifikaci mRNA s velmi nízkou abundancí, které by nemohly být detekovány pomocí nespecifických primerů RT. Aby se předešlo potenciálně vysokým odchylkám mezi jednotlivými testy RT, které se mohou vyskytnout u genově specifických primerů, mohou být k vytvoření souboru cDNA použity nespecifické primery. Následně lze provést samostatné qPCR testy pro každý cíl s alikvotami ze souboru cDNA. Pokud se všechny cíle qPCR nacházejí v blízkosti 3'-konce polyadenylované mRNA, je vhodným primerem oligo-dT (O4387). Na druhou stranu, pokud jsou cíle qPCR vzdáleny více než několik kilobází od 3'-konce nebo pokud RNA není polyadenylovaná, spolehlivější detekci zajistí náhodné primery. Pokud se relativní 3' umístění cílů qPCR liší nebo požadované transkripty obsahují kombinaci polyadenylované a nepolyadenylované RNA, směs oligo-dT (O4387) a náhodných oligomerů poskytne nejlepší výsledky
Primování reverzní transkripce pro dvoukrokové reakce RT
Pro dvoukrokové primování RT se běžně používají dva přístupy; oligo-dT (O4387) a náhodné primování. Metoda oligo-dT (O4387) spočívá v hybridizaci oligodT (obvykle 15merů) na poly-A ocásek, který je přítomen na 3' konci většiny molekul mRNA, za účelem primování a selektivní reverzní transkripce mRNA.
Tento přístup je sice koncepčně velmi jednoduchý, ale jsou s ním spojeny problémy: Při reakčních teplotách používaných pro RT není vazba primeru oligo-dT (O4387) specifická pro mRNA, a proto se oligo-dT (O4387) bude nespecificky vázat na jiné oblasti RNA. Kromě toho jsou detekovány také úseky rRNA, protože ATrich oblasti v těchto molekulách jsou primerovány oligo-dT (O4387). Některé mRNA, například kódující histony, neobsahují polyA ocásky a nemusí být ve výsledné cDNA zastoupeny.
Druhá metoda využívá náhodný priming. Náhodné primery se skládají z náhodných sekvencí, často z hexamerů (6merů) nebo nonamerů (9merů). Ty se používají k primování RT reakce, což vede k syntéze různě dlouhých fragmentů cDNA, které představují původní RNA. Náhodné primery hybridizují po celé délce transkriptu a bývají tolerantnější k sekundární struktuře než oligo-dT (O4387) nebo genově specifické primery.
Aby bylo možné využít výhod příslušných technik, vyžadují některé protokoly kombinaci obou typů primerů. Specifický primer k cílové sekvenci může být použit také ve dvoukrokovém protokolu RT, ale častěji se používá v jednokrokovém postupu (viz níže).
Primery pro reverzní transkripci u jednokrokových reakcí RT
V jednokrokovém protokolu RT se k reverzní transkripci jednoho cíle používají primery specifické pro daný gen. Návrh genově specifického primeru je kritický; musí ležet v otevřené, přístupné oblasti cílové mRNA při predikci teploty reakce RT. Za těchto podmínek existuje lineární vztah mezi vstupní RNA a cDNA (obrázek 8.4). Tento primer může být (a obvykle je) společný s primerem PCR.

Obrázek 8.4.Celková RNA byla naředěna sériovým desetinásobným ředěním a k detekci GAPDH byla provedena jednostupňová RT-qPCR. Každá reakce byla provedena ve třech opakováních. Výsledkem byl lineární vztah mezi koncentrací RNA a výtěžkem cDNA (obrázek s laskavým svolením Prof. Stephena Bustina, Anglia Ruskin University, UK).
Reverzní transkripce je vysoce variabilní proces a je třeba vzít v úvahu všechny kroky, aby složky reakce byly co nejkonstantnější. Provedení jednokrokové reakce RT-qPCR sníží počet potřebných pipetovacích kroků a sníží chybovost. Proto může být metodou volby, pokud je třeba měřit malé rozdíly a přesnost je prvořadá. Opačnou úvahou však je, že stanovení poměru zájmového genu (GOI) k referenčnímu genu (viz Analýza dat) vyžaduje dvě samostatné jednokrokové RT reakce namísto jedné cDNA z dvoukrokové reakce a tyto dva cíle nelze detekovat pomocí multiplexního přístupu qPCR.
Účinnost reverzní transkripce
Obvykle se předpokládá, že veškerá RNA/mRNA v reakci RT je převedena na cDNA a že všechny transkripty jsou převedeny v 1:1 nebo úměrně výchozí koncentraci RNA. Nedávno byly provedeny studie, které zkoumaly každý z těchto předpokladů. Je zřejmé, že množství RNA, které se přemění na cDNA, je velmi variabilní. Dvoustupňový proces RT je variabilní a specificky závisí na koncentraci RNA, enzymu, složení pufru a protokolu primingu. Protože je proces variabilní, je důležité zachovat co nejvíce konstantních podmínek1,3,5.
Pro dvoukrokové reakce RT je obecně nutné usilovat o stejnou koncentraci vstupní RNA a také udržovat konstantní podmínky primingu, enzymu RT a pufru. Pokud nelze stanovit konstantní vstupní koncentraci, doporučuje se použít jednokrokový proces, zahrnout nosič, jako je polyetylenglykol (PEG)6, nebo zvolit komerční soupravu, u které bylo ověřeno, že vede k lineární reakci, jako je ReadyScript® RT.
Výběr strategie primingu ovlivňuje jak absolutní výtěžnost, tak poměr různých cílů cDNA z celkového vzorku RNA. Obrázek 8.5 ukazuje výtěžnost počtu kopií cDNA pro tři různé geny (znázorněno barvou histogramu) ve vzorku RNA, který byl rovněž podroben řízené, enzymatické degradaci. Je zřejmé, že různé metody primingu vedou k různým absolutním počtům kopií a poměrům mezi vzorky. Kromě toho se zdá, že genově specifický priming je tolerantnější k degradaci RNA než oligo-dT priming.

Obrázek 8.5.Celková RNA byla inkubována na nahé lidské ruce po uvedenou dobu (1, 2,5, 5 a 7,5 min). cDNA byla připravena pomocí genově specifického, náhodného nebo oligo-dT (O4387) primingu. Byl stanoven počet kopií tří genů, jak ukazují fialové, tyrkysové a oranžové histogramy. Je zřejmé, že různé strategie primingu ovlivňují detekci jednotlivých genů, přičemž oligo-dT (O4387) priming vede k tomu, že není detekován gen 3 (oranžový), který je ve vzorku RNA jasně patrný, protože je detekován pomocí genově specifického primingu (údaje poskytl Prof. Stephen Bustin, Anglia Ruskin University, UK).
Teplota použitá pro RT reakce může ovlivnit specifičnost, zejména při hybridizaci genově specifických primerů. Primery, které mohou tvořit silný 3'-duplex, budou snadněji hybridizovat při nižších teplotách. Vzhledem k tomu, že enzymy RT mohou z primeru DNA na templát DNA vystupovat, může se během kroku RT začít tvořit primer-dimer. Zvýšení teploty inkubace RT na nejvyšší teplotu, při které je enzym plně aktivní, nebo použití vysokoteplotního enzymu může snížit množství produktu primer-dimeru. Primery použité na obrázku 8.6 vedly k významně většímu množství specifického produktu ve srovnání s nespecifickým produktem po jednokrokovém RT-qPCR, když byla RT provedena s Moloney Murine Leukemia Virus-Reverse Transcriptase (MMLV-RT) při 45 °C (obrázek 8.6).6B) než při RT prováděné při 37 °C (obrázek 8.6A). Podobně při provádění dvoukrokové RT-qPCR s nespecifickým primerem pro RT a polymerázou Hot Start Taq pro qPCR vzniklo méně produktu primer-dimer (obrázek 8.6D) než při použití jednostupňové RT-qPCR s primery specifickými pro daný gen, které mohly tvořit 3'-duplex (obrázek 8.6B).
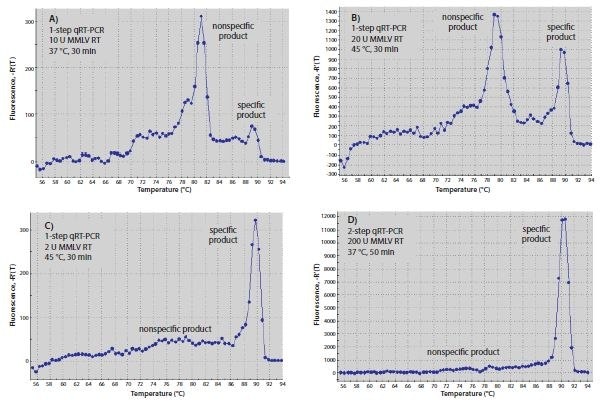
Obrázek 8.6.Optimalizace RT. (A-C) Křivky tání produktů RT-qPCR získaných pomocí jednokrokové nebo (D) dvoukrokové RT-qPCR. Reakce A-C obsahovaly vždy 10 μl směsi SYBR® Green JumpStart™ Taq ReadyMix™, 0,02 μl referenčního barviva, oba genově specifické primery o koncentraci 0,4 μM a 10 ng lidské celkové RNA v konečném objemu 20 μl. Genově specifické primery byly 5'-CGGGCTTCAACGCAGACTA-3´a 5'-CTGGTCGAGATGGCAGTGA-3´ pro c-fos (Accession NM_005252). Reakce A a B obsahovaly také 20 jednotek MMLV- RT, zatímco reakce C obsahovala 2 jednotky. Reakce A byla před qPCR inkubována při 37 °C po dobu 30 minut, zatímco reakce B a C byly před qPCR inkubovány při 45 °C po dobu 30 minut. V reakci D obsahovala RT reakce 1x MMLV pufr (produkt č. D8559), 0,5 mM dNTPs, 1 μM oligo-dT (O4387), 0,8 jednotek/μl inhibitoru RNázy, 200 jednotek MMLV-RT a 10 ng lidské celkové RNA v konečném objemu 20 μl. Reakce byla inkubována při 25 °C po dobu 10 min, 37 °C po dobu 50 min a 80 °C po dobu 10 min. 2 μl produktu RT reakce byly přidány do qPCR obsahující 10 μl SYBR® Green JumpStart™ Taq ReadyMix™ 0,02 μl referenčního barviva a oba genově specifické primery v koncentraci 0,4 μM jako u jednokrokových reakcí (A-C). Všechny reakce qPCR byly inkubovány při 94 °C po dobu 3 minut pro denaturaci a poté 40 cyklů při 94 °C po dobu 15 sekund a 60 °C po dobu 1 minuty.
Výsledky RT-qPCR může ovlivnit také množství enzymu RT na reakci. Jak ukazuje obrázek 8.6, jednokrokové reakce s 2 jednotkami MMLV-RT (obrázek 8.6C) byly specifičtější než reakce s 20 jednotkami (obrázek 8.6B). Dvoustupňová RT-PCR s použitím oligo-dT (O4387) nebo náhodných primerů pro RT vedla k větší specifitě než jednostupňová RT-PCR (obrázek 8.6D). To lze přičíst skutečnosti, že primery specifické pro daný gen nejsou přítomny během reakce RT při nízké teplotě, čímž se zabrání tvorbě nespecifických produktů. Vyšší koncentrace RT mohou poskytnout lepší výsledky ve dvoustupňových reakcích, ale protože enzym RT může interferovat s aktivitou Taq DNA polymerázy7, množství produktu RT přeneseného do qPCR by mělo být omezeno na maximálně 10 % konečného objemu reakce. Výjimkou z tohoto doporučení by bylo použití ReadyScriptu, ve kterém může být 25 % objemu PCR reakce RT bez ovlivnění účinnosti PCR.
Tento popis proměnných, které jsou vlastní procesu RT, ukazuje, že stanovení genové
exprese z RT závisí na použité metodě RT, množství a kvalitě vzorků, kromě zohlednění množství templátu, a mělo by být pečlivě zaznamenáno, jak je popsáno v pokynech MIQE
.sup>8 (viz Kvantitativní PCR).
Odkazy
Abyste mohli pokračovat ve čtení, přihlaste se nebo vytvořte účet.
Nemáte účet?