Fordított átírás
A Technical Guide to PCR Technologies
A génexpresszió elemzésének egyik megközelítése a gén mRNS-koncentrációjának mérése. Az ilyen elemzésnek számos kihívása van, például a különböző transzkriptumok közötti felezési időbeli különbségek, a transzkripció időbeli mintázata és az mRNS és a fehérje közötti korreláció hiánya. Az RNS PCR-alapú módszerrel történő elemzéséhez cDNS-t kell előállítani reverz transzkripció (RT) segítségével. Ez a folyamat egy reverz transzkriptáz enzimet és dNTP-ket használ.
Az RT lépés történhet teljes RNS-en, így számos transzkript globális cDNS-reprezentációja jön létre (általában kétlépéses protokoll segítségével), vagy génspecifikus megközelítésben, amikor csak az érdeklődésre számot tartó RNS-t alakítjuk át cDNS-vé (általában egylépéses protokollt követve).
Mivel bebizonyosodott, hogy a kétlépéses RT-reakció nem mindig lineáris a bemeneti RNS és a cDNS hozam1 tekintetében, fontos meghatározni és ellenőrizni az RT-reakciókban kivont és belekerült RNS teljes mennyiségét. Az RNS koncentrációjának mérése bizonytalanságot jelent, és az abszolút érték a méréshez használt műszertől vagy rendszertől függ. Amint az 8.1. ábra is látható, 5 RNS-mintán (A-E) végzett koncentrációmérések között nagy a szórás (lásd Mintatisztítás és minőségértékelés) a Nanodrop, a hagyományos UV-spektrofotometria, a Ribogreen, az Agilent 2100 BioAnalyzer vagy a Bio-Rad Experion használata esetén. Vegye figyelembe, hogy az EDTA jelenléte (D és E minta), amely gátolná a downstream RT-t és PCR-t, valamint a lebomlás (C minta) eltérő hatással van a koncentrációmérésekre attól függően, hogy melyik rendszert használja. Ez a megfigyelés jól illusztrálja a további minőségellenőrzési lépések elvégzésének fontosságát, mielőtt a mintákat a downstream reakciókban használnánk (lásd Mintatisztítás és minőségértékelés).

8.1. ábra.A teljes RNS öt mintájának (A-E) koncentrációját Nanodrop, hagyományos UV-spektrofotometria, Bio-Rad Experion, Agilent 2100 BioAnalyzer (minden duplikált mérés) vagy Ribogreen (egyszeri mérés) segítségével mértük. Amint látható, az abszolút koncentráció és a relatív koncentráció is változott a minták között. Ennek oka, hogy a C minta lebomlott, a D és E minták pedig EDTA-t tartalmaztak, ami pontatlan méréseket okozott az Experion és a BioAnalyzer rendszerekben.
A fordított átírás linearitása
A teljes RNS relatív koncentrációja befolyásolhatja az RT hatékonyságát és az adott transzkriptből előállított cDNS koncentrációját. Ezért kívánatos, hogy minden kétlépéses cDNS-szintézisreakcióba azonos vagy nagyon hasonló koncentrációjú RNS-t tegyünk, kivéve, ha az RT-rendszerről igazolták, hogy lineáris válaszreakcióval rendelkezik. Amint az 8.2. ábra látható, hagyományos RT protokollt használva a bemeneti RNS 100-szoros hígítása nem eredményez megfelelő 100-szoros különbséget a cDNS-hozamban a vizsgált sablonok esetében. Érdekes, hogy a bemutatott adatok kétszeres RT-reakciókon lefuttatott duplikált qPCR-ek. Mint látható, a linearitás hiánya reprodukálható a két RT-reakció között.

8.2. ábra.A teljes RNS-t 100-szorosára hígítottuk, és kétlépéses véletlenszerű priming alkalmazásával reverz transzkripciót végeztünk; két független RT-reakciót végeztünk. β-aktint minden RT-reakcióban kétszeres qPCR-rel detektáltunk. Az RT reprodukálható, de a cDNS-hozam nem arányos a bemeneti RNS-koncentrációval. Ezért, ha a kísérleti korlátozások megkövetelik, hogy változó RNS-koncentráció szerepeljen az RT-ben, kritikus annak ellenőrzése, hogy a protokoll és a reagensek kombinációja lineáris választ eredményez-e.
Az 8. ábrán látható példában.3, a ReadyScript® RT reagens (RDRT) segítségével reverz transzkripciót végeztünk a teljes RNS-nek a templát 2-szeres és 10-szeres sorozathígításából, kétlépéses protokoll és az oligo-dT (O4387) és a véletlenszerű priming kombinációjával (lásd alább). A CANX-gént mindkét hígítási sorozatban a bemeneti RNS-koncentrációval egyenes arányosságban detektáltuk.

8.3. ábra.A ReadyScript® RT reagens (RDRT) a 2-szeres és 10-szeres sorozathígítású teljes RNS reverz transzkripciójához szolgált. A CANX gént mindkét hígítási sorozatban kimutattuk, ami egyenes arányosságot eredményezett a bemeneti RNS-koncentrációval (az EMBL Advanced qPCR Workshopon részt vevő diákcsoportok adatai).
Reverz transzkripciós primerelés
A reverz transzkripció indításához használt primerek kiválasztása nagyban befolyásolhatja az RT-qPCR eredményeit. Az egylépéses
RT-qPCR esetében génspecifikus primereket használunk. Kétlépéses vizsgálat esetén reverz génspecifikus primer, oligo-dT (O4387), véletlenszerű hexamerek, nonamerek, dekamerek, dodekamerek vagy pentadekamerek2 vagy az oligo-dT (O4387) és véletlenszerű primerek kombinációja használható. A génspecifikus primereket általában minden egyes cél-RNS esetében külön reakcióban futtatják. Ezek a különálló reakciók nagyon eltérő hatékonyságúak lehetnek, ami megnehezíti az RNS-koncentrációk összehasonlítását. Másrészt, génspecifikus primerek használata esetén az összes RT-termék a kívánt gént kódolja, és lehetővé teheti a nagyon alacsony abundanciájú mRNS-ek mennyiségi meghatározását, amelyeket nemspecifikus RT-primerekkel nem lehetne kimutatni. A génspecifikus primerekkel előforduló, a vizsgálatok közötti potenciálisan nagy RT-variációk elkerülése érdekében nemspecifikus primereket lehet használni a cDNS-pool létrehozásához. Ezt követően a cDNS-poolból származó aliquotokkal külön qPCR-vizsgálatokat lehet végezni az egyes célpontokra. Ha minden qPCR-célpont a poliadenilált mRNS-ek 3'-végéhez közel van, az oligo-dT (O4387) megfelelő primer választás. Másrészt, ha a qPCR-célpontok néhány kilobázisnál távolabb vannak a 3'-végtől, vagy ha az RNS nem poliadenilált, a véletlenszerű primerek megbízhatóbb kimutatáshoz vezetnek. Ha a qPCR-célpontok relatív 3' helyzete változik, vagy a kívánt transzkriptek poliadenilált és nem poliadenilált RNS-ek kombinációját tartalmazzák, akkor az oligo-dT (O4387) és a random oligomerek keveréke fogja a legjobb eredményt adni
Reverse transzkripciós priming a kétlépéses RT-reakciókhoz
A kétlépéses RT priminghez általában két megközelítést használnak; oligo-dT (O4387) és random priming. Az oligo-dT (O4387) módszer egy oligodT (általában 15mer) hibridizációjára támaszkodik az mRNS molekulák többségének 3' végén található poli-A farokhoz, hogy az mRNS-t primerelje és szelektíven reverz transzkribálja.
Ez a megközelítés, bár koncepcionálisan nagyon egyszerű, kihívásokkal jár: Az oligo-dT (O4387) primer kötődése nem specifikus az mRNS-re az RT-hez használt reakcióhőmérsékleteken, így az oligo-dT (O4387) nem specifikusan kötődik az RNS más régióihoz. Ezenkívül az rRNS-ek szakaszai is kimutathatók, mivel az ezekben a molekulákban lévő ATrich régiók az oligo-dT (O4387) által alapozottak. Egyes mRNS-ek, például a hisztonokat kódoló mRNS-ek nem tartalmaznak poli A-farkat, és előfordulhat, hogy nem reprezentálódnak az így kapott cDNS-ben.
A második módszer véletlenszerű priminget használ. A véletlenszerű primerek véletlenszerű szekvenciákból állnak, gyakran hexamerekből (6mer) vagy nem-amerekből (9mer). Ezeket az RT-reakció primereléséhez használják, ami különböző hosszúságú cDNS-fragmentumok szintéziséhez vezet, amelyek az eredeti RNS-t képviselik. A véletlenszerű primerek a transzkript hosszában hibridizálnak, és általában toleránsabbak a másodlagos szerkezettel szemben, mint az oligo-dT (O4387) vagy a génspecifikus primer.
Az egyes technikák előnyeinek kihasználása érdekében egyes protokollok a két primertípus kombinációját igénylik. A célszekvenciára specifikus primer is használható kétlépéses RT protokollban, de gyakrabban használják egylépéses eljárásban (lásd alább).
Reverz transzkripciós primerek az egylépéses RT-reakciókhoz
Az egylépéses RT protokollban génspecifikus primereket használnak egyetlen célszekvencia reverz transzkripciójához. A génspecifikus primer megtervezése kritikus fontosságú; az RT-reakció hőmérsékletén előre jelezve az mRNS-célpont nyitott, hozzáférhető régiójában kell elhelyezkednie. Ilyen körülmények között lineáris kapcsolat áll fenn a bemeneti RNS és a cDNS között (8.4. ábra). Ez a primer lehet (és általában közös is) a PCR primerrel.

8.4. ábra.A teljes RNS-t egy 10-szeres hígítási sorozaton keresztül hígítottuk, és egylépéses RT-qPCR-t végeztünk a GAPDH kimutatására. Minden reakciót három példányban végeztünk. Ez lineáris összefüggést eredményezett az RNS-koncentráció és a cDNS-hozam között (a képet Prof. Stephen Bustin, Anglia Ruskin University, UK).
A reverz transzkripció rendkívül változó folyamat, és minden lépést figyelembe kell venni, hogy a reakciókomponensek a lehető legállandóbbak legyenek. Az egylépéses RT-qPCR-reakció elvégzése csökkenti a szükséges pipettázási lépések számát és csökkenti a hibákat. Ezért ez lehet a választandó módszer, ha kis különbségeket kell mérni, és a pontosság a legfontosabb. Ezzel ellentétes megfontolás azonban az, hogy az érdeklődésre számot tartó gén (GOI) és a referencia gén arányának meghatározása (lásd Adatelemzés) két külön egylépéses RT-reakciót igényel a kétlépéses reakcióból származó egyetlen cDNS helyett, és a két célpont nem mutatható ki multiplex qPCR-megközelítéssel.
A fordított transzkripció hatékonysága/span>
Az általános feltételezés szerint egy RT-reakcióban az összes RNS/mRNS átalakul cDNS-vé, és az összes transzkriptum 1:1 arányban vagy a kiindulási RNS-koncentrációval arányosan. A közelmúltban tanulmányokat végeztek e feltételezések mindegyikének vizsgálatára. Egyértelmű, hogy a cDNS-vé átalakuló RNS mennyisége nagymértékben változó. A kétlépéses RT-folyamat változó és specifikusan függ az RNS-koncentrációtól, az enzimtől, a puffer összetételétől és a priming protokolltól. Mivel a folyamat változó, fontos, hogy minél több állandó körülményt tartsunk fenn1,3,5.
A kétlépéses RT-reakciók esetében általában azonos bemeneti RNS-koncentrációra kell törekedni, valamint állandónak kell tartani a priming körülményeket, az RT enzimet és a puffert. Ha az állandó bemeneti koncentráció nem határozható meg, célszerű egylépéses eljárást alkalmazni, hordozót, például polietilénglikolt (PEG)6 tartalmazni, vagy olyan kereskedelmi kitet választani, amely validáltan lineáris választ eredményez, mint például a ReadyScript® RT.
A priming-stratégia megválasztása befolyásolja mind az abszolút hozamot, mind a különböző cDNS-célpontok arányát egy teljes RNS-mintából. A 8.5. ábra három különböző gén cDNS-kópiaszámának hozamát mutatja (a hisztogram színe mutatja) egy olyan RNS-mintában, amelyet kontrollált, enzimatikus lebontásnak is alávetettünk. Látható, hogy a különböző priming módszerek eltérő abszolút kópiaszámokat és arányokat eredményeznek a minták között. Emellett úgy tűnik, hogy a génspecifikus priming toleránsabb az RNS lebomlásával szemben, mint az oligo-dT priming.

8.5. ábra.A teljes RNS-t egy meztelen emberi kézen inkubáltuk a megadott ideig (1, 2,5, 5 és 7,5 perc). cDNS-t génspecifikus, véletlenszerű vagy oligo-dT (O4387) primerrel állítottunk elő. Három gén kópiaszámát határoztuk meg, amint azt a lila, türkiz és narancssárga színű hisztogramok jelzik. Világos, hogy a különböző priming stratégiák befolyásolják az egyes gének kimutatását, az oligo-dT (O4387) priming nem eredményezi a 3. gén (narancssárga) kimutatását, amely egyértelműen látható az RNS-mintában, mivel génspecifikus priminggel detektálták (az adatokat Prof. Stephen Bustin, Anglia Ruskin University, UK szolgáltatta).
A RT-reakciókhoz használt hőmérséklet befolyásolhatja a specificitást, különösen génspecifikus primerek hibridizációja esetén. Azok a primerek, amelyek erős 3'-duplexet képesek kialakítani, alacsonyabb hőmérsékleten könnyebben hibridizálódnak. Mivel az RT-enzimek a DNS-primerről a DNS-templátra terjedhetnek, a primer-dimerek képződése már az RT-lépés során megkezdődhet. Az RT inkubációs hőmérsékletének megemelése a legmagasabb hőmérsékletre, amelyen az enzim teljesen aktív, vagy magas hőmérsékletű enzim használata csökkentheti a primer-dimer termék mennyiségét. A 8.6. ábra használt primerek az egylépéses RT-qPCR után szignifikánsan több specifikus terméket eredményeztek a nem specifikus termékhez képest, amikor az RT-t Moloney Murine Leukémiavírus-Reverse Transcriptase (MMLV-RT) segítségével 45 °C-on végeztük (8. ábra.6B), mint amikor az RT-t 37 °C-on végezték (8.6A ábra). Hasonlóképpen, a kétlépéses RT-qPCR elvégzése egy nem specifikus primerrel az RT-hez és Hot Start Taq polimerázzal a qPCR-hez kevesebb primer-dimer terméket eredményezett (8.6D ábra), mint az egylépéses RT-qPCR alkalmazása génspecifikus primerekkel, amelyek 3'-duplexet képezhetnek (8.6B ábra).
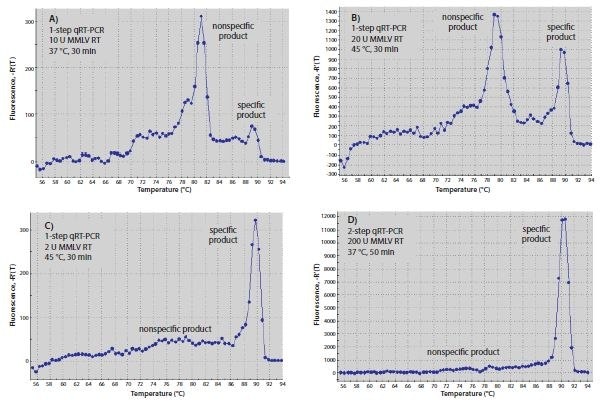
8.6. ábra.Az RT optimalizálása. (A-C) Az egylépéses vagy (D) kétlépéses RT-qPCR-rel előállított RT-qPCR-termékek olvadási görbéi. Az A-C reakciók mindegyike 10 μL SYBR® Green JumpStart™ Taq ReadyMix™-et, 0,02 μL referenciafestéket, mindkét génspecifikus primert 0,4 μM-os koncentrációban és 10 ng humán teljes RNS-t tartalmazott 20 μL végső térfogatban. A génspecifikus primerek 5'-CGGGCTTCAACGCAGCAGACTA-3´és 5´ -CTGGTCGAGATGGCAGTGA-3´ voltak a c-fos (Accession NM_005252) esetében. Az A és B reakciók 20 egység MMLV- RT-t is tartalmaztak, míg a C reakció 2 egységet. Az A reakciót 37 °C-on inkubáltuk 30 percig a qPCR előtt, míg a B és C reakciót 45 °C-on inkubáltuk 30 percig a qPCR előtt. A D reakcióban az RT-reakció 1x MMLV puffert (D8559 termékszám: D8559), 0,5 mM dNTP-t, 1 μM oligo-dT-t (O4387), 0,8 egység/μL RNáz-inhibitort, 200 egység MMLV-RT-t és 10 ng humán teljes RNS-t tartalmazott 20 μL végső térfogatban. A reakciót 10 percig 25 °C-on, 50 percig 37 °C-on és 10 percig 80 °C-on inkubáltuk. 2 μL RT-reakcióterméket adtunk a qPCR-hez, amely 10 μL SYBR® Green JumpStart™ Taq ReadyMix™ 0,02 μL referenciafestéket és mindkét génspecifikus primert 0,4 μM-ban tartalmazta, mint az egylépéses reakciók esetében (A-C). Minden qPCR-reakciót 3 percig 94 °C-on inkubáltunk denaturálás céljából, majd 40 cikluson keresztül 94 °C-on 15 másodpercig és 60 °C-on 1 percig.
A RT-qPCR-eredményeket a reakciónkénti RT-enzim mennyisége is befolyásolhatja. Amint az 8.6. ábra mutatja, a 2 egység MMLV-RT-t tartalmazó egylépéses reakciók (8.6. ábraC) specifikusabbak voltak, mint a 20 egységet tartalmazó reakciók (8.6. ábraB). A kétlépéses RT-PCR az oligo-dT (O4387) vagy véletlenszerű RT-primerek használatával nagyobb specificitást eredményezett, mint az egylépéses RT-PCR (8.6D ábra). Ez annak tulajdonítható, hogy a génspecifikus primerek nincsenek jelen az alacsony hőmérsékletű RT-reakció során, így megakadályozzák a nemspecifikus termékek képződését. A kétlépéses reakciókban a magasabb RT-koncentrációk jobb eredményeket adhatnak, de mivel az RT-enzim zavarhatja a Taq DNS-polimeráz aktivitását7, a qPCR-re átvitt RT-termék mennyiségét a végső reakciótérfogat legfeljebb 10%-ára kell korlátozni. Ez alól az ajánlás alól kivételt képez a ReadyScript használata, amelyben a PCR térfogat 25%-a lehet RT-reakció a PCR hatékonyságának befolyásolása nélkül.
Az RT-folyamatban rejlő változók leírása azt mutatja, hogy a gén
expresszió RT-ből történő meghatározása a template mennyiségének figyelembevétele mellett az alkalmazott RT-módszertől, a minták mennyiségétől és minőségétől is függ, és gondosan jelenteni kell, ahogyan azt a MIQE-irányelvek
leírják.sup>8 (lásd Kvantitatív PCR).
Hivatkozások
Az olvasás folytatásához jelentkezzen be vagy hozzon létre egy felhasználói fiókot.
Még nem rendelkezik fiókkal?