MISSION® esiRNA Specifikace
Vlastní &; předpřipravené esiRNA
Počínaje koncem října / začátkem listopadu 2020 budou produkty esiRNA, které jsou dodávány ve zkumavkách -. Individual esiRNA, esiOPEN a esiSEC - se začnou dodávat lyofilizované namísto roztoku (produkty esiRNA v destičkách - esiLibrary a esiFLEX - se budou nadále dodávat v roztoku). Tuto změnu oproti produktům ve zkumavkách (většina esiRNA se dodává ve zkumavkách) provádíme z následujících důvodů:
- Lyofilizované materiály lze dodávat při pokojové teplotě namísto zmrazených na suchém ledu. Toto snížení nákladů na přepravu nám pomůže udržet ceny produktů na co nejnižší úrovni.
- Odstraněním suchého ledu z přeprav budeme používat méně obalů, což nám zase pomůže splnit závazek naší společnosti snížit dopad na životní prostředí. Stejně jako RNA je esiRNA stabilní při pokojové teplotě po dobu nejméně 3 týdnů. Po obdržení esiRNA ji prosím uložte do -20 °C, dokud nebudete připraveni ji použít. Příloha 2 v Technickém bulletinu produktu obsahuje protokol resuspenze. Bude existovat přechodné období, ve kterém může být část esiRNA stále dodávána v roztoku a zároveň část dodávána v suchém stavu. Co nejrychleji urychlíme přechod na všechny jednotky v suché formě.
Všechny možnosti objednání a cenové nabídky jsou uvedeny v tabulce esiRNA specifikace
MISSION® esiRNA-powered by Eupheria Biotech - poskytují výzkumníkům RNAi osvědčený, nákladově efektivní a jednoduchý způsob, jak provádět posttranskripční umlčování genů kódujících proteiny a lncRNA (dlouhé nekódující RNA). Biologicky připravené esiRNA se skládají z heterogenního souboru siRNA (přírodní RNA, bez modifikací), které se všechny zaměřují na stejnou sekvenci mRNA. Tyto vícenásobné spouštěče umlčování vedou k vysoce specifickému a účinnému vyřazení genů s nižšími účinky mimo cíl než jednotlivé, chemicky syntetizované siRNA (obrázek 1).

Obrázek 1.Knockdown cílové mRNA lze provést chemicky syntetizovanou siRNA nebo enzymaticky připravenou siRNA (esiRNA). A) Chemicky syntetizovaná siRNA se skládá z jediného umlčovacího spouštěče o délce 21 bp, který je komplementární k cílové mRNA. Vysoká koncentrace siRNA v transfekční reakci vede k výrazným účinkům mimo cíl. B) Naproti tomu esiRNA se skládá z poolu stovek siRNA (21 bp), které pokrývají oblast 300 - 600 bp cílové mRNA. Každá jednotlivá siRNA má v poolu nižší koncentraci, což vede k nižším off-target efektům a zároveň k účinnému knockdownu.
Výhody produktu
- Zaručené umlčení genů
- Menší necílové účinky než u jednotlivých, chemicky syntetizovaných siRNA
- Vysoká specifičnost na cíli z něj dělá účinný primární screeningový nástroj (obrázek 2)
- Dostupný screeningový nástroj RNAi v genomovém měřítku

Obrázek 2.Hodnocení knockdownu genů v HeLa buňkách transfekovaných esiRNA namířenou proti Renilla Luciferase (negativní kontrola) a různým exprimovaným cílům na úrovni mRNA a proteinu. A) Data qPCR ověřují knockdown 24 hodin po transfekci. Množství mRNA bylo u všech transfekcí sníženo o > 90 %. B) Data z kvantitativního Western Blotu potvrzují efektivní knockdown 72 hodin po transfekci. Hladiny proteinů byly sníženy o 45 až 90 %.
Vlastnosti produktu
Následující údaje platí pro všechny možnosti produktu uvedené v tabulce specifikací esiRNA:
- Čištění: Q-Sepharose separace, srážení isopropanolem & promývání etanolem
- Sekvenční forma: IiRNA se sráží pomocí isopropanolu a etanolu:
- Kontrola kvality: Pool stovek siRNA s průměrnou délkou duplexu 21 bp
- Kontrola kvality: V případě, že siRNA není v pořádku, je nutné provést kontrolu kvality: Provádí se ve dvou fázích
o produkt PCR klonu cDNA se analyzuje pomocí gelové elektroforézy &; sekvenování DNA
o Digestační reakce se analyzuje pomocí gelové elektroforézy - Stabilita:
V případě čehokoli označeného níže jako "Dotaz" nebo pokud máte potřeby, které se liší od ostatních uvedených obecných specifikací, zašlete prosím požadavek na sirnarequest@milliporesigma.com.
Prohlédněte si kompletní seznam předem navržené esiRNA.
Kontrola esiRNA
K dispozici je řada pozitivních a negativních kontrol (obrázek 3):
- Pozitivní kontroly: Lidské Eg5 (KIF11) & myší Eg5 (Kif11) esiRNA, stejně jako všechny validované esiRNA (viz tabulka níže), aby byla zajištěna optimalizace transfekce & experimentálního nastavení
- Negativní kontroly: RLUC, FLUC & EGFP (EGFP lze také použít jako pozitivní kontrolu v buňkách exprimujících EGFP) esiRNA k rozlišení sekvenčně specifického umlčení od nespecifických účinků

Obrázek 3.Fenotypová analýza HeLa buněk transfekovaných esiRNA proti A) RLUC (negativní kontrola) a B) Eg5/KIF11 (pozitivní kontrola). RLUC nevyvolává žádné fenotypové změny, zatímco Eg5/KIF11 vyvolává mitotickou zástavu, což se projevuje zakulacenými buňkami.
Validované esiRNA
Mnoho běžných genových cílů bylo validováno pro ≥70% knockdown mRNA (qPCR a Western Blot validační data). Seznam běžně řazených validovaných esiRNA podle symbolu genu naleznete v tabulce. Validované esiRNA jsou vhodné pro optimalizaci transfekce a jako pozitivní kontroly.
lncRNA esiRNA
Analýzy transkriptomu ukazují, že až 90 % genomu je přepisováno do nekódující RNA, mezi kterou patří mimo jiné i lncRNA. S lncRNA jsou spojeny různé funkce, včetně modifikace chromatinu, koaktivace transkripčních faktorů, transkripce, interakce s proteiny vážícími RNA a represe promotorů. Kromě toho se lncRNA podílejí na vzniku onemocnění, jako je rakovina. Podrobnosti fungování lncRNA je třeba dále objasnit. EsiRNA představují účinný screeningový nástroj pro studium funkce lncRNA (obrázek 4).
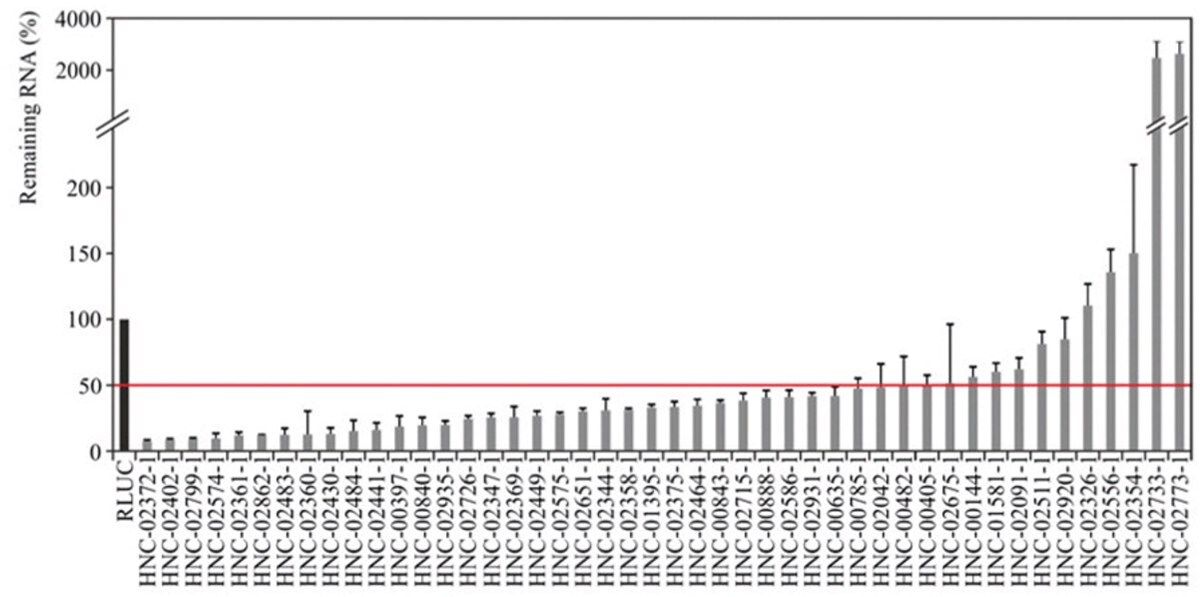
Obrázek 4.Data qPCR ověřující knockdown lncRNA v buňkách HeLa 24 hodin po transfekci odpovídající esiRNA. Kontroly byly transfekovány esiRNA proti RLUC a použity pro normalizaci.
Výroba produktu
Biologicky připravená (obrázek 5) esiRNA se vyrábí štěpením dlouhé dsRNA (dvouvláknové RNA) pomocí endoribonukleázy, E. coli RNázy III

Obrázek 5.Přehled výroby esiRNA pro jednotlivé geny. A) DEQOR se používá k výběru cílové oblasti mRNA, která vytvoří největší počet vysoce účinných siRNA, pokryje všechny známé varianty transkriptu a minimalizuje necílové účinky. B) Cílová oblast je extrahována z klonu cDNA a amplifikována pomocí PCR (oba primery jsou použity k zavedení promotorů T7. C) Produkt PCR je přepsán in vitro s využitím RNA polymerázy. D) Žíhaná a dlouhá dvouvláknová RNA (dsRNA) se štěpí RNázou III a purifikací se odstraní DNA templát, zbytky NTP a neúplně natrávená dsRNA. Nakonec je esiRNA připravena jako pool stovek siRNA s průměrnou délkou duplexu 21 bp.
esiRNA záruka
MISSION® esiRNA sníží hladiny cílové mRNA o 70 %, v kultivovaných buňkách, při transfekci v koncentraci ≥30 nM. Pokud esiRNA nesníží cílový gen o 70 %, poskytneme pro daný gen další esiRNA zdarma. Pokud další esiRNA pro daný gen nemáme, vrátíme vám kupní cenu.
Pro poskytnutí záruky je nutné obdržet příslušné podpůrné údaje o účinnosti transfekce. Vhodná podpůrná data pro účinnost transfekce by zahrnovala data qPCR porovnávající hladiny cílové mRNA validované esiRNA MISSION® transfekované v koncentraci ≥ 30 nM s vhodnou negativní kontrolou (jako je mock transfekce, RLUC, FLUC nebo eGFP esiRNA), která prokazuje knockdown cílové mRNA ve výši 70 %.
Vzhledem k variabilitě protilátek a poločasů rozpadu proteinů nemůžeme akceptovat údaje z metod detekce založených na proteinech.
Často kladené otázky
Chcete-li se o esiRNA dozvědět více, navštivte naši Často kladené otázky sekce.
Video tutoriál
Naučte se o screeningu v savčích buňkách pomocí esiRNA.
Pokud potřebujete další pomoc, obraťte se na naši skupinu technických služeb na sirnarequest@sial.com.
Dozvědět se více o RNA interferenci: od studií jednotlivých genů po celogenomové screeny - Dr. Julia Krüger Senior Scientist ve společnosti Eupheria Biotech GmbH
Výběr citací
Jednogenová esiRNA
Screeny celého genomu a dílčích knihoven
lncRNA esiRNA
Abyste mohli pokračovat ve čtení, přihlaste se nebo vytvořte účet.
Nemáte účet?