Custom & Predesigned esiRNA
從 2020 年 10 月底 / 11 月初開始,管裝的 esiRNA 產品Individual esiRNA、esiOPEN 和 esiSEC - 將開始以凍乾狀而非溶液狀出貨(esiRNA 板裝產品 - esiLibrary 和 esiFLEX - 將繼續以溶液狀出貨)。我們對試管中的產品(大多數 esiRNA 以試管裝運送)作出這項變更,原因如下:
- 凍乾的材料可以在環境溫度下運送,而不是用乾冰冷凍。
- 運送過程中不使用乾冰,我們將使用較少的包裝,這也有助於我們實現公司減少對環境影響的承諾。作為 RNA,esiRNA 在室溫下至少可穩定 3 週。收到您的 esiRNA 後,請放置於 -20 °C 儲存,直到您準備使用為止。產品技術公告中的附錄 2 包含重懸浮方案。在過渡期間,有些 esiRNA 可能仍以溶液形式寄送,有些則以乾貨寄送。我們將儘快轉換成所有乾貨。
所有訂購和報價選項請參閱 Eupheria Biotech-為 RNAi 研究人員提供一種行之有效、具成本效益且簡單的方法,以執行蛋白質編碼基因和 lncRNA(長非編碼 RNA)的轉錄後沉默。由生物製備的 esiRNA 是由異質的 siRNA (天然 RNA,未經修飾) 組成,這些 siRNA 都是針對相同的 mRNA 序列。與單一化學合成的 siRNA 相比,這些多重沉默觸發因子能以較低的脫靶(圖 1)效果,達到高度特異性和有效的基因敲除。

圖 1.可以使用化學合成的 siRNA 或酵素製備的 siRNA (esiRNA) 來敲除目標 mRNA。A) 化學合成的 siRNA 由 21 bp 的單一沉默觸發因子組成,與目標 mRNA 互補。轉染反應中高濃度的 siRNA 會導致明顯的脫靶效應。B) 相比之下,esiRNA 由數百個 siRNA(21 bp)組成,覆蓋目標 mRNA 的 300 - 600 bp 區域。每條 siRNA 在池中的濃度較低,因此脫靶效應較低,同時可產生有效的基因敲除。
產品優點
- 保證 基因沉默
- 比單一化學合成的 siRNA 更低的脫靶效應
- 高靶上特異性使其成為有效的初級篩選工具(圖 2)
- 經濟實惠的基因組規模 RNAi 篩選工具

圖 2.用針對 Renilla Luciferase(陰性對照)和不同表達靶點的 esiRNA 轉染 HeLa 細胞後,在 mRNA 和蛋白水平上對基因敲除的評估。A) qPCR 數據驗證了轉染後 24 小時的基因敲除。所有轉染的 mRNA 豐度都降低了 >90%。B) 定量 Western 印迹数据验证了转染后 72 小时的高效基因敲除。蛋白水平降低了 45% 到 90%。
產品特色
以下適用於esiRNA規格表中的所有產品選項:
- 純化:Q-Sepharose分離、異丙醇沉澱及乙醇洗滌
- 序列形式:數百個 siRNA 的匯集,平均雙工長度為 21 bp
- 品質控制:在兩個階段進行
o cDNA 克隆的 PCR 產物通過凝膠電泳和 DNA 測序進行分析
o 消化反應通過凝膠電泳進行分析 - 穩定性:儲存於 -20 °C 時可存活 2 年
對於以下標示為「詢問」的任何事項,或如果您有不同於其他一般規格的需求,請發送請求至 [email protected]。
。| esiRNA產品選項與規格 | |
|---|---|
| |
esiLibrary |
|
esiFLEX |
|
esiOPEN |
|
esiSEC |
|
對照 esiRNA
提供多種陽性和陰性對照(圖 3):
- 陽性對照:人類 Eg5 (Kif11) esiRNA 以及所有經驗證的 esiRNA (見下表),以確保轉染 & 實驗設定最佳化
- 陰性對照: RLUC, FLUC & EGFP (EGFP也可在表達EGFP的細胞中作為陽性對照) esiRNA以區分序列特異性沉默和非特異性效果

圖 3.使用針對 A) RLUC(陰性對照)和 B) Eg5/KIF11(陽性對照)的 esiRNA 轉染 HeLa 細胞的表型分析。RLUC 不會誘發任何表型變化,而 Eg5/KIF11 則會誘發細胞有絲分裂停滯,如圓形細胞所示。
Validated Western Blot 驗證資料)。請參閱表中按基因符號排序的常見已驗證 esiRNA 清單。已驗證的 esiRNA 適合轉染最佳化及作為陽性對照。
lncRNAesiRNA
轉錄組分析顯示,高達 90% 的基因組被轉錄成非編碼 RNA,其中包括 lncRNA 等類型。lncRNA 與不同的功能有關,包括染色質修飾、共同啟動轉錄因子、轉錄、與 RNA 結合蛋白互動,以及抑制啟動子。此外,lncRNA 也與癌症等疾病有關。lncRNA 如何運作的細節必須進一步闡明。esiRNA 是研究 lncRNA 功能的有效篩選工具(圖 4)。
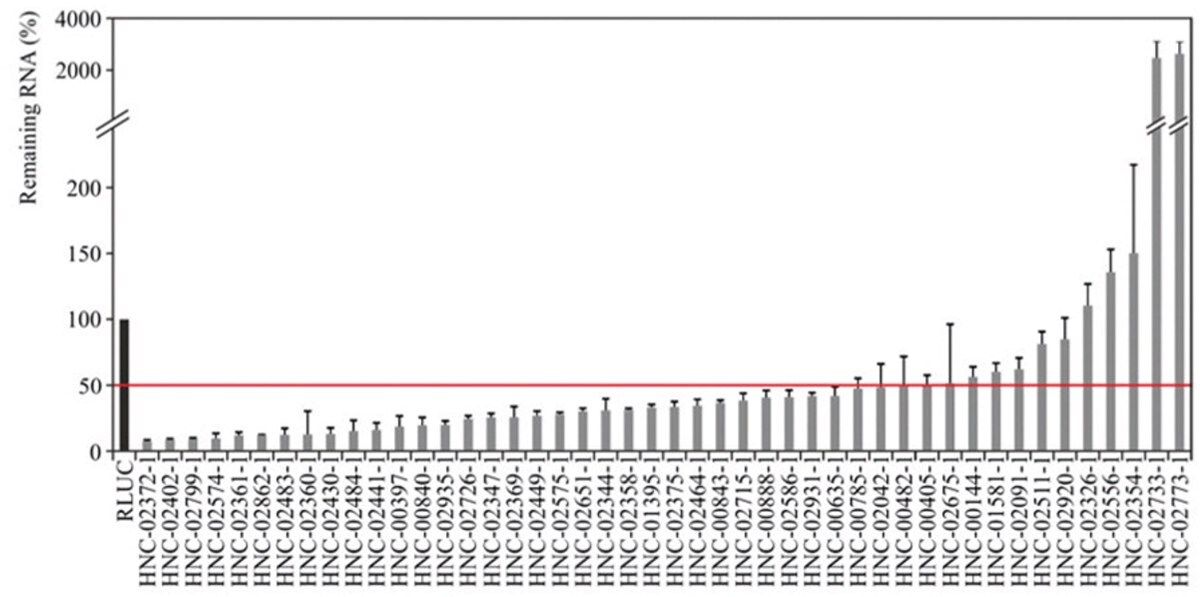
圖 4.用相應的 esiRNA 轉染 HeLa 細胞 24 小時後,驗證敲除 lncRNA 的 qPCR 資料。對照組轉染了針對 RLUC 的 esiRNA 並用於標準化。
產品製造
生物製備 (圖 5),esiRNA 是用內切核酸酶 (endoribonuclease, E. coli RNase III) 裂解長 dsRNA (double-stranded RNA) 而製成
。
圖 5.單基因 esiRNA 的製造概覽。A) 使用 DEQOR 來選擇 mRNA 目標區域,以產生最大數量的高效 siRNA,涵蓋所有已知的轉錄本變異,並將脫靶效應降至最低。B) 從 cDNA 克隆中提取目標區域,並通過 PCR 進行擴增(兩種引物均用於引入 T7 啟動子。 C) 利用 RNA 聚合酶在體外轉錄 PCR 产物。D) 用 RNase III 消化退火的長雙鏈 RNA (dsRNA),並純化去除 DNA 模板、殘留的 NTP 和未完全消化的 dsRNA。最後,esiRNA 即成為由數百個 siRNA 組成的池子,平均雙工長度為 21 bp。
常見問題
若要進一步瞭解 esiRNA,請造訪我們的 常見問題 部分。
Video Tutorial
學習使用 esiRNA 在哺乳動物細胞中進行篩選。
如果需要其他協助,請至 瞭解更多有關 RNA 干擾:從單基因研究到全基因篩選 - Dr. Julia Krüger Eupheria Biotech GmbH 的資深科學家
。選擇引文
單基因 esiRNA
全基因組與子庫篩選
lncRNA esiRNA
若要繼續閱讀,請登入或建立帳戶。
還沒有帳戶?為便利客戶閱讀,此頁面中文以機器翻譯完成。雖然我們已盡力確保機器翻譯的準確性,但機器翻譯並非完美。如果您對機器翻譯的內容不滿意,請參考英文版本。