Úvod
Fluorescenční barviva nebo sondy jsou součástí směsí PCR, aby bylo možné sledovat změnu koncentrace amplikonu DNA v průběhu reakce. V této kapitole je popsáno několik oblíbených detekčních metod, které jsou dobře zavedené pro nepřímé měření templátu v qPCR. Kromě toho bylo prokázáno, že sondy s dvojím značením a některá barviva vázající DNA dobře fungují při digitální PCR (dPCR).
Přehled sekcí
Pomocí obsahu vpravo můžete přejít k dalším částem Technického průvodce technologiemi PCR.
Barviva vázající dsDNA
Barviva vázající dvouřetězcovou DNA (dsDNA) fungují jako interkalační činidla a/nebo činidla vázající minoritní drážky a emitují detekovatelnou fluorescenci, když jsou vázána na dsDNA, ale mají velmi nízké pozadí, když jsou volná v roztoku. Intenzita fluorescenčního signálu se proto zvyšuje úměrně množství přítomného amplikonu. Barviva vázající dvouřetězcovou DNA jsou oblíbená, protože představují levnou možnost detekce a nevyžadují další konstrukční úvahy.
Barvivo SYBR® Green I
Toto barvivo je nejoblíbenějším barvivem vázajícím dsDNA a má dlouhou historii používání v molekulární biologii. Pokud je barvivo SYBR Green I volné v roztoku a je přítomna pouze jednořetězcová DNA (ssDNA), vyzařuje signál nízké intenzity (obrázek 5.1, tabulka 5.1). Jak PCR postupuje a množství dsDNA se zvyšuje, na amplikony se váže více barviva, a proto se intenzita signálu zvyšuje (viz animace na následující webové stránce sigma.com/sybr-animation). Protože se však barvivo váže na všechny amplifikované produkty bez rozdílu, přispívají k celkové fluorescenci také artefakty, jako jsou artefakty vznikající v důsledku dimerů primerů nebo nespecifické vazby primerů. To může ztížit přesnou kvantifikaci, zejména při nízkých koncentracích templátu. Analýza křivky tání po provedení PCR však může pomoci určit specifičnost reakce1 (obrázek 5.2).

Obrázek 5.1.Barvivo SYBR Green I v průběhu reakce přechází mezi nenavázaným (denaturovaným) a vázaným (žíháním a prodlužováním) stavem a intenzita signálu se zvyšuje s rostoucím množstvím amplikonů.
aPři komplexu s dsDNA

Obrázek 5.2.Příklad analýzy křivky tání. dsDNA-vázaná barviva se vážou reverzibilně, takže intenzita fluorescence klesá s rostoucí teplotou reakce nad teplotou tání (Tm). Ve spojení s kontrolami umožňuje tento typ analýzy detekovat nespecifické produkty, které tají při jiných teplotách než specifický produkt.
Sondy
Ve všech aplikacích qPCR je amplifikační reakce řízena specifickými dopřednými a zpětnými primery. Na rozdíl od testů, které se spoléhají na detekci pomocí barviv vázajících dsDNA, však systémy detekce sond neobsahují volné barvivo, ale spíše třetí oligonukleotid (a někdy čtvrtý) konjugovaný s reportérovým barvivem a/nebo zhášecí částí.
Dvojitě značené sondy
Dvojitě značené sondy (známé také jako hydrolyzované nebo alternativně TaqMan®. sondy) se používají v 5' nukleázovém testu2,3, což je nejoblíbenější chemie detekce sond (obrázek 5.3; viz také animace na následující webové stránce: sigma.com/probe-animation). Dvojitě značená sonda je jednořetězcový oligonukleotid, který je značen reportérovým barvivem a zhášecí částí. Reporter je umístěn na 5' konci a zhášeč na 3' konci. Zhášeč absorbuje přirozenou fluorescenční emisi reporteru, obvykle přenosem energie Forsterova typu, častěji označovaným jako fluorescenční rezonanční přenos energie (FRET). Po amplifikaci z předního primeru se Taq DNA polymeráza setká se sondou. Aktivita 5' exonukleázy, která je vlastní Taq DNA polymeráze, pak oddělí 5' reporter od 3' zhášeče (Tabulka 5.2, Obrázek 5.3), který poskytuje fluorescenční signál, jenž je úměrný výtěžku amplikonu.
Test hydrolyzační sondy je specifický a přesný pro kvantifikaci cílů s nízkým počtem kopií. Specifičnost lze ještě zlepšit zahrnutím modifikovaných nukleotidů, jako je uzamčená nukleová kyselina v sondě (jak je popsáno níže). Sondy modifikované uzamčenou nukleovou kyselinou jsou zvláště užitečné pro rozlišení jednonukleotidových polymorfismů (SNP) nebo jiných podobných sekvencí. Sondy s dvojím značením vyžadují pečlivý návrh (PCR/qPCR/dPCR Assay Design) a jsou obecně dražší než barviva vázající dsDNA. Kromě toho, zatímco amplifikace nespecifických produktů může zůstat nezjištěna, vedlejší reakce mohou způsobit, že celková reakce bude méně účinná, a proto mohou testy obsahující sondy stále těžit z optimalizace (Purifikace vzorků a hodnocení kvality).

Obrázek 5.3.Mechanismus dvojitě značených sond. Taq DNA polymeráza prodlužuje primer umístěný na stejném vlákně jako sonda, dokud nedosáhne pozice sondy. Vrozená exonukleázová aktivita hydrolyzuje sondu z 5' na 3', čímž se do roztoku uvolní reportérové barvivo, a tím dojde ke zvýšení fluorescence. Naměřený fluorescenční signál je přímo úměrný množství cílové DNA.
Většina termálních cyklérů PCR v reálném čase má více detekčních kanálů, které umožňují flexibilní výběr značek sond. Je velmi důležité vybrat reportérová barviva, která jsou kompatibilní s detekčními kanály přístroje, a zajistit správné filtry a kalibraci. Při multiplexování je třeba kombinovat reporterová barviva, která se od sebe co nejvíce liší, aby se minimalizoval optický křížový hovor. Mezi typické reportéry patří: FAM, HEX, TxRd (Sulforhodamin 101-X) a kyanin 5. Za stejných podmínek je obvyklé pozorovat rozdíly v intenzitě emise různých reporterů. Z tohoto důvodu je vhodné analyzovat údaje z každé kombinace reporterů nezávisle (s použitím různých prahových nastavení podle toho, jak je to vhodné pro emisi sondy).
Molekulární majáky
Molekulární majáky (známé také jako hybridizační sondy) jsou jednořetězcové sondy, které jsou drženy v konformaci vlásenkové smyčky (20-25 nukleotidů) komplementárními kmenovými sekvencemi (4-6 nukleotidů) na každém konci4 (obrázek 5.4). Vlasová smyčka je komplementární k templátu a vodíkově vázané kmenové sekvence umožňují 3' zhášeči potlačit fluorescenci 5' reportéru (Tabulka 5.3), když je sonda volná v roztoku.
Obrázek 5.4.Mechanismus molekulárních majáků. Molekulární majáky hybridizují se specifickou cílovou sekvencí, což způsobí otevření struktury vlásenkové smyčky, která oddělí 5' reportér od 3' zhášeče. Protože zhášeč již není v blízkosti reporteru, dochází k emisi fluorescence. Na rozdíl od dvojitě značených sond není mechanismus detekce molekulárních majáků závislý na degradaci během reakce. Naměřený fluorescenční signál je přímo úměrný množství cílové DNA.
LightCycler® Sondy
Sonda LightCycler nebo systém FRET (známý také jako duální hybridizační sondy) se skládá z dvojice jednovláknových fluorescenčně značených oligonukleotidů5,6 (Obrázek 5.5). Oligo sonda 1 je na 3' konci označena donorovým fluoroforovým barvivem a oligo sonda 2 je na 5' konci označena jedním z několika dostupných akceptorových fluoroforových barviv (Tabulka 5.4). Volná 3' hydroxylová skupina Oligo Probe 2 musí být blokována fosfátovou skupinou, aby se zabránilo prodloužení DNA polymerázy.

Obrázek 5.5.Mechanismus FRET sond LightCycler. Během žíhání dochází k hybridizaci primerů a obou sond s jejich specifickými cílovými oblastmi, čímž se donorové barvivo dostane do těsné blízkosti akceptorového barviva (sondy jsou obvykle od sebe vzdáleny 1 až 5 nukleotidů). Když je donor excitován světlem z přístroje pro PCR v reálném čase, dochází k přenosu energie FRET z donoru na akceptor. Detekuje se emisní vlnová délka barviva na akceptorové sondě. Zvýšení fluorescenčního signálu je přímo úměrné množství cílové DNA.
Scorpions® sondy
Scorpions® sondy jsou k dispozici ve dvou formách: uni-probe a bi-probe. Uni-sonda se skládá ze struktury stonkové smyčky, podobně jako Molecular Beacon, která je však připojena k dopřednému primeru s blokátorem PCR umístěným mezi dvěma částmi oligo7 (blokátor zabraňuje Taq DNA polymeráze v prodloužení primeru (obrázek 5.6A, tabulka 5.5).
Struktura bi-sondy je duplex s 5' reportérem, sekvencí sondy, blokátorem PCR a dopředným primerem na jednom vlákně a 3' zhášecím vláknem na druhém vlákně; zhášecí vlákno je duplexováno s reportérovým vláknem (obrázek 5.6B).

Obrázek 5.6.Mechanismus sond Scorpions®. Uni-sonda A) má všechny složky sondy na jednom vlákně, zatímco bi-sonda B) má složky sondy na dvou vláknech. Oba formáty sond Scorpions® obsahují dopředný primer PCR. Tento dopředný primer je prodloužen a stává se součástí nově vytvořeného amplikonu. Během žíhání/rozšíření se sekvence sondy v přípravku Scorpions® hybridizuje s templátem, čímž dojde k oddělení reportéru od zhášedla, a tím k fluorescenčnímu signálu. Protože ocásek Scorpions® a amplikon jsou součástí stejného vlákna, je detekční interakce intramolekulární, a proto rychlejší než u jiných systémů detekce sond. Templát se obvykle volí tak, aby se nacházel v rozmezí 5 až 50 bází od 3' konce primeru Scorpions®. Jak uni-sonda, tak bi-sonda Scorpions® vyžadují samostatný reverzní primer.
Quenchers
Většina systémů detekce sond vyžaduje zhášecí část. Některé z těch, které se používaly v původních strukturách sond, byly akceptorová fluorescenční barviva, například TAMRA, která dobře fungují s FAM, ale nejsou vhodná pro jiná barviva. TAMRA jako samotné reportérové barvivo produkuje fluorescenci, a proto může mít za následek špatný poměr signál/šum. Z tohoto důvodu byly společností Biosearch Technologies vyvinuty tmavé zhášeče, které místo světla emitují teplo, jako je Black Hole Quencher® (BHQ®). Volba tmavých zhášečů jako oblíbené alternativy k molekulám barviv poskytuje zhášení v širokém rozsahu vlnových délek a otevírá možnost multiplexních reakcí obsahujících větší počet kombinací cíl/sonda.
Onyx Quencher™ (OQ™) je patentovaný tmavý zhášeč od společnosti Sigma-Aldrich. K dispozici jsou čtyři deriváty (OQA, OQB, OQC a OQD). Jak je uvedeno v tabulce 5.6, čtyři kverulanty Onyx jsou kompatibilní s řadou populárních reportérových barviv.
Údaje uvedené v obrázku 5.7 ukazují amplifikaci umělého templátu, který byl odvozen ze syntetického oliga pro optimalizaci cílového testu Schistosoma mansoni8. K detekci byla použita sonda značená FAM se srovnatelným tmavým zhášečem, CDQ (A) nebo OQA (B). Z těchto údajů je zřejmé, že jak CDQ, tak OQA mají ekvivalentní výkon, s podobnou fluorescencí pozadí a podobnými hodnotami Cq pro analyzovaná data z templátu o stejné koncentraci.
Shrnem lze říci, že OQ má ekvivalentní výkon jako CDQ a co je důležité, je k dispozici bez licence, omezení a licenčních poplatků pro jakoukoli aplikaci. Díky tomu je Onyx Quencher vynikající a cenově výhodnou volbou pro vývoj komerčních souprav a reagencií pro molekulární diagnostiku, které obsahují qPCR sondy.
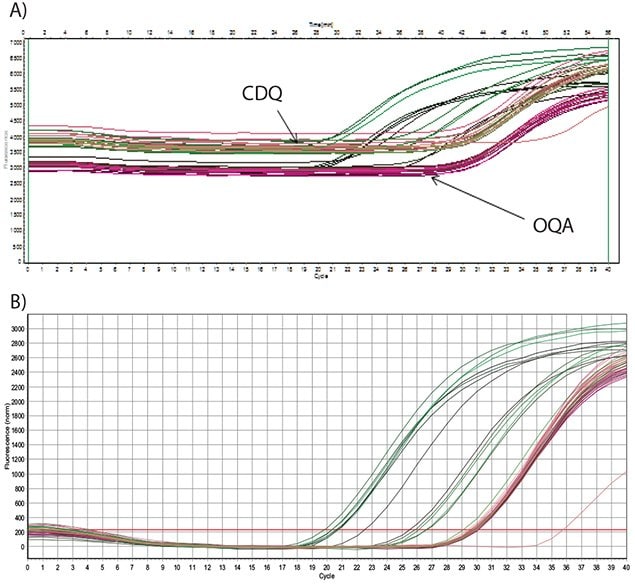
Obrázek 5.7.Ředění umělého oliga bylo amplifikováno pomocí primerů o velikosti 250 nM a amplikon byl detekován pomocí dvojitě značené sondy o velikosti 200 nM. A) Sonda byla značena FAM a zhášena CDQ nebo OQA (podle pokynů) a jsou zobrazena nezpracovaná fluorescenční data. B) Sonda byla značena FAM a zhášena CDQ nebo OQA a jsou zobrazena data korigovaná na základní linii. Mezi výkony obou sond nejsou žádné významné rozdíly.
Analogy nukleových kyselin
K výrobě oligonukleotidů se změněnými biochemickými vlastnostmi lze použít řadu modifikací. Tyto modifikace obvykle poskytují vyšší Tm který lze manipulovat tak, aby poskytoval lepší specifitu. To umožňuje navrhovat testy v oblastech s náročnou sekvencí nebo v případech, kdy je jeden oligonukleotid potřebný k detekci několika sekvencí, např, všechny sérotypy viru.
Zamčená nukleová kyselina
Zamčená nukleová kyselina je analogická báze RNA (obrázek 5.8), která při začlenění do sond poskytuje zvýšenou citlivost a specifičnost9. Sondy s bázemi Locked Nucleic Acid mají větší tepelnou stabilitu, a proto silněji hybridizují s templátem. Každá uzamčená báze nukleové kyseliny může zvýšit Tm sondy až o 8 °C10, což z uzamčené nukleové kyseliny činí výkonný nástroj v testech diskriminace SNP.sup>11 (neshoda jediné báze má větší destabilizující účinek na tvorbu duplexu než bez Locked Nucleic Acid), multiplexování (umožňuje jednodušší Tsub>m optimalizaci) a problematické cílové sekvence (sondy s uzamčenou nukleovou kyselinou mohou být kratší, což umožňuje jejich návrh kolem problematických oblastí, jako jsou oblasti bohaté na AT nebo GC, repetitivní sekvence nebo sekvence s významnou sekundární strukturou). Bylo také zjištěno, že primery PCR obsahující uzamčenou nukleovou kyselinu jsou výhodné v aplikacích, jako je genotypování SNP12.
Uzamčená nukleová kyselina také poskytuje ochranu proti trávení nukleázami, takže je vhodná pro použití in vivo13.

Obrázek 5.8.Srovnání struktur nukleotidů uzamčené nukleové kyseliny a DNA. Uzamčená nukleová kyselina se od DNA liší tím, že obsahuje ribózu s methylenovým můstkem mezi 2' kyslíkem a 4' uhlíkem, který "uzamyká" ribózu v 3' endo konformaci, zatímco DNA obsahuje 2'-deoxyribózu bez methylenového můstku.
Summary
Při výběru metody detekce pro konkrétní aplikaci je třeba zvážit mnoho faktorů. Ačkoli jsou barviva SYBR Green I a testy s dvojitě značenou sondou oblíbené a obecně fungují dobře, existují situace, kdy mohou být vhodnější jiné detekční systémy. Tabulka 5.7 může sloužit jako vodítko při počátečním vývoji testu.
Prázdná místa znamenají, že metoda detekce se nedoporučuje; X = dobrý výkon; a XX = lepší výkon.
Odkazy
Abyste mohli pokračovat ve čtení, přihlaste se nebo vytvořte účet.
Nemáte účet?