Uzamčená nukleová kyselina a pojivo pro menší drážky / zhášeč tmy Eclipse
Locked Nucleic Acid a Minor Groove Binder / Elipse Dark Quencher (MGB:EDQ) jsou modifikace, které zvyšují výkonnost oligonukleotidů. Zvyšují stabilitu a zlepšují specifičnost a afinitu pro párování bází s cílovými sekvencemi. Uzamčená nukleová kyselina zlepšuje primery a sondy (v kombinaci s BHQ™) v qPCR i účinnost terapeutických modalit, jako jsou antisense oligonukleotidy (ASO). Mezitím MGB:EDQ se svým obsaženým zhášečem specificky zlepšuje sondy v qPCR.
Zamčená nukleová kyselina
Uzamčená nukleová kyselina je nový typ analogu nukleové kyseliny, který obsahuje 2'-O, 4'-C methylenový můstek (Obrázek 1). Tento můstek - uzamčený v 3'-endo konformaci - omezuje flexibilitu ribofuranózového kruhu a uzamyká strukturu do rigidní bicyklické formace. To zvyšuje výkonnost testů a rozšiřuje možnosti použití.

Obrázek 1.Struktura uzamčené nukleové kyseliny a monomerů DNA v nativním stavu.
Locked Nucleic Acid v PCR primerech, qPCR sondách a dalších typech oligonukleotidů je rozpustná ve vodě a standardních pufrech a dodržuje pravidla Watson-Crickova párování bází.1
Výhody uzamčené nukleové kyseliny
Při zabudování do oligonukleotidů nabízí uzamčená nukleová kyselina několik výhod ve srovnání s bázemi DNA pouze v nativním stavu, včetně následujících:
- Zvýšená tepelná stabilita a hybridizační specifičnost
- Přesnější kvantifikace genů a alelická diskriminace
- Snadnější a flexibilnější návrhy pro problematické cílové sekvence
Zvýšená tepelná stabilita a specifičnost hybridizace
Zavedení uzamčené nukleové kyseliny do oligonukleotidů zvyšuje tepelnou stabilitu duplexu2 a zlepšuje specifičnost hybridizace oligonukleotidů s cílovými sekvencemi.3 V případě qPCR se tím snižuje fluorescence pozadí způsobená falešnou vazbou, což zvyšuje poměr signálu k šumu. Kromě toho může lepší hybridizace primeru PCR nebo sondy qPCR k cílovému vzorku zvýšit teplotu tání (Tm) až o 8°.C na jednu substituci monomeru uzamčené nukleové kyseliny v podmínkách střední soli ve srovnání s oligonukleotidy DNA v nativním stavu4 (Tabulka 1). Toto zvýšení hybridizace vytváří významné rozšíření rozsahu podmínek testů a umožňuje úspěšnější multiplexování.5
Přesnější kvantifikace genů a rozlišení alel
Schopnost oligonukleotidů rozlišovat mezi alelami prostřednictvím SNP je výrazně zvýšena začleněním bází uzamčených nukleových kyselin.sup>6-8 (Obrázek 2). Přítomnost neshody jediné báze má větší destabilizující účinek na tvorbu duplexu mezi oligonukleotidem Locked Nucleic Acid a jeho cílem než u oligonukleotidu DNA v nativním stavu. Kratší oligonukleotidy obsahující báze uzamčené nukleové kyseliny lze používat při stejných teplotách jako delší oligonukleotidy DNA v nativním stavu.

Obrázek 2.Dvojitě značené sondy s uzamčenou nukleovou kyselinou diskriminují lépe než dvojitě značené sondy s DNA při analýze genotypů SNP9. Růžová) analýza mutantní DNA s mutantní sondou Locked Nucleic Acid (16merů se 3 bázemi Locked Nucleic Acid). Zelená) analýza mutantní DNA s mutantní sondou DNA v nativním stavu (25mer). Červená) analýza DNA divokého typu s nativním stavem mutantní sondy DNA (25mer). Fialová) DNA divokého typu s mutantní sondou Locked Nucleic Acid (16merů se 3 bázemi Locked Nucleic Acid).
Snadnější a flexibilnější návrhy problematických cílových sekvencí
Díky lepším hybridizačním vlastnostem uzamčené nukleové kyseliny s doprovodným zvýšením Tm, lze oligonukleotidy Locked Nucleic Acid syntetizovat tak, aby byly kratší, což překonává určitá omezení návrhu, která se vyskytují u oligonukleotidů DNA v nativním stavu. Konkrétně lze oligonukleotidy s uzamčenou nukleovou kyselinou navrhnout tak, aby řešily tradičně problematické cílové sekvence, jako jsou oblasti bohaté na AT nebo GC. Například oligonukleotidy DNA bohaté na AT musí být často delší než 30 bází (někdy více než 40 bází), aby vyhovovaly pokynům pro návrh amplikonu, ale přesto mohou mít špatný výkon. U oligonukleotidů s uzamčenou nukleovou kyselinou usnadňuje selektivní umístění bází s uzamčenou nukleovou kyselinou optimální návrh vysoce specifických, kratších oligonukleotidů, které dobře fungují i při délce pouhých 13 až 20 bází. Také návrh oligonukleotidů pro cílení obtížných SNP, jako je například relativně stabilní neshoda G:T, je díky Locked Nucleic Acid značně usnadněn.
Další výhody
Primery PCR a sondy qPCR Locked Nucleic Acid jsou kompatibilní se všemi termocyklery reálného času a přístroji pro detekci koncového bodu analýzy. Nejsou nutné žádné specializované přístroje.
Aplikace uzamčené nukleové kyseliny
Uzamčenou nukleovou kyselinu lze začlenit do všech dostupných qPCR detekčních chemikálií, včetně:
- SYBR® zelené primery
- dvojitě značené sondy
- molekulární majáky
- LightCycler® Sondy
- Scorpions® Sondy
Je užitečné pro následující účely:
- Detekce SNP
- Diskriminace alel
- Detekce patogenů
- Multiplexování
- Dekomplexování Kvantifikace virové zátěže
- Analýza exprese genu
- Stanovení kopií genu
Může být také použit v těchto sekvencích:
- Antisense oligonukleotidy
- Dekoy oligonukleotidy
- Zachytávací sondy
- Aptamery
- Ribozy
MGB:EDQ
Sonda MGB:EDQ je jednořetězcový oligonukleotid značený dvěma různými barvivy. Na 5' konci je umístěno reportérové barvivo a na 3' konci je umístěn Minor Groove Binder / Eclipse Dark Quencher (Obrázek 1). Molekula zhášeče inhibuje přirozenou fluorescenční emisi reporteru pomocí přenosu energie fluorescenční rezonance (FRET, obrázek 2).
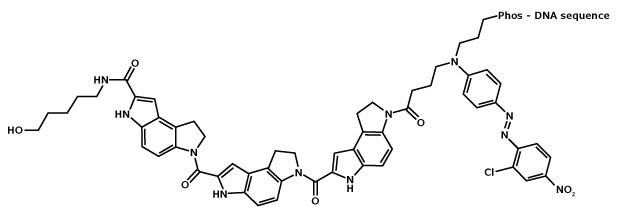
Obrázek 1.Struktura MGB:EDQ. MGB neboli CDPI3 je tripeptid dihydropyrroloindol-karboxylátu a jeho bumerangové zakřivení mu umožňuje přizpůsobit se tvaru a tím se silně vázat na minoritní drážku B-formy DNA.
1. Sonda v roztoku vyzařuje nízkou fluorescenci
2. Emise fluorescence hydrolýzou
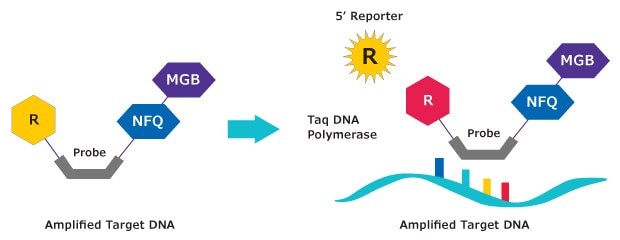
Obrázek 2.Mechanismus účinku. MGB:EDQ sondy jsou hydrolyzní sondy. Primer je prodloužen polymerázou a sonda se váže na specifický templát DNA. Hydrolýza uvolní reportér z hybridu sondy a cíle, což způsobí zvýšení fluorescence. Naměřený fluorescenční signál je přímo úměrný množství cílové DNA.
VÝHODY MGB
MGB zvyšuje výkonnost qPCR několika způsoby:
- Zvýšená specifičnost: MGB zvyšuje specifičnost hybridizace sonda-cíl, čímž snižuje pravděpodobnost nespecifické amplifikace
- Vyšší citlivost: MGB umožňuje použití kratších sond, které zvyšují citlivost testů qPCR
- Nastavení teploty tání: MGB umožňuje nastavení Tm sondy, což může být výhodné pro návrh sond qPCR s optimálními hodnotami Tm
- Snížení signálu pozadí: MGB přispívá ke snížení fluorescence pozadí, což vede ke zlepšení poměru S:N nebo poměru signál/šum
- Zvýšená stabilita: MGB může zlepšit stabilitu hybridu sondy a cíle, čímž se snižuje pravděpodobnost degradace sondy
- Pružnost návrhu: Použití MGB rozšiřuje možnosti návrhu sond, což usnadňuje vytváření účinných testů qPCR
Sondy qPCR se zvýšeným obsahem MGB jsou cenným nástrojem pro dosažení spolehlivých a robustních výsledků qPCR.
APLIKACE MGB:EDQ
MGB:EDQ lze začlenit do této chemie pro detekci qPCR:
- Dvojitě značené sondy (s 6-FAM™, HEX™ a TET™; na další se zeptejte)
Je užitečné pro následující:
- Detekce SNP pomocí diskriminace neshod
Odkazy
Abyste mohli pokračovat ve čtení, přihlaste se nebo vytvořte účet.
Nemáte účet?