
在研究醣蛋白和醣肽時,有各種不同的策略來使 N 連結醣脫醣。這些策略可能涉及 PNGase F、PNGase A,甚至是使用內含糖苷酶和外含糖苷酶進行原生和順序脫糖。請繼續閱讀,詳細瞭解不同類型的 N 連結聚糖脫糖作用,並找出最適合您研究的策略。
閱讀更多
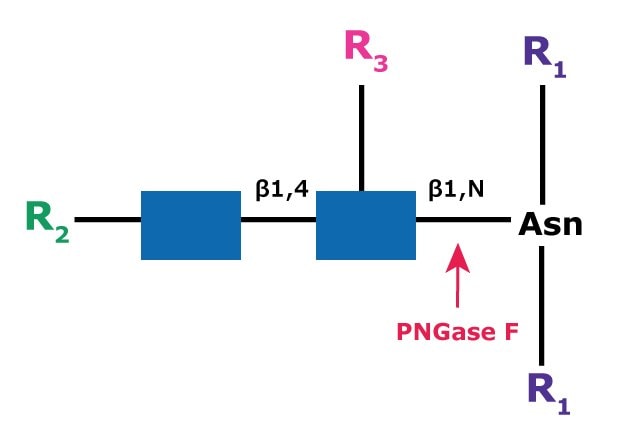
圖 1.PNGase F 的裂解位點和結構要求。
R1 = 由 H 以外的基團取代 N 和 C
R2 = H 或寡糖結構的其餘部分
R3 = H 或 α(1→6)岩藻糖
去除聚糖的天門冬酰胺殘基會被脫氨基為天門冬氨酸(圖 2)。寡糖被完整地保留下來,適合進一步分析。

圖 2.PNGase F 處理 N-聚糖的裂解產物。
鏈梗阻會減緩或抑制 PNGase F 對糖蛋白某些殘基的作用。用 SDS 和 2-巯基乙醇加熱使糖蛋白變性,可大大提高脫糖基化的速率。
蛋白組學級 PNGase F
蛋白組學級 PNGase F 是從稀磷酸鉀緩衝液中經廣泛純化和凍乾而製成的穩定產品。本產品不含甘油及其他可能干擾敏感性糖蛋白分析方法的穩定劑。特點包括:
- 優秀的應用於需要N-連接的脫糖基化(圖3)。
- 優異的性能,適用於印片、凝膠和溶液消化方法。
- ≥25,000單位/mg的高比活度。
- 相容於MALDI-TOF質譜分析。

圖 3.原生和 PNGase F 處理的α-1 抗胰蛋白酶的 SDS-PAGE 分析。在 SDS-PAGE 分離之前,測試樣品(第 5 層)在溶液中與 5 單位的 PNGase F 於 37 °C 下進行 1 小時的脫糖作用。注意脫糖基化後帶的移動性改變。
Lanes
1:分子量標記
2, 3, 4:對照,原生α-1抗胰蛋白酶
5: 溶液中脫糖的α1 抗胰蛋白酶
此 PNGase F 也可在 GlycoProfile™ I, In-Gel Deglycosylation Kit 中使用。本試劑組經過優化,可提供方便且可重複的方法,從 1D 或 2D 聚丙烯酰胺凝膠片中對蛋白質樣本進行 N-去糖基化和胰蛋白酶消化,以進行後續的 MS 或 HPLC 分析。
PNGase A (GLYCOPEPTIDASE A)
PNGase A(又名 Glycopeptidase A 或 Peptide-N-Glycosidase A)會水解含有岩藻糖殘基 α(1→3)- 連接至天冬醯鏈結 N-acetylglucosamine 的寡糖(圖 4)。與 PNGase F 相似,移除聚糖的天冬酰胺殘基會被脫氨基為天冬氨酸。然而,當N-連結寡糖上有穀氨酸時,PNGase A 則無效。
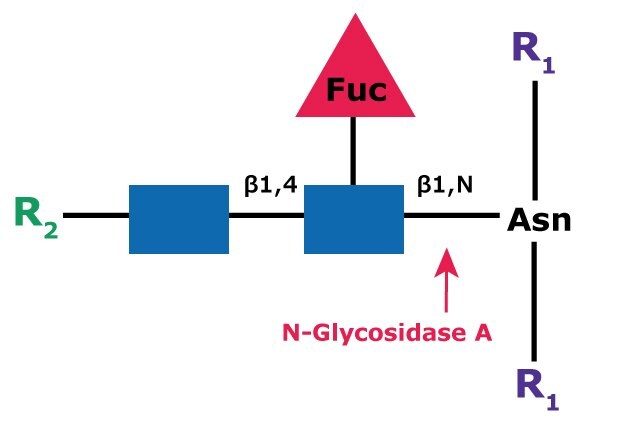
圖 4.PNGase A(糖肽酶 A)的裂解位點和結構要求。
R1 = H 以外的基團取代 N 和 C
R2 = H 或寡糖結構的其餘部分
Native and SEQUENTIAL N-LINKED GLYCAN STRATEGIES
糖蛋白可能有埋藏或不可用的糖結合物。因此,當從整個糖蛋白中移除聚糖時,可能需要變性來達到完全的脫糖基化,通常在酵素步驟前需要使用去垢劑來螯合。請參閱 Freeze, H.1
如果使用蛋白水解消化法,例如使用胰蛋白酶消化法,則脫糖基化步驟不需要變性,但您可能需要移除蛋白酶以容納脫糖基化酵素。
其他常用的內糖苷酶,例如內糖苷酶 H 和內糖苷酶 F 系列,因為其特異性有限,而且會留下一個 N-乙酰葡糖胺殘基附著在天冬酰胺上,所以不適合一般的 N 連鎖糖的脫糖基化。這些方法適用於原生蛋白質的脫糖基化。可以使用外糖苷酶進行順序脫糖。
ENDOGLYCOSIDASE H
內切糖苷酶 H (又名 β-N-乙酰葡糖胺苷酶 H)會裂解高甘露糖 N-連接聚糖中 N-乙酰葡糖胺殘基 GlcNac1 和 GlcNac2 之間的β-1,4 糖苷鏈,也稱為殼寡糖核心(圖 5)。裂解後會留下附著在蛋白質上天冬醯胺的聚糖 N-乙醯葡萄糖胺殘基,但會移除聚糖的大部分。這對於在原生條件下的脫糖基化特別有用,但聚糖可能需要外糖苷酶處理,以形成高甘露糖糖形。由於裂解位點與氨基酸序列的距離較遠,因此被認為更容易被酵素接觸到。
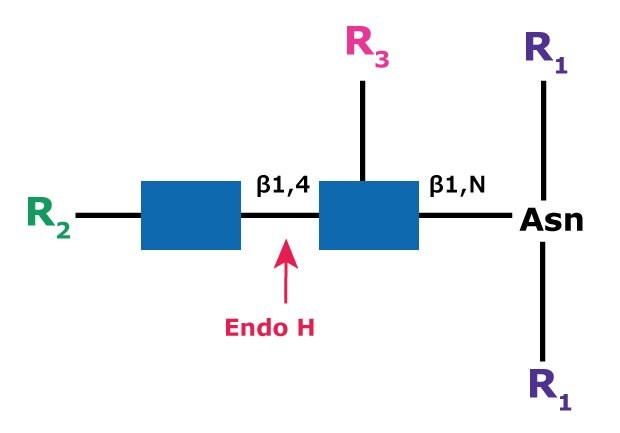
圖 5.內切糖苷酶 H (Endo H) 的裂解位點和結構要求。
R1 = 由 H 以外的基團取代 N 和 C
R2 = H 或寡糖結構的其餘部分
R3 = H 或 α(1→6)岩藻糖
這種酵素的特異性使得低聚甘露糖和大多數混合類型的聚糖(包括那些在核心結構上附有岩藻糖殘基的聚糖)會被裂解,而複合型聚糖則不會被釋放。因此,這種酵素對於選擇性地釋放糖蛋白中的高甘露糖 (低聚甘露糖) 或混合型聚糖非常有用。
內切糖苷酶 H 的功能 pH 值範圍為 5.0 到 6.0,最佳 pH 值為 5.5。在 4.5 到 8.5 的 pH 範圍內,於 37 °C 培養 48 小時也沒有觀察到活性的流失。然而,pH 值低於 4.5 時,活性會快速流失。內糖苷酶 H 可以 含反應緩衝液的重組溶液。
ENDOGLYCOSIDASE F
內切糖苷酶 F1、F2 和 F3 對蛋白質構象的敏感度比 PNGase F 低,更適合原生蛋白質的脫糖基化。內切糖苷酶 F1(Endo F1)可裂解天冬醯胺連結或游離的高甘露糖(oligomannose)和雜合結構,而內切糖苷酶 F2(Endo F2)和內切糖苷酶 F3(Endo F3)則可裂解複合結構。內切糖苷酶 F1、F2 和 F3 的連結特異性提出了蛋白質脫糖基化的一般策略,可在不使蛋白質變性的情況下移除所有類別的 N-連結寡糖。Native Protein Deglycosylation Kit 提供所有這三種酵素(Endo F1、Endo F2 和 Endo F3)的反應緩衝液和詳細互動。
內切糖苷酶 F1
Endo F1 (aka Endo-β-N-乙酰葡糖胺苷酶 F1)會在寡糖的 N 連結的二乙酰基曱基糖核中的兩個 N-乙酰葡糖胺殘基之間裂解,生成一個截短的糖分子,其中天冬氨酰胺上剩餘一個 N-乙酰葡糖胺殘基(圖 6)。
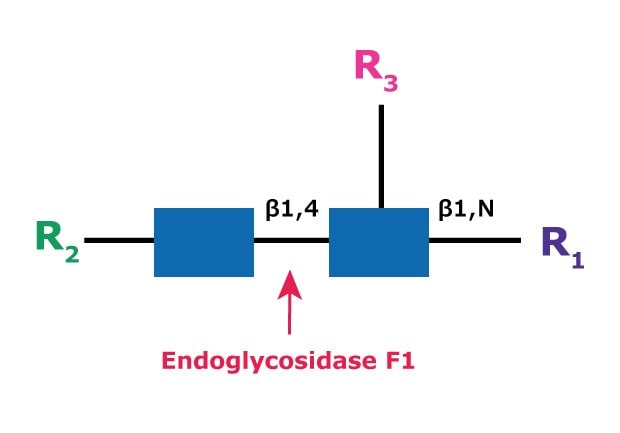
圖 6.
R1 = H 或 Asn
R2 = 低聚甘露糖或混合構型
R3 = H 或 α(1→6)岩藻糖
Endo F1 能裂解天冬醯胺連結或游離的高甘露糖(oligomannose)和雜交結構,但不能裂解複雜的寡糖(圖 7)。雜交結構的核心岩藻糖基化會使 Endo F1 的裂解率降低 50 倍以上。Endo F1 會裂解硫酸化的高甘露糖寡糖,而 Endoglycososidase H 則不會。Endo F1 可在原生或非變性脫糖條件下使用。
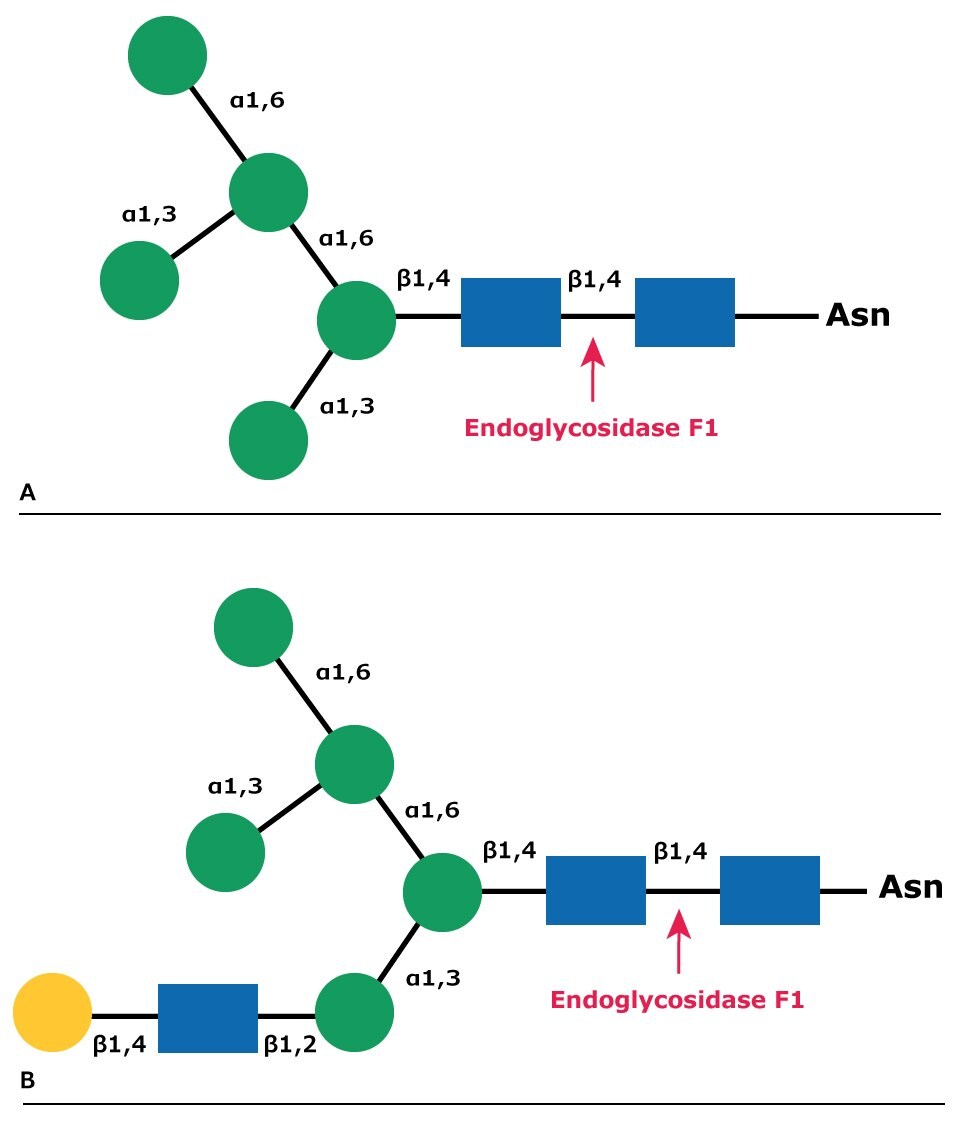
圖 7.內切糖苷酶 F1 在 (A) 高甘露糖和 (B) 混合糖中的裂解位點。
內切糖苷酶 F2
內切糖苷酶 F2 (aka Endo-β-N-乙酰葡糖胺苷酶 F2)會在寡糖的 N-連接的二乙酰基-chitobiose 聚糖核心的兩個 N-乙酰葡糖胺殘基之間裂解,生成一個截短的糖分子,其中天冬氨酰胺上剩餘一個 N-乙酰葡糖胺殘基(圖 8)。
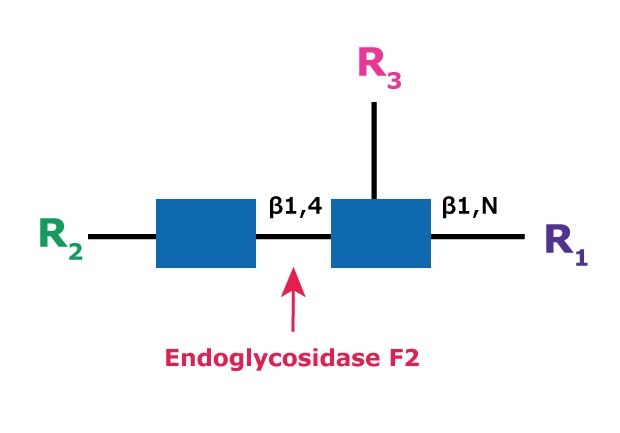
圖 8.
R1 = H 或 Asn
R2 = 雙甘露糖或低聚甘露糖配置
R3 = H 或 α(1→6)岩藻糖
Endo F2 會裂解雙年複合寡糖(圖 9)。與複合結構相比,高甘露糖寡糖的裂解率降低了 20 倍。Endo F2 不會裂解複合結構。Fucosylation 對 Endo F2 裂解雙年根結構的影響很小。Endo F2 可在原生或非變性脫糖基化條件下使用。
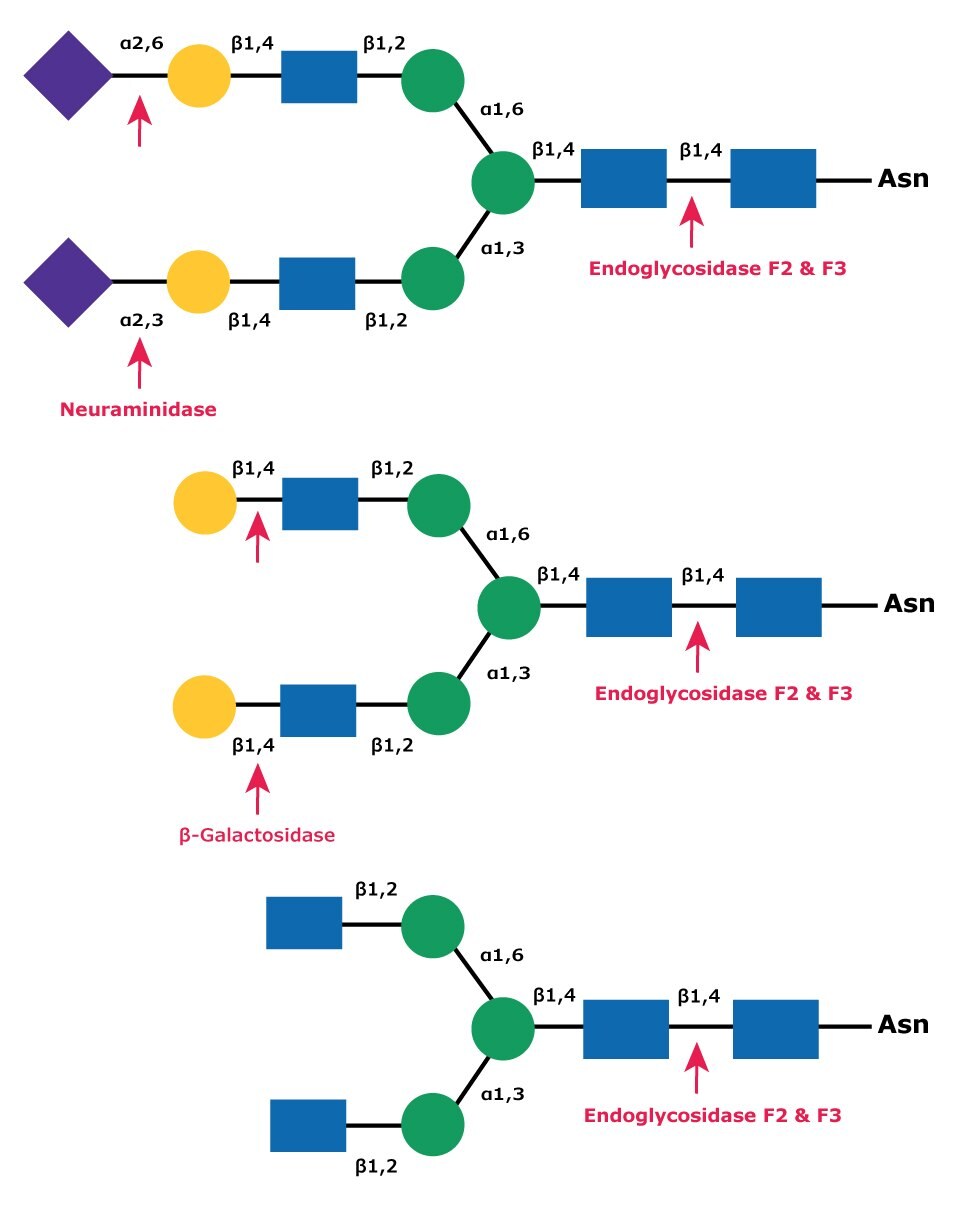
圖 9.內糖苷酶 F2 和 F3 在複雜雙年齡聚糖中的裂解位點。圖中顯示使用外糖苷酶神經氨酸酶(上)和β-半乳糖苷酶(中)去除末端單糖的順序降解過程。
內切糖苷酶 F3
內切糖苷酶 F3 (aka Endo-β-N-乙酰葡糖胺苷酶 F3)會在寡糖的 N-連接的二乙酰基-chitobiose 聚糖核心的兩個 N-乙酰葡糖胺殘基之間裂解,生成一個截短的糖分子,其中天冬氨酰胺上剩餘一個 N-乙酰葡糖胺殘基(圖 10)。
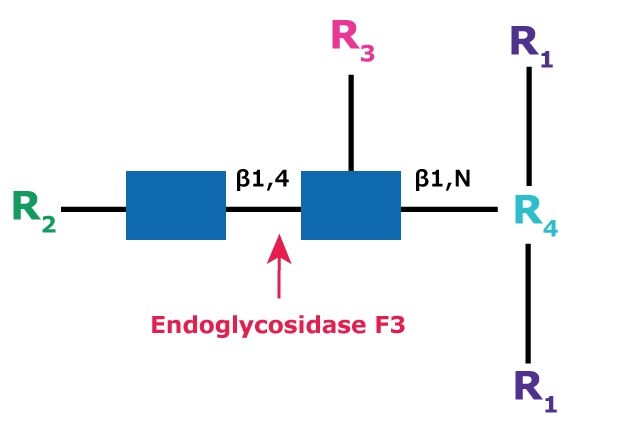
圖 10.內糖苷酶 F3 的裂解位點和結構要求。
R1 = 由 H 以外的基團取代 N 和 C
R2 = 雙元或三元複合寡糖或三甘露聚糖 (Man3) 核心
R4 = Asn (如果在 R3 處被岩藻糖基化,則為 Asn 或 H)
Endo F3 對低聚甘露糖和雜交分子沒有活性。Endo F3 能以緩慢的速度裂解非岩藻糖基化的雙胞寡糖和三胞寡糖複合寡糖,但只有在肽鏈結的情況下(圖 9 和 11)。雙ennary 結構的核心岩藻糖基化可增加活性達 400 倍。核心岩藻糖基化的雙胞結構是 Endo F3 的有效底物,即使是游離的寡糖。Endo F3 也會裂解游離寡糖和蛋白連結寡糖上的岩藻糖基化三甘露糖基核心結構。
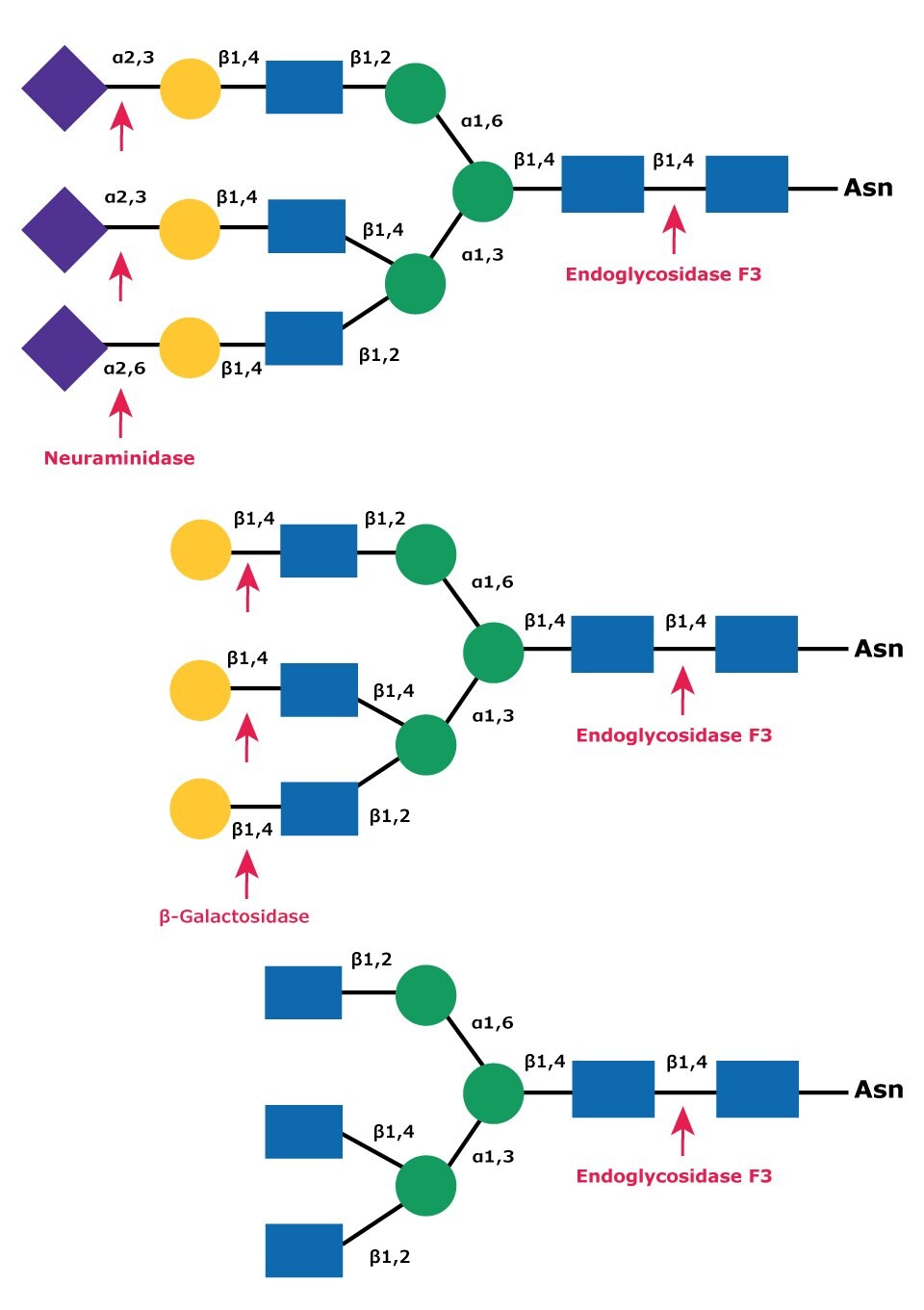
圖 11.內糖苷酶 F3 在複合三元糖中的裂解位點。使用外糖苷酶神經氨酸酶(上)和β-半乳糖苷酶(中)去除末端單糖的順序降解圖示。
複雜的四元戊糖的原生脫糖基化需要在使用 Endo F3 裂解之前,先順序水解至三單胞二乙酰基膽固醇(Man3GlcNAc2)核心(圖 12)。
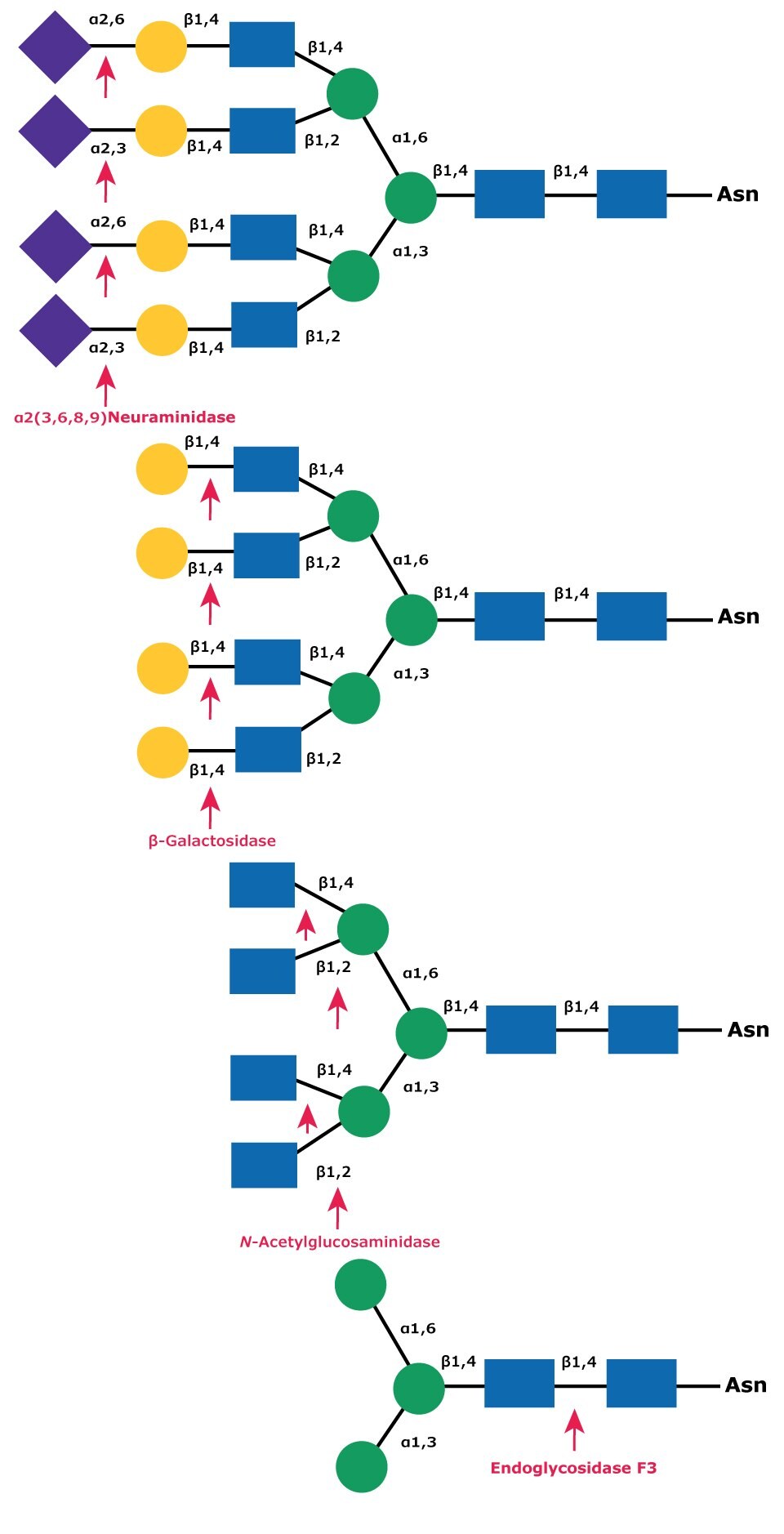
圖 12.複合四元糖的順序脫糖基化。使用外糖苷酶神經氨酸苷酶(上)、β-半乳糖苷酶(中)和 N-乙酰葡糖苷酶(下)依序去除末端單糖,直到剩下三甘露糖基二乙酰基膽固醇核,供後續的內切酶 F3 裂解。
EXOGLYCOSIDASES
外糖苷酶透過使用外糖苷酶對單糖進行順序脫糖基化,所有複雜的寡糖都可以還原為三單胞二乙酰基膽固醇糖 (Man3GlcNAc2) 核心。複雜的 N 連結聚糖可用神經醯胺酶、β-半乳糖苷酶和 N-乙酰葡糖胺苷酶選擇性地水解,可作為 Enzymatic Deglycosylation Kit 的一部分提供。在某些情況下,可能需要使用岩藻糖苷酶進行額外的裂解。剩餘的三單胞二乙酸基千itobiose 核心結構可以使用 Endoglycosidase F3 來去除。
在我們的 Glycobiology hub page 上找到更多應用資源。
Network error: Failed to fetch
參考資料
若要繼續閱讀,請登入或建立帳戶。
還沒有帳戶?為便利客戶閱讀,此頁面中文以機器翻譯完成。雖然我們已盡力確保機器翻譯的準確性,但機器翻譯並非完美。如果您對機器翻譯的內容不滿意,請參考英文版本。