Odwrotna transkrypcja
A Technical Guide to PCR Technologies
Jednym z podejść do analizy ekspresji genów jest pomiar stężenia mRNA genu. Istnieje kilka wyzwań związanych z taką analizą, takich jak różnice w okresie półtrwania między różnymi transkryptami, czasowe wzorce transkrypcji i brak korelacji między mRNA a białkiem. Aby przeanalizować RNA przy użyciu metody opartej na PCR, cDNA musi zostać wyprodukowane przy użyciu odwrotnej transkrypcji (RT). Proces ten wykorzystuje enzym odwrotnej transkryptazy i dNTP.
Etap RT może być wykonywany na całkowitym RNA, dzięki czemu wytwarzana jest globalna reprezentacja cDNA wielu transkryptów (zwykle za pomocą dwuetapowego protokołu) lub w podejściu specyficznym dla genu, w którym tylko interesujący RNA jest przekształcany w cDNA (zwykle zgodnie z protokołem jednoetapowym).
Ponieważ wykazano, że dwuetapowa reakcja RT nie zawsze jest liniowa w odniesieniu do wejściowego RNA i wydajności cDNA1, ważne jest, aby określić i kontrolować całkowitą ilość RNA ekstrahowanego i zawartego w reakcjach RT. Pomiar stężenia RNA jest obarczony niepewnością, a wartość bezwzględna zależy od przyrządu lub systemu użytego do wykonania pomiaru. Jak widać na Rysunku 8.1, istnieje duża zmienność między pomiarami stężenia 5 próbek RNA (A-E) (patrz Oczyszczanie próbek i ocena jakości) podczas korzystania z Nanodrop, konwencjonalnej spektrofotometrii UV, Ribogreen, Agilent 2100 BioAnalyzer lub Bio-Rad Experion. Należy zauważyć, że obecność EDTA (próbki D i E), która hamowałaby RT i PCR, oraz degradacja (próbka C) mają różny wpływ na pomiary stężenia w zależności od używanego systemu. Ta obserwacja ilustruje znaczenie wykonywania dodatkowych etapów kontroli jakości przed użyciem próbek w dalszych reakcjach (patrz Oczyszczanie próbek i ocena jakości).

Rysunek 8.1.Stężenie pięciu próbek całkowitego RNA (A-E) zmierzono za pomocą Nanodrop, konwencjonalnej spektrofotometrii UV, Bio-Rad Experion, Agilent 2100 BioAnalyzer (wszystkie zduplikowane pomiary) lub Ribogreen (pojedynczy pomiar). Jak widać, stężenie bezwzględne, jak również stężenie względne różniły się między próbkami. Wynikało to z faktu, że próbka C była zdegradowana, a próbki D i E zawierały EDTA, co powodowało niedokładne pomiary w systemach Experion i BioAnalyzer.
Liniowość odwrotnej transkrypcji.
Względne stężenie całkowitego RNA może wpływać na wydajność RT i stężenie cDNA wytwarzanego z danego transkryptu. W związku z tym pożądane jest uwzględnienie tego samego lub bardzo podobnego stężenia RNA we wszystkich dwuetapowych reakcjach syntezy cDNA, chyba że zweryfikowano, że system RT ma odpowiedź liniową. Jak widać na Rysunku 8.2, przy użyciu konwencjonalnego protokołu RT, 100-krotne rozcieńczenia wejściowego RNA nie skutkują odpowiadającą 100-krotną różnicą w wydajności cDNA dla testowanych szablonów. Co ciekawe, przedstawione dane to zduplikowane qPCR przeprowadzone na zduplikowanych reakcjach RT. Jak pokazano, brak liniowości jest powtarzalny między dwiema reakcjami RT.

Rysunek 8.2.Całkowity RNA rozcieńczono 100-krotnie i poddano odwrotnej transkrypcji przy użyciu dwuetapowego losowego primingu; przeprowadzono dwie niezależne reakcje RT. β-aktynę wykryto w duplikatach qPCR dla każdej reakcji RT. Reakcja RT jest powtarzalna, ale wydajność cDNA nie jest proporcjonalna do stężenia wejściowego RNA. Dlatego też, jeśli ograniczenia eksperymentalne wymagają, aby zmienne stężenie RNA było zawarte w RT, krytyczne jest sprawdzenie, czy protokół i kombinacja odczynników skutkują liniową odpowiedzią.
W przykładzie pokazanym na Rysunku 8.3, odczynnik ReadyScript® RT (RDRT) został użyty do odwrotnej transkrypcji całkowitego RNA z 2-krotnego i 10-krotnego seryjnego rozcieńczenia matrycy przy użyciu dwuetapowego protokołu i kombinacji oligo-dT (O4387) i losowego primingu (opisanego poniżej). Gen CANX wykryto w obu seriach rozcieńczeń z bezpośrednią proporcjonalnością do stężenia wejściowego RNA.

Rysunek 8.3.Odczynnik ReadyScript® RT (RDRT) został użyty do odwrotnej transkrypcji całkowitego RNA z 2-krotnego i 10-krotnego seryjnego rozcieńczenia. Gen CANX został wykryty w obu seriach rozcieńczeń, co skutkowało bezpośrednią proporcjonalnością do stężenia wejściowego RNA (dane z grup studenckich uczestniczących w warsztatach EMBL Advanced qPCR).
Reverse Transcription Priming
Wybór starterów, które zostaną użyte do zainicjowania odwrotnej transkrypcji, może znacząco wpłynąć na wyniki RT-qPCR. W przypadku jednoetapowego
RT-qPCR stosowane są startery specyficzne dla danego genu. Podczas wykonywania testu dwuetapowego można użyć startera odwrotnego specyficznego dla genu, oligo-dT (O4387), losowych heksamerów, nonamerów, dekamerów, dodekamerów lub pentadekamerów2 lub kombinacji oligo-dT (O4387) i losowych starterów. Starterowanie specyficzne dla genu jest zwykle przeprowadzane w oddzielnych reakcjach dla każdego docelowego RNA. Te oddzielne reakcje mogą mieć bardzo różne wydajności, komplikując w ten sposób porównania między stężeniami RNA. Z drugiej strony, przy użyciu starterów specyficznych dla genów, cały produkt RT będzie kodował interesujący gen i może pozwolić na kwantyfikację mRNA o bardzo niskiej liczebności, których nie można wykryć przy użyciu niespecyficznych starterów RT. Aby uniknąć potencjalnie dużych różnic między testami w RT, które mogą wystąpić w przypadku starterów specyficznych dla genu, można użyć niespecyficznych starterów do wygenerowania puli cDNA. Oddzielne testy qPCR dla każdego celu można następnie przeprowadzić z podwielokrotnościami z puli cDNA. Jeśli wszystkie cele qPCR znajdują się w pobliżu 3'-końca poliadenylowanych mRNA, oligo-dT (O4387) jest odpowiednim wyborem startera. Z drugiej strony, jeśli cele qPCR znajdują się więcej niż kilka kilobaz od 3'-końca lub jeśli RNA nie jest poliadenylowany, losowe startery spowodują bardziej niezawodne wykrywanie. Jeśli względna lokalizacja 3' celów qPCR jest różna lub pożądane transkrypty zawierają kombinację poliadenylowanych i niepoliadenylowanych RNA, mieszanina oligo-dT (O4387) i losowych oligomerów da najlepsze wyniki
Priming odwrotnej transkrypcji dla dwuetapowych reakcji RT
Dwa podejścia są powszechnie stosowane do dwuetapowego primingu RT; oligo-dT (O4387) i losowego primingu. Metoda oligo-dT (O4387) polega na hybrydyzacji oligodT (zwykle 15-merowego) do ogona poli-A, obecnego na 3' końcu większości cząsteczek mRNA, w celu primingu i selektywnej odwrotnej transkrypcji mRNA.
Podejście to, choć koncepcyjnie bardzo proste, wiąże się z wyzwaniami: Wiązanie startera oligo-dT (O4387) nie jest specyficzne dla mRNA w temperaturach reakcji stosowanych w RT, a zatem oligo-dT (O4387) będzie wiązać się niespecyficznie z innymi regionami RNA. Dodatkowo, odcinki rRNA są również wykrywane, ponieważ regiony ATrich w tych cząsteczkach są primowane przez oligo-dT (O4387). Niektóre mRNA, takie jak te kodujące histony, nie zawierają ogonów poli A i mogą nie być reprezentowane w wynikowym cDNA.
Druga metoda wykorzystuje losowe gruntowanie. Startery losowe składają się z losowych sekwencji, często z heksamerów (6-merów) lub nonamerów (9-merów). Są one używane do gruntowania reakcji RT, prowadząc do syntezy fragmentów cDNA o różnej długości, które reprezentują oryginalny RNA. Startery losowe hybrydyzują wzdłuż długości transkryptu i są bardziej tolerancyjne na strukturę drugorzędową niż oligo-dT (O4387) lub primery specyficzne dla genów.
Aby skorzystać z zalet poszczególnych technik, niektóre protokoły wymagają połączenia obu typów starterów. Specyficzny starter do sekwencji docelowej może być również stosowany w dwuetapowym protokole RT, ale częściej jest stosowany w procedurze jednoetapowej (patrz poniżej).
Podkłady do odwrotnej transkrypcji dla jednoetapowych reakcji RT
W jednoetapowym protokole RT startery specyficzne dla genów są używane do odwrotnej transkrypcji pojedynczego celu. Projekt startera specyficznego dla genu jest krytyczny; musi on znajdować się w otwartym, dostępnym regionie docelowego mRNA, gdy jest przewidywany w temperaturze reakcji RT. W tych warunkach istnieje liniowa zależność między wejściowym RNA a cDNA (rysunek 8.4). Starter ten może być (i zwykle jest) wspólny ze starterem PCR.

Rysunek 8.4.Całkowity RNA rozcieńczono w serii 10-krotnych rozcieńczeń i przeprowadzono jednoetapową reakcję RT-qPCR w celu wykrycia GAPDH. Każdą reakcję przeprowadzono w trzech powtórzeniach. Zaowocowało to liniową zależnością między stężeniem RNA a wydajnością cDNA (zdjęcie dzięki uprzejmości prof. Stephena Bustina, Anglia Ruskin University, Wielka Brytania).
Reakcja odwrotnej transkrypcji jest procesem wysoce zmiennym i należy wziąć pod uwagę wszystkie etapy, aby utrzymać składniki reakcji na możliwie stałym poziomie. Przeprowadzenie jednoetapowej reakcji RT-qPCR zmniejsza liczbę wymaganych etapów pipetowania i zmniejsza błąd. Może to być zatem metoda z wyboru, jeśli trzeba zmierzyć małe różnice, a dokładność jest najważniejsza. Jednak przeciwną kwestią jest określenie stosunku genu zainteresowania (GOI) do genu referencyjnego (patrz Analiza danych) wymaga dwóch oddzielnych jednoetapowych reakcji RT zamiast pojedynczego cDNA z dwuetapowej reakcji, a dwóch celów nie można wykryć przy użyciu podejścia multipleksowego qPCR.
Wydajność odwrotnej transkrypcji
Zwykle zakłada się, że całe RNA/mRNA w reakcji RT jest przekształcane w cDNA i że wszystkie transkrypty są przekształcane w stosunku 1:1 lub proporcjonalnie do początkowego stężenia RNA. Niedawno przeprowadzono badania w celu zbadania każdego z tych założeń. Oczywiste jest, że ilość RNA przekształcanego w cDNA jest bardzo zmienna. Dwuetapowy proces RT jest zmienny i zależy w szczególności od stężenia RNA, enzymu, składu buforu i protokołu primingu. Ponieważ proces jest zmienny, ważne jest, aby utrzymać jak najwięcej stałych warunków1,3,5.
W przypadku dwuetapowych reakcji RT, ogólnie rzecz biorąc, konieczne jest dążenie do tego samego stężenia wejściowego RNA, a także utrzymanie stałych warunków primingu, enzymu RT i buforu. Gdy nie można określić stałego stężenia wejściowego, zaleca się zastosowanie procesu jednoetapowego, włączenie nośnika, takiego jak glikol polietylenowy (PEG)6, lub wybranie komercyjnego zestawu, który został zatwierdzony jako zapewniający liniową odpowiedź, takiego jak ReadyScript® RT.
Wybór strategii primingu wpływa zarówno na bezwzględną wydajność, jak i stosunek różnych docelowych cDNA z całkowitej próbki RNA. Rysunek 8.5 przedstawia wydajność liczby kopii cDNA dla trzech różnych genów (pokazanych kolorem histogramu) w próbce RNA, która również została poddana kontrolowanej, enzymatycznej degradacji. Oczywiste jest, że różne metody primingu skutkują różnymi bezwzględnymi liczbami kopii i stosunkami między próbkami. Ponadto priming specyficzny dla genu wydaje się być bardziej tolerancyjny na degradację RNA niż priming oligo-dT.

Rysunek 8.5.Całkowite RNA inkubowano na nagiej ludzkiej dłoni przez wskazane czasy (1, 2,5, 5 i 7,5 min). cDNA przygotowano przy użyciu primingu specyficznego dla genu, losowego lub oligo-dT (O4387). Określono liczbę kopii trzech genów, jak wskazano na fioletowym, turkusowym i pomarańczowym histogramie. Oczywiste jest, że różne strategie primingu wpływają na wykrywanie każdego genu z primingiem oligo-dT (O4387), co skutkuje brakiem wykrycia genu 3 (pomarańczowego), który jest wyraźnie widoczny w próbce RNA, ponieważ jest wykrywany przy użyciu primingu specyficznego dla genu (dane dostarczone przez Prof. Stephen Bustin, Anglia Ruskin University, UK).
Temperatura stosowana w reakcjach RT może wpływać na specyficzność, zwłaszcza podczas hybrydyzacji starterów specyficznych dla genów. Primery, które mogą tworzyć silny 3'-dupleks, będą łatwiej hybrydyzować w niższych temperaturach. Ponieważ enzymy RT mogą rozciągać się od startera DNA na matrycy DNA, tworzenie dimerów starterów może rozpocząć się podczas etapu RT. Zwiększenie temperatury inkubacji RT do najwyższej temperatury, w której enzym jest w pełni aktywny lub użycie enzymu wysokotemperaturowego może zmniejszyć ilość produktu primer-dimer. Startery zastosowane na Rysunku 8.6 dały znacznie bardziej specyficzny produkt w stosunku do produktu niespecyficznego po jednoetapowej RT-qPCR, gdy RT przeprowadzono z Moloney Murine Leukemia Virus-Reverse Transcriptase (MMLV-RT) w temperaturze 45 °C (Rysunek 8.6B) niż gdy RT przeprowadzono w temperaturze 37 °C (Rys. 8.6A). Podobnie, wykonanie dwuetapowego RT-qPCR z niespecyficznym starterem do RT i polimerazą Hot Start Taq do qPCR skutkowało mniejszą ilością produktu primer-dimer (Rys. 8.6D) niż przy użyciu jednoetapowego RT-qPCR ze starterami specyficznymi dla genu, które mogą tworzyć 3'-dupleks (Rys. 8.6B).
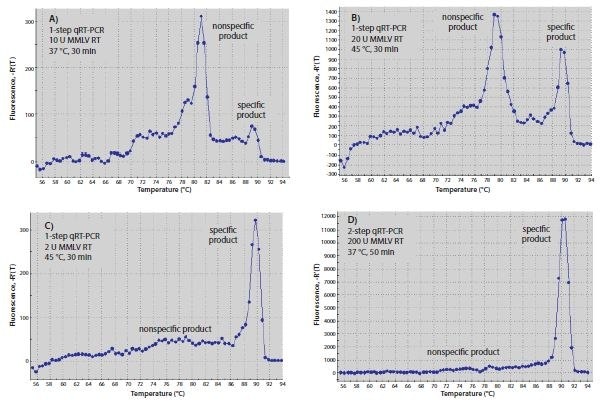
Rysunek 8.6.Optymalizacja RT. (A-C) Krzywe topnienia produktów RT-qPCR wytworzonych za pomocą jednoetapowego lub (D) dwuetapowego RT-qPCR. Reakcje A-C zawierały po 10 μL SYBR® Green JumpStart™ Taq ReadyMix™, 0,02 μL barwnika referencyjnego, oba startery specyficzne dla genów w stężeniu 0,4 μM i 10 ng ludzkiego całkowitego RNA w końcowej objętości 20 μL. Startery specyficzne dla genów to 5'-CGGCTTCAACGCAGACTA-3' i 5'-CTGGTCGAGATGCAGTGA-3' dla c-fos (przystąpienie NM_005252). Reakcje A i B zawierały również 20 jednostek MMLV- RT, podczas gdy reakcja C zawierała 2 jednostki. Reakcję A inkubowano w temperaturze 37 °C przez 30 minut przed qPCR, podczas gdy reakcje B i C inkubowano w temperaturze 45 °C przez 30 minut przed qPCR. W D reakcja RT zawierała 1x bufor MMLV (nr produktu D8559), 0,5 mM dNTP, 1 μM oligo-dT (O4387), 0,8 jednostek/μL inhibitora RNazy, 200 jednostek MMLV-RT i 10 ng ludzkiego całkowitego RNA w końcowej objętości 20 μL. Reakcję inkubowano w temperaturze 25°C przez 10 minut, 37°C przez 50 minut i 80°C przez 10 minut. 2 μL produktu reakcji RT dodano do qPCR zawierającego 10 μL SYBR® Green JumpStart™ Taq ReadyMix™ 0,02 μL barwnika referencyjnego i oba startery specyficzne dla genu w stężeniu 0,4 μM, jak w przypadku reakcji jednoetapowych (A-C). Wszystkie reakcje qPCR inkubowano w temperaturze 94 °C przez 3 minuty w celu denaturacji, a następnie przez 40 cykli 94 °C przez 15 sekund i 60 °C przez 1 minutę.
Ilość enzymu RT na reakcję może również wpływać na wyniki RT-qPCR. Jak pokazano na Rysunku 8.6, jednoetapowe reakcje z 2 jednostkami MMLV-RT (Rysunek 8.6C) były bardziej specyficzne niż reakcje z 20 jednostkami (Rysunek 8.6B). Dwuetapowa RT-PCR z użyciem oligo-dT (O4387) lub losowych starterów dla RT, skutkowała większą specyficznością niż jednoetapowa RT-PCR (Rys. 8.6D). Można to przypisać faktowi, że startery specyficzne dla genu nie są obecne podczas reakcji RT w niskiej temperaturze, zapobiegając w ten sposób tworzeniu się niespecyficznych produktów. Wyższe stężenia RT mogą dawać lepsze wyniki w reakcjach dwuetapowych, ale ponieważ enzym RT może zakłócać aktywność polimerazy Taq DNA7, ilość produktu RT przeniesionego do qPCR powinna być ograniczona do nie więcej niż 10% końcowej objętości reakcji. Wyjątkiem od tego zalecenia jest użycie ReadyScript, w którym 25% objętości PCR może być reakcją RT bez wpływu na wydajność PCR.
Ten opis zmiennych, które są nieodłącznie związane z procesem RT, pokazuje, że określenie ekspresji genów
na podstawie RT zależy od zastosowanej metody RT, ilości i jakości próbek, oprócz uwzględnienia ilości matrycy i powinno być starannie raportowane, jak opisano w wytycznych MIQE8 (patrz Quantitative PCR).
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?