Locked Nucleic Acid 和 Minor Groove Binder / Elipse Dark Quencher (MGB:EDQ) 是可增強寡核苷酸性能的修飾。它們可增加穩定性,並改善與目標序列進行碱基配对的特異性和親和力。鎖定核酸可增強 qPCR 中引物和探針(與 BHQ™ 結合時)以及反意寡核苷酸 (ASO) 等治療方式的效果。同時,MGB:EDQ 含有淬滅劑,可特別增強 qPCR 中探針的效果。
Read more about
鎖定核酸
鎖定核酸是一種新型的核酸類似物,含有 2'-O, 4'-C 亞甲基橋 (圖 1)。此橋鎖定在 3'-endo 結構中,限制了核呋喃糖環的彈性,並將結構鎖定為剛性的雙環結構。這可增強檢測性能並擴大應用範圍。

圖 1.鎖定核酸和原生狀態 DNA 單體的結構。
PCR 引物、qPCR 探針和其他類型寡核苷酸中的鎖定核酸可溶解於水和標準緩衝液,並且遵循 Watson-Crick 碱基配对規則。1
鎖定核酸的優點
鎖定核酸納入寡核苷酸時,與僅原生狀態的 DNA 基相比,具有以下幾項優點:
- 增加熱穩定性和雜交特異性
- 更精確的基因量化和等位基因分辨
- 更容易和更靈活地設計有問題的目標序列
增加熱穩定性和雜交特異性
在寡核苷酸中加入鎖定核酸可增加雙工體的熱穩定性2 並改善寡核苷酸與目標序列雜交的特異性。3就 qPCR 而言,這可減少因假結合而產生的背景螢光,從而提高信噪比。此外,PCR 引物或 qPCR 探針與其目標的雜交改善,可能會使熔解溫度 (Tm) 增加高達 8 ° C。C (表 1)。這種雜交的增加大大拓寬了檢測條件的範圍,並允許更成功的多重化5
; (表 1)。更精確的基因定量和等位基因區別
透過 SNPs 區別等位基因的寡核苷酸能力,透過鎖定核酸碱基的加入而大幅提升6-8 (圖 2)。與原生狀態的 DNA 寡核苷酸相比,單一碱基錯配的存在對鎖定核酸寡核苷酸及其目標之間的雙工形成具有更大的不穩定作用。包含鎖定核酸碱基的較短寡核苷酸可在與較長的原生態 DNA 寡核苷酸相同的溫度下使用。

圖 2.在 SNP 基因分型分析中,鎖定核酸雙標示探針比 DNA 雙標示探針有更好的分辨能力9。粉紅色)使用鎖定核酸突變探針(16mer,含 3 個鎖定核酸基序)進行突變 DNA 分析。綠色)用原生態 DNA突變探針(25mer)分析突變 DNA。紅色)野生型 DNA 與原生態 DNA突變探針(25mer)。紫色)野生型 DNA 與鎖定核酸突變探針(16mer,含 3 個鎖定核酸碱基)。
更輕鬆、更靈活地設計有問題的目標序列
由於鎖定核酸的雜交特性隨著Tm,鎖定核酸寡核苷酸可以合成得更短,這克服了原生狀態 DNA 寡核苷酸的某些設計限制。具體來說,鎖定核酸寡核苷酸可以設計來處理傳統上有問題的目標序列,例如富含 AT 或 GC 的區域。例如,富含 AT 的原生態 DNA 寡核苷酸通常需要超過 30 個碱基(有時超過 40 個碱基)的長度,才能符合擴增子設計準則,但性能可能仍然不佳。有了鎖定核酸寡核苷酸,鎖定核酸碱基的選擇性放置有助於優化高特異性、較短寡核苷酸的設計,即使只有 13 到 20 個碱基的長度,也能有良好的表現。此外,鎖定核酸也大大方便了針對困難 SNP(例如相對穩定的 G:T 錯配)的寡核苷酸設計。不需要專用的儀器。
鎖定核酸的應用
鎖定核酸可以整合到所有可用的&.nbsp;qPCR檢測化學試劑,包括:
- SYBR® 綠色引物
- 雙標示探針
- 分子信標 ® 探針
- Scorpions® 探針
它對以下方面很有用:
- SNP檢測
- 等位基因分辨
- 病原檢測
- 多重複合
- 基因表達分析
- 基因拷貝測定
它還可以用於這些序列:
- 反義寡核苷酸
- 誘捕寡核苷酸
- 捕獲探針
- 適應子
- 誘酶
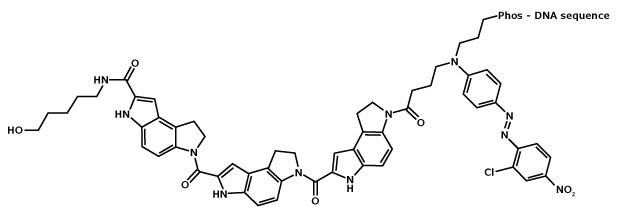
圖 1.MGB:EDQ 的結構。MGB 或 CDPI3 是二氫吡咯吲哚-羧酸的三肽,其回旋槳曲度可使其與 B 型 DNA 的小溝形狀一致,從而與小溝強烈結合。
1. 溶液中的探針會發出低螢光
。2.水解釋放螢光
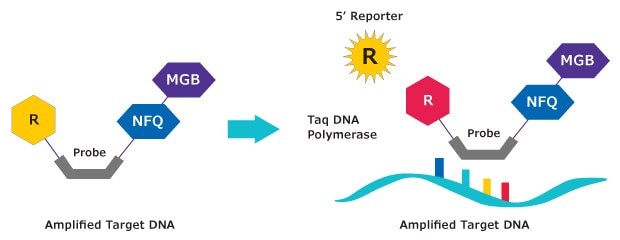
圖 2.作用機制。MGB:EDQ 探針是水解探針。引物被聚合酶拉長,探針與特定的 DNA 模板結合。水解會釋放探針/目標雜交體中的報告器,導致螢光增加。測得的螢光信號與目標 DNA 的數量成正比。
MGB 的優點
MGB 從幾個方面增強了 qPCR 的性能:
- 增加特異性:MGB 增加了探針與目標雜交的特異性,降低了非特異擴增的可能性
- 靈敏度更高:MGB 可以使用更短的探針,從而提高 qPCR 分析的靈敏度
- 熔融溫度調整:MGB 允許調整探針的 Tm,有助於設計具有最佳 Tm 值的 qPCR 探針
- 降低背景信號:MGB 有助於減少背景螢光,從而改善 S:N 或信噪比
- 增強穩定性:MGB 可以提高探針-目標雜交的穩定性,降低探針降解的可能性
- 設計靈活性:使用 MGB 擴大了探針設計的選擇範圍,有助於創建高效的 qPCR 分析
MGB 增強的 qPCR 探針是獲得可靠、穩健的 qPCR 結果的重要工具。
APPLICATIONS OF MGB:EDQ
MGB:EDQ 可以整合到此 qPCR 檢測化學中:
- 雙標示探針(含 6-FAM™、HEX™ 和 TET™;其他請咨詢)
它對以下方面很有用:
- 透過錯配判別進行SNP偵測
參考資料
若要繼續閱讀,請登入或建立帳戶。
還沒有帳戶?