Fehlerbehebung in der RT-PCR/ RT-qPCR
Auf dieser Seite
- Entwicklung eines Protokolls für die Fehlerbehebung
- Optimierung des Oligo-Designs
- PCR-Assay-Optimierung
- Assay-Planung in der RT-PCR und qPCR
- Qualität des RNA- oder DNA-Templates
- Assay PCR-Programm
- Fehlfunktion des Thermocyclers
- Beispiele zur Fehlerbehebung: Werkzeuge für die Diagnostik
- Dissoziations- / Schmelzkurven
- Fallstudien zur Fehlerbehebung in der RT-PCR
- Zusammenfassung - PCR-Checkliste für die Fehlerbehebung
Entwicklung eines PCR- oder RT-PCR-Protokolls für die Fehlerbehebung
Mögliche Fehlerquellen und/oder Probleme durch Bedienerfehler
Es gibt viele Möglichkeiten für Bedienerfehler. Die Ursachen für diese Fehler bleiben oft unerkannt. Der erste Schritt bei der Fehlerbehebung besteht darin, das Protokoll zu überprüfen und den Versuch zu wiederholen. Es ist wichtig, das Protokoll zu überprüfen (siehe Anhang A, Protokolle, in diesem Leitfaden) und einen erfahrenen Molekularbiologen zu bitten, den Versuchsplan zu überprüfen. Die unglaubliche Geschichte von einem Postdoktoranden, der mehrere fehlgeschlagene PCR durchgeführt hat, bevor er bemerkt hat, dass die dNTP in dem PCR-Mastermix gefehlt haben, macht deutlich, dass selbst die besten Wissenschaftler einfache Fehler machen können.
Mastermix
Fehler oder Probleme mit dem Mastermix der Reaktionskomponenten können die Ursache für ein katastrophales Versagen der Amplifikation in allen Proben und Positivkontrollen sein. Überprüfen Sie vor deshalb vor der Wiederholung von Versuchen alle Komponenten und ihre Konzentrationen. Wenn eine neue Reagenziencharge verwendet wird, ist es sinnvoll, das neue Reagenz im Vergleich mit dem alten durchlaufen zu lassen, bevor eine größere Versuchsreihe gestartet wird.
Beim Wechsel von Mastermix-Produkten ist es wichtig zu wissen, dass einige Assays besonders empfindlich auf Kombinationen aus Pufferzusammensetzung/Hybridisierungstemperatur (Ta) und Primerkonzentration reagieren. Die Änderung eines dieser Elemente kann zu einer abweichenden Leistung führen. Überprüfen Sie daher alle Assays in ausgewählten Mastermixen und auf allen gewünschten Geräten, bevor grundlegende Änderungen vorgenommen werden. Es ist darüber hinaus wichtig, die mit jedem Mastermix gelieferten Anweisungen zu prüfen, da sie die empfohlenen Bedingungen spezifizieren, die für das jeweilige Enzym, den Hot-Start-Mechanismus und die Pufferkomponenten optimiert sind.
| Problemstellung | JumpStart™ Taq ReadyMix™ funktioniert nicht so gut wie ein ähnliches Produkt eines anderen Anbieters |
|---|---|
| Mögliche Ursachen |
|
| Diagnostischer Test |
|
| Lösung |
|
| Problemstellung | PCR ReadyMix funktioniert für die PCR, aber nicht für qPCR. Gleichwertige Produkte von einem anderen Anbieter funktionieren gut |
|---|---|
| Mögliche Ursachen |
|
| Diagnostischer Test |
|
| Lösungen |
|
Es ist gute Laborpraxis sicherzustellen, dass eine ausreichende Menge an Reaktions-Mastermix für alle Proben vorbereitet wird, die zusammen durchgeführt werden sollen. Stellen Sie sicher, dass alle Komponenten sorgfältig aufgetaut und gut gemischt sind und dass das Mastermix für den Versuch sehr gut gemischt ist, bevor die Proben aliquotiert werden. Dies gilt insbesondere für einige der 2×-Puffer wie KiCqStart®, die zähflüssiger sind als normale PCR-Puffer.
Oligo-Optimierung
Oligos können Probleme verursachen, wenn sie eine falsche Sequenz oder ein schlechtes Design haben, in einer suboptimalen Konzentration oder einer suboptimalen Ta laufen oder nicht ausreichend markiert oder gequencht sind (bei Sonden). Ein Assay, der unter suboptimalen Bedingungen für das Oligo oder unter Verwendung eines schlechten Designs durchgeführt wird, kann zwar einige Daten liefern, diese spiegeln jedoch möglicherweise nicht die tatsächliche Biologie wider, die untersucht wird. Bei Erhalt eines gefriergetrockneten Oligos ist es wichtig:
- Die Sequenz zu verifizieren
- Sicherzustellen, dass die gesamte DNA vor der Verwendung resuspendiert wird
- Zu bestätigen, dass die Lösung die erwartete Konzentration aufweist
Resuspendieren Sie die Oligos, indem Sie das Oligo 5 Minuten lang auf 90 °C erhitzten und dann gut mischen. Wiederholte Gefrier-Auftau-Zyklen können ebenfalls die Leistung des Oligos beeinträchtigen. Daher sollen alle Oligos in einer Stammkonzentration (in der Regel 100 μM) aliquotiert und bei -20 °C bzw. langfristig bei -80 °C gelagert werden.
Während der Fehlerbehebungsphase ist es wichtig, zu überprüfen, ob die richtige Sequenz bestellt wurde, indem man zur Zielsequenz zurückkehrt und bestätigt, dass die Oligosequenzen tatsächlich vorhanden sind. Vergewissern Sie sich, dass die Oligoqualität korrekt war, indem Sie sich mit dem Oligoanbieter in Verbindung setzen. Messen Sie die Arbeitskonzentration des Oligos und überprüfen Sie die fluoreszierenden Moleküle visuell, um zu bestätigen, dass diese markiert sind. Testen Sie die Primer der Sondenassays in einem SYBR® Green I qPCR-Mix, um die Amplifikation zu überprüfen. Ziehen Sie eine Optimierung der Primerkonzentrationen oder des Ta-Werts in Betracht (siehe Assay-Optimierung und Validierung). Wenn Sie eine Sonde zum ersten Mal verwenden, sammeln Sie Fluoreszenzdaten für so viele potenzielle Wellenlängen wie möglich, damit eventuelle Signalverluste zwischen den Kanälen beobachtet und Fehler in der Markierung erkannt werden können.
Unzureichende PCR-Optimierung
Die Auswirkungen der Assay-Optimierung wurden in Assay-Optimierung und Validierung beschrieben und dargelegt. Wenn ein Assay fehlschlägt oder suboptimal abläuft, aber keine Fehler in der Planung oder in den Arbeitsabläufen vorliegen, kann dieser von einer Optimierung der Versuchsbedingungen profitieren. Testen Sie bei der Fehlerbehebung die Primer bei 100 nM, 500 nM und 900 nM Endkonzentration und/oder einem Ta-Wert zwischen 55 °C und 70 °C (unter Verwendung eines Temperaturgradienten), um festzustellen, ob sich der Assay durch weitere Optimierung verbessert.
Assay-Planung in der RT-PCR und qPCR
Die Assay-Planung wurde in Assay-Planung für PCR/qPCR/dPCR beschrieben. Stellen Sie bei der Fehlerbehebung in einem Assay sicher, dass die Planung überprüft wurde. Stellen Sie sicher, dass die PCR/qPCR-Primer und die Amplikonposition mit dem RT-Priming-Protokoll übereinstimmen. Stellen Sie zum Beispiel sicher, dass Assays, die auf cDNA angewendet werden, die nach Oligo-dT-Priming hergestellt wurde, in Richtung 3' des Transkripts liegen. Stellen Sie darüber hinaus sicher, dass die Sequenzinformationen zuverlässig sind und dass geeignete Spleißvarianten und SNP berücksichtigt wurden.
| Problemstellung | Der Assay ist unsensibel und die Amplifikationskurven sehen ungewöhnlich aus (Abbildung 11.1) |
|---|---|
| Mögliche Ursachen |
|
| Diagnostischer Test |
|
| Lösungen |
|

Abbildung 11.1A.Der Assay weist ein ungewöhnliches Profil der Amplifikationskurven mit einem ausgeprägten Drift der Basislinie auf.

Abbildung 11.1B.Die Sequenz für die Sonde, die in den Assay aufgenommen wurde, wurde in die mfold-Software zur Faltungsvorhersage eingegeben. Es ist klar, dass die Sonde in Lösung eine stabile gefaltete Struktur annehmen könnte, was wahrscheinlich zu dem beobachteten Problem führt.
Qualität des RNA- oder DNA-Templates
Die Auswirkung der Templatequalität auf die Assay-Leistung wurde in Probenaufreinigung und Qualitätsbewertung beschrieben. Die Qualität des Templates umfasst die Quantität, die Integrität und das Vorhandensein von Inhibitoren. Es ist von entscheidender Bedeutung, dass die RNA-Qualität auf das am besten geeignete RT-Priming-Protokoll abgestimmt ist (siehe Reverse Transkription) und dass das bestmögliche Template verwendet wird. Ebenso muss die RNA-Menge, die den RT-Reaktionen zugesetzt wird, im Umfang des Protokolls liegen, und in vielen Fällen sollte diese für alle Reaktionen gleich sein. ReadyScript® ist eine bemerkenswerte Ausnahme von dieser Richtlinie, da die Verwendung dieses Reagenz und Protokolls eine lineare cDNA-Konzentration ergibt, die proportional zur Menge der Eingabe-RNA ist. Bei der Fehlerbehebung in einer Probe, die einen höheren als den erwarteten Cq,-Wert aufweist, führen Sie den SPUD-Assay durch oder verdünnen die Probe durch eine 1:5- oder 1:10-Verdünnungsreihe und wiederholen den Assay (Abbildung 11.2), um Proben zu identifizieren, die Inhibitoren enthalten.
| Problemstellung | Die Cq-Daten für Verdünnungen der Standardkurven weisen unregelmäßige Abstände auf (Abbildung 11.2) |
|---|---|
| Mögliche Ursachen |
|
| Diagnostischer Test |
|
| Lösungen |
|


Abbildung 11.2.Amplifikation einer 10-fachen seriellen Verdünnung eines DNA-Templates. Die Replikate sind präzise, aber der ΔCq-Wert ist inkonsistent und nimmt mit zunehmender Verdünnung ab. Die Daten zeigen auch ein positives Signal in der Negativkontrolle (NTC), was auf eine Kontamination oder Primer-Dimer-Bildung hinweist, und dass die Verdünnungen auf weniger als 105 Kopien identische Daten wie die NTC aufweisen.
| Verdünnung | ΔCq |
|---|---|
| 107 → 106 | 5,4 |
| 106 → 105 | 5,0 |
| 105 → 104 | 3,6 |
Auch die Anzahl der Templates ist ein nicht zu vernachlässigender Aspekt. Die Aufnahme einer zu hohen oder zu niedrigen Template-Menge in die PCR führt zu fehlgeschlagenen Reaktionen und qPCR-Amplifikationskurven, die abnormal sind. Abbildung 11.3A zeigt eine Reaktion mit einer 10-fachen seriellen Verdünnung eines künstlichen Oligo-Templates. Die niedrigeren Verdünnungen sind zu konzentriert für einen effizienten Reaktionsablauf oder eine effiziente Verarbeitung der Grundliniendaten durch das Gerät (Abbildung 11.3B), was in abnormalen Amplifikationskurven und unzuverlässigen Daten resultiert.

Abbildung 11.3. A)Amplifikation einer 10-fachen seriellen Verdünnung eines künstlichen Templates mit spezifischen Primern und einer FAM-markierten Sonde. Der Cq-Wert ist bei den konzentrierten Proben sehr niedrig, die Amplifikationskurven weisen keine regelmäßigen Abstände auf und sind abnormal. B) Zeigt die Rohdaten für diese Amplifikationskurven auf. Die Reaktionen mit der höchsten Konzentration des Ziels weisen auch eine deutlich höhere Hintergrundfluoreszenz und eine minimale Fluoreszenzausbeute während der Reaktion auf.
Assay PCR-Programm
Die PCR-Zyklusbedingungen müssen sowohl für den Versuch als auch für die Reagenzien geeignet sein (siehe z. B. Mastermix). Es ist nicht ratsam, die Standardeinstellungen des Geräts ungeprüft zu übernehmen.
Fehlfunktion des Thermocyclers
Gerätefehler können schleichend auftreten und daher schwer zu diagnostizieren sein. Es ist wichtig sicherzustellen, dass alle Bediener von Geräten umfassend geschult und anfänglich beaufsichtigt werden, damit hohe Reparaturkosten vermieden werden. Einige Gerätefehler führen zu schwerwiegenden Ausfällen, sodass ggf. keine Amplifikation oder Fluoreszenzdaten vorliegen, während in anderen Fällen die Daten verzerrt oder die Proben uneinheitlich behandelt sind, wodurch künstliche Unterschiede zwischen identischen biologischen Proben entstehen. Die Verwendung von Kontrollproben mit Kontrollassays ist für die Fehlerbehebung von unschätzbarem Wert. Wenn ein Gerätefehler vermutet wird, sollte ein zuverlässiger, optimierter Assay in allen Wells durchgeführt werden. Durch diese Homogenitätsprüfung werden Probleme aufgedeckt, die für bestimmte Bereiche des Geräts spezifisch sind und, darüber hinaus, separate Probleme mit dem Assay und dem Gerät.
Beispiele für die Fehlerbehebung in der PCR zur Demonstration der Verwendung der Diagnosewerkzeuge
Nach der Durchführung einer gut geplanten PCR stehen mehrere Diagnosewerkzeuge für die Fehlerbehebung zur Verfügung:
- Kontrollproben und Assays
- Gel-Endpunkt-/SYBR Green I-Farbstoffreagenz
- Amplifikationskurven (Replikate und Profil der Amplifikationskurve prüfen)
- Standardkurven (Gradient und R2)/Verdünnungsreihen
- Schmelz-/Dissoziationsdiagramme (SYBR Green I-Farbstoff, Molecular Beacons, Scorpions® Sonden)
- Rohdaten/Mehrkomponentenansichten
Kontrollproben/Reaktionen
Die Verwendung von Kontrollen wird dringend empfohlen. Es ist nahezu unmöglich, einen fehlgeschlagenen Assay ohne Informationen aus einer geeigneten Reihe von Kontrollen zu beheben.
| Kontrolle | Beispielmaterial | Erwartetes Ergebnis | Mögliche Gründe für ein positives Ergebnis | Mögliche Gründe für ein negatives Ergebnis |
|---|---|---|---|---|
| Positive Probe | Eine Probe, von der bekannt ist, dass sie die Assay-Sequenzen enthält, wie z. B. RNA/gDNA, die das Ziel exprimiert/enthält | Positiv | Richtig | Fehlgeschlagener Assay. Alle positiven Daten aus anderen Proben sind unzuverlässig. |
| Positive Assaykontrolle | Jede Nukleinsäure, die mit dem Aufbau des PCR-Assays kompatibel ist, wie z. B. ein künstliches Oligonukleotid oder Plasmid, welches das PCR-Amplikon enthält. | Positiv | Richtig | Fehlgeschlagener Assay. Alle positiven Daten aus anderen Proben sind unzuverlässig. |
| Negativkontrolle | Eine Probe, von der bekannt ist, dass sie die Assay-Sequenzen nicht enthält, wie z. B. RNA/gDNA, die das Ziel nicht exprimiert/nicht enthält. | Negativ | Der Assay ist unspezifisch oder die Kontrolle wurde während der PCR-Vorbereitung kontaminiert | Richtig |
| Kontamination der Negativkontrolle des Assays (NTC) | Wasser | Negativ | Die Primer sind Selbstdimere, was zu einem Primer-Dimer-Produkt führt, oder es gab eine Kontamination der Kontrolle während der PCR-Vorbereitung. | Richtig |
| RT-spezifische Kontrollen | Beispielmaterial | Erwartetes Ergebnis | Mögliche Gründe für ein positives Ergebnis | Mögliche Gründe für ein negatives Ergebnis |
|---|---|---|---|---|
| Minus RT-Enzym Negativkontrolle | RNA-Probe und alle Komponenten der RT-Reaktion mit Ausnahme des RT-Enzyms. Dies soll bei allen Proben durchgeführt werden, um sicherzustellen, dass sie keine Sequenzen enthalten, die unter PCR-Bedingungen ohne erforderliche RT amplifiziert werden, z. B. gDNA-Kontaminationen. | Negativ | Die Probe enthält gDNA. Die Reaktion wurde während des Aufbaus kontaminiert. Die Primer haben Primer-Dimer-Produkte gebildet. In Verbindung mit der NTC analysieren. | Richtig |
| Genotypisierungsspezifisch | Beispielmaterial | Erwartetes Ergebnis | Mögliche Gründe für ein positives Ergebnis | Mögliche Gründe für ein negatives Ergebnis |
|---|---|---|---|---|
| Assay-spezifisches positives Ziel. | Das Wildtyp-Template (WT) wurde mit dem WT-Assay und das mutierte Ziel mit dem Mutanten-Assay nachgewiesen. | Positiv | Richtig |
|
| Assay-spezifisches negatives Ziel | WT-Template wurde mit dem Mutanten-Assay und das mutierte Ziel mit dem WT-Assay nachgewiesen. | Negativ (oder relativ hoher Cg-Wert im Vergleich zur Positivkontrolle). |
| Richtig |
| Heterozygotie-Kontrolle im Assay | Heterozygoten-Template oder eine 1:1-Mischung jeder Homozygote, die mit dem WT-Assay und mit dem Mutanten-Assay nachgewiesen wurde. | Positiv bei beiden Assays. | Richtig |
|
| Problemstellung | Die positive Kontrolle amplifiziert, aber es gibt keine Amplifikationsergebnisse aus einer Probe, von der bekannt ist, dass sie ein Ziel enthält (Abbildung 11.4A) |
|---|---|
| Mögliche Ursachen |
|
| Diagnostischer Test |
|
| Lösungen |
|

Abbildung 11.4. A)Das unverdünnte Template kann nicht amplifiziert werden, während Verdünnungen eine erhöhte Amplifikationseffizienz aufweisen. B) Die Zugabe von 0,3 % BSA zum qPCR-Mix unterstützt die Amplifikation aus dem unverdünnten Template.
Die Untersuchung eines vollständig fehlgeschlagenen Assays kann schwierig sein, da nur wenige Informationen für die Fehlerbehebung zur Verfügung stehen. Da viele Assay-Ausfälle auf einen schwerwiegenden Fehler zurückzuführen sind, sollte die erste Kontrolle darin bestehen, den Versuchsaufbau zu überprüfen und dann die PCR zu wiederholen. Gelingt dies nicht, sind für den Prozess der Fehlerbehebung Informationen zu den einzelnen Komponenten des Versuchs erforderlich (Abbildung 11-5).

Abbildung 11.5.Grundlegender Prozess der Fehlerbehebung in der PCR.
Wenn ein qPCR-Versuch vollständig fehlschlägt, besteht der erste Schritt darin, die Planung des Assays, die Oligosequenzen und die QC-Daten des Oligo-Herstellers zu überprüfen. Auch wenn der Assay fehlgeschlagen ist, können qPCR-Multikomponenten-/-Rohdaten verwendet werden, um weitere Informationen zu erhalten. In Abbildung 11.6A wird die Rohdatenkurve für zwei Assays dargestellt, die entweder eine 6-FAM™- oder eine HEX™ (VIC®)-markierte Sonde enthalten. Obwohl beide Assays eine Amplifikation aufweisen, ist das HEX-Signal etwa halb so groß wie das FAM-Signal. Da es sich um einen von Natur aus schwächeren Farbstoff handelt, ist dies eine normale Beobachtung. In der Agarosegelanalyse (Abbildung 11.6B) wird deutlich, dass beide Reaktionen eine ähnliche Produktkonzentration ergeben, was die Beobachtung unterstützt, dass die Cq-Werte der qPCR ähnlich sind.

Abbildung 11.6. A)Die Rohdatenkurven eines Duplex-Assays mit einer FAM- und einer HEX-markierten Sonde. Die FAM-Sonde weist naturgemäß eine höhere Fluoreszenz auf. B) Durch das Agarosegel wird deutlich, dass in jeder Reaktion die gleichen Produktmengen entstanden sind, und es wird die Beobachtung der qPCR Cq bestätigt.
Die Prüfung der Rohdaten ist eine nützliche Kontrolle, um sicherzustellen, dass die Sonde korrekt markiert ist und der Reaktion zugesetzt wurde. In Abbildung 11.7 werden die Rohdaten für die Amplifikation von drei Zielen in einem Dreifachversuch dargestellt. Die für jedes Ziel spezifischen Sonden sind mit FAM, HEX und TAMRA markiert. Die HEX- und TAMRA-Sonden weisen einen geringen Hintergrund und eine effiziente Amplifikation auf, während das FAM-Signal während des gesamten Versuchs konstant hoch ist und keine Anzeichen einer Amplifikation aufweist. Dies stimmt mit einer zu hohen Sondenkonzentration in der Reaktion oder einem Fehler in der Sonde überein, sodass es kein anfängliches Quenchen des Signals gibt. In diesen Fällen sollen die Sondenkonzentration und die Assay-Planung überprüft werden, um sicherzustellen, dass die Sonde über eine kompatible Markierung und einen kompatiblen Quencher verfügt, und ggf. eine neue Sonde getestet wird.
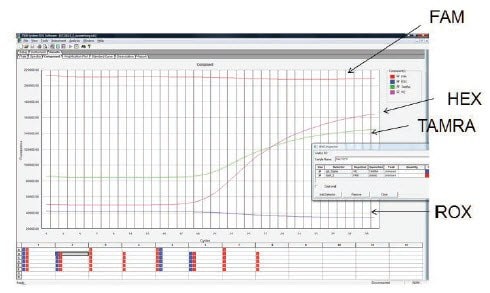
Abbildung 11.7.In einer Triplex-Reaktion wurden drei Ziele mit Sonden nachgewiesen, die mit FAM, HEX und TAMRA markiert waren. Bei den HEX- und TAMRA-Sonden wurde eine Amplifikation aus den Zielen erreicht, während die FAM-Sonde keine Amplifikation aufwies. Die Prüfung der Rohdaten ergab, dass die Hintergrundfluoreszenz außergewöhnlich hoch war und während der gesamten Reaktion kein Unterschied festgestellt werden konnte. Dies ist auf eine zu hohe Sondenkonzentration in der Reaktion oder eine fehlerhafte Sonde mit unzureichendem Quenching zurückzuführen.
Wenn der ursprüngliche Versuch auf dem Sondennachweis beruhte, sollte der Assay unter Verwendung von SYBR Green I-Reagenzien wiederholt werden, einschließlich einer Positiv- und einer Negativkontrolle (aber ohne wertvolle Proben). Alternativ dazu können die Produkte einer fehlgeschlagenen Reaktion auf einem mit Ethidiumbromid gefärbten Agarosegel überprüft werden. Die Wiederholung des Versuchs mit SYBR Green I ist vorzuziehen, da das Risiko einer Kontamination vermieden und eine Wiederholung des Versuchs möglich wird, damit der anfängliche Ausfall überprüft werden kann. Wenn der SYBR Green I-Versuch Daten liefert, ist es möglich, dass der ursprüngliche Sondenausfall entweder auf einen technischen Fehler oder einen Sondenfehler zurückzuführen ist. Um zwischen einem experimentellen Fehler oder einem Defekt der Sonde zu unterscheiden, wird das Sondenexperiment wiederholt; wenn die Reaktion erneut ausfällt, wird die Sonde ausgetauscht. Mit diesem Ansatz können Reaktionen untersucht werden, die schlechte Daten liefern. In dem in Abbildung 11.8 dargestellten Beispiel war die Sondenreaktion suboptimal, und im Vergleich mit der Reaktion, die mit SYBR Green I durchgeführt wurde, ist zu erkennen, dass das Sondensignal nicht dem Versuch entspricht. In diesen Fällen ist es zu empfehlen, die Planung des Assays zu überprüfen und eine neue Sonde zu testen.

Abbildung 11.8.Es wurden identische Reaktionen durchgeführt, die entweder eine qPCR-Sonde oder den Farbstoff SYBR Green I (wie angegeben) enthielten. Die SYBR Green I-Reaktion war etwa elf Zyklen empfindlicher und ergab eine viel höhere Fluoreszenz des Endpunkts. Dies deutet auf einen Fehler in der Sonde oder ein Problem mit der Sondenkonstruktion hin (Daten wurden freundlicherweise von Prof. Stephen Bustin, UK, zur Verfügung gestellt).
Validierung der Sondenmarkierung
Die Rohdaten oder das Mehrkomponenten-Diagramm sind ein nützliches Diagnoseinstrument, um zu untersuchen, ob die geeignete Konzentration der Sonde in die Reaktion integriert wurde und ob die Sonde angemessen markiert und gequencht ist. In Abbildung 11.9 wird ein Mehrkomponenten-Diagramm für eine Reaktion mit drei Sonden dargestellt. Die ersten beiden erzeugen Amplifikationskurven und die Hintergrundfluoreszenz ist offensichtlich. Von der dritten Sonde liegen keine Daten vor, und eine Untersuchung der Rohdaten zeigt, dass die Hintergrundfluoreszenz der Leerwertkontrolle mit Wasser entspricht, die keine Sonde enthält. Daher sind diese Daten auf das Fehlen von Fluoreszenz in der Reaktion zurückzuführen. Dies könnte auf einen Fehler bei der Einrichtung zurückzuführen sein, bei der die Sonde nicht einbezogen wurde. Oder darauf, dass die Sonde nicht markiert war.

Abbildung 11.9.Drei Gene wurden in der gleichen Template-Probe nachgewiesen. Zwei Reaktionen führten zu einer Amplifikation (1 und 2), die dritte war jedoch negativ. Eine Untersuchung der Mehrkomponenten-Ansicht macht deutlich, dass die Hintergrundfluoreszenz für die 3. Reaktion der der Wasserkontrolle entspricht, was auf ein fehlendes Signal hinweist.
Eine weitere Überprüfung der Sondenmarkierung kann mit einem DNase I-Verdau durchgeführt werden. Dies muss mit äußerster Sorgfalt geschehen, um sicherzustellen, dass die Sonden- und Primer-Stammlösungen nicht mit Enzymen verunreinigt werden, was zu katastrophalen Ergebnissen führen würde. Ein Aliquot einer fehlerhaften Sonde (Abbildung 11.10A), das der in einer Reaktion enthaltenen Sonde entspricht, z. B. 300 nM, wird mit und ohne DNase I inkubiert. Dies kann in Echtzeit erfolgen (Abbildung 11.10B), sodass die Fluoreszenzausbeute in Abhängigkeit von der Zeit gemessen wird, oder alternativ liefern der Anfangs- und Endpunkt (nach 10 min) ausreichende Informationen. Bei der Durchführung dieses Tests ist es wichtig, die Daten mit einer gut funktionierenden Sonde zu vergleichen, welche die gleiche Fluoreszenzmarkierung und den gleichen Quencher aufweist (Abbildung 11.10B).

Abbildung 11.10. A)Zwei Templates wurden mit verschiedenen Sonden nachgewiesen, die beide FAM-markiert waren. Während der Nachweis mit einer Sonde zu einem hohen Fluoreszenzsignal führte, war das zweite Signal viel schwächer. B) Eine Kontrolle und eine Testsonde (300 nM) wurden bei 37 °C in einem Echtzeitgerät in DNase-I-Puffer in Gegenwart oder Abwesenheit von DNase-I-Enzym inkubiert. Die Fluoreszenzfreisetzung von Sonde 1 war etwa doppelt so hoch wie die von Sonde 2, wodurch deutlich wird, dass die Markierung von Sonde 2 unzureichend war.
| Problemstellung | Geringe oder fehlende Fluoreszenz sowohl in der Testprobe als auch in der Positivkontrolle. Das richtige PCR-Produkt ist auf dem Gel sichtbar und das Design ist überprüft | ||
|---|---|---|---|
| SYBR Green Farbstoff-basierter Nachweis | Sondennachweis | ||
| Mögliche Ursache | Schlecht bindender SYBR Green I-Farbstoff. |
|
|
| Diagnostischer Test |
|
|
|
| Lösungen | Kaufen Sie einen neuen bindenden SYBR Green I-Farbstoff oder einen neuen qPCR-Mix mit dem bindenden Farbstoff SYBR Green |
| Kaufen Sie eine neue Sonde. |
Amplifikationskurven
Die Struktur der Amplifikationskurven und die Reproduzierbarkeit der technischen Replikate liefern eine Fülle von Informationen über die Qualität des qPCR-Assays und können auch erste Warnhinweise darauf geben, dass nicht alles so ist, wie es sein sollte. Die Amplifikationskurven in Abbildung 11.11A sind untypisch, stark verrauscht und eine genaue Auswertung wäre schwierig. Eine weitere Untersuchung der dR-Fluoreszenzwerte zeigt, dass die Fluoreszenzausbeute am Endpunkt nur 400 Einheiten beträgt, was darauf hindeutet, dass die Reaktion unzureichend ist, aber die Amplifikationskurven von der Gerätesoftware erstellt und automatisch skaliert wurden. In ähnlicher Weise zeigen die Daten in Abbildung 11.11B zu Beginn des Profils einen ausgeprägten Fuchsschwanz (abnehmende Kurve) auf, bevor sie nach einem Grundlinienabschnitt wieder ansteigen. Das Erscheinungsbild des Fuchsschwanzes ist bei zwei Reaktionen gleich, aber eine Reaktion hat einen viel niedrigeren Endpunkt (Abbildung 11.11C), was zu einem verstärkten, relativen Fuchsschwanz führt.
Abbildung 11.11. A) Amplifikationskurven, die aufgrund der automatischen Skalierung von schlechten Daten mit geringer Fluoreszenz durch die Gerätesoftware verrauscht sind. B) Reaktionen, die einen niedrigen Endpunkt dR-Wert ergeben, haben einen ausgeprägten Anfangsfuchsschwanz. C) Der Fuchsschwanz wird als normaler Effekt angesehen, wenn er im Verhältnis zum hochwertigen Assay steht.
In ähnlicher Weise sind die Amplifikationskurven in Abbildung 11.12A eindeutig abnormal und können in der vorliegenden Form nicht verwendet werden. Eine Amplifikationskurve, die unter Null dR einbricht (Abbildung 11.12A), ist ein klassischer Hinweis darauf, dass ungeeignete Grundlinieneinstellungen vorgenommen wurden. Die Untersuchung der Rohdaten für diese Reaktion (Abbildung 11.12B) zeigt, dass die tatsächlichen Amplifikationskurven ein normales Profil aufweisen, wodurch bestätigt wird, dass die analysierten Daten das Ergebnis eines Problems der Gerätesoftware sind. Die geeignete Grundlinie kann aus den Rohdaten abgeleitet und in der Software angewendet werden. In diesem Fall stellen die Zyklen 6 bis 16 die anfängliche lineare Grundlinienphase der Reaktion dar und führen bei Anwendung zu normalen Amplifikationskurven (Abbildung 11.12C).

Abbildung 11.12. A)Die Amplifikationskurven waren eindeutig abnormal, wobei ein Abschnitt des Profils unter die Grundlinie abfiel. B) Die Untersuchung der Rohdaten zeigt, dass die Reaktionsdaten den Erwartungen entsprechen. C) Durch Einstellung der Grundlinie des Geräts in Übereinstimmung mit den entsprechenden Zyklen wird das normale Profil der Daten der analysierten Amplifikationskurven wieder hergestellt.
Das Profil der Amplifikationskurve kann auch ausgewertet werden, um Informationen über die Qualität des Assays und die Optimierung zu erhalten. In Abbildung 11.13 wird die versuchte Amplifikation einer 10-fachen seriellen Verdünnung des Templates dargestellt, wobei jede Konzentration zweifach in qPCR durchgeführt wurde. Die Reproduzierbarkeit zwischen den Replikaten ist schlecht, die Zyklusdifferenz (ΔCq) zwischen den Daten ist nicht konstant und liegt nicht bei 3,323 Zyklen, wie für eine 10-fache serielle Verdünnung erwartet. Die Untersuchung der Amplifikationskurven unter Berücksichtigung, dass es sich um eine Standardkurve handelt, deutet darauf hin, dass der Assay unter dem Standard liegt und nicht für die Analyse verwendet werden kann. Die Gründe dafür müssen weiter untersucht werden, könnten aber das Ergebnis einer schlechten Assay-Planung (siehe Assay-Planung für PCR/qPCR/dPCR), suboptimaler Assay-Bedingungen (siehe Assay-Optimierung und -Validierung) oder einer schlechten Pipettierung (Wiederholung des Assays) sein.

Abbildung 11.13.Eine cDNA-Probe wurde durch eine 10-fache serielle Verdünnung verdünnt und das spezifische Template durch zweifache qPCR für jede Verdünnung nachgewiesen. Die Replikate sind von schlechter Qualität, was auf ein Problem beim Pipettieren oder bei der Assay-Optimierung hinweist.
| Problemstellung | Die Fluoreszenzkurven steigen plötzlich an (Abbildung 11.14). |
|---|---|
| Mögliche Ursachen | Schlechter Referenzfarbstoff oder Gerätefehler (z. B. Tür wurde während des Durchlaufs geöffnet). |
| Diagnostischer Test |
|
| Lösungen |
|

Abbildung 11.14.Bei einer Standard-qPCR steigen die Daten plötzlich mit einem untypischen Profilverlauf an.
| Problemstellung | Abnehmende oder hakenförmige Fluoreszenzkurven (Abbildung 11.15) |
|---|---|
| Mögliche Ursachen | Während sich das PCR-Produkt ansammelt, konkurriert der komplementäre Strang mit dem Primer und/oder der Sonde um die Hybridisierung am Template. |
| Diagnostischer Test | Siehe Abbildung 11.15 |
| Lösungen | Ignorieren, wenn der Cq-Wert nicht betroffen ist. |

Abbildung 11.15.Bei abnehmenden oder hakenförmigen Fluoreszenzkurven kann die mögliche Ursache darin liegen, dass der komplementäre Strang mit dem Primer und/oder der Sonde um die Hybridisierung am Template konkurriert. Ignorieren, solange Ct nicht betroffen ist
Dissoziations- / Schmelzkurven
Die Dissoziations- oder Schmelzkurvenanalyse wird nach der qPCR durchgeführt und ist ein Analysewerkzeug, das in Verbindung mit DNA-bindenden Farbstoffen (wie SYBR Green I) oder nicht degradierenden Sonden wie Molecular Beacons oder Scorpions® Sonden verwendet wird, um zu verifizieren, dass ein einzelnes Produkt amplifiziert wurde. Nach der PCR-Amplifikation wird das resultierende Amplikon bei steigenden Temperaturen inkubiert und die Veränderungen des Fluoreszenzsignals werden beim Übergang der DNA zwischen doppelsträngigen und einzelsträngigen Zuständen nachgewiesen. Wenn die Reaktion ein einzelnes Amplikon enthält, schmilzt dieses gleichmäßig und die Kurve von dF/dT (Änderungsrate der Fluoreszenz in Bezug auf die Temperatur) weist einen einzelnen Peak auf. Die Untersuchung der Schmelzkurve ist besonders effektiv, wenn sie mit Daten von Kontrollen kombiniert wird. Abbildung 11.16A zeigt das Post-qPCR-Schmelzprofil für eine Reihe von experimentellen Testproben, die Positivkontrolle und die Negativkontrolle (NCT). Das Schmelzprofil der Testproben ist identisch mit dem der Positivkontrolle und zeigt jeweils einen einzelnen Peak für dF/dT auf. Das Schmelzprofil für die Negativkontrolle hat ein breiteres Profil und einen niedrigeren Tm-Wert. Diese Beobachtungen stimmen mit dem Vorhandensein von Primer-Dimeren in der Negativkontrolle überein. Dies wird durch ein mit Ethidiumbromid gefärbtes Agarosegel bestätigt (Abbildung 11.16B), das auch zeigt, dass die Primer-Dimere sichtbar werden, wenn das Template in geringer Konzentration vorhanden ist. Dies führt zu einer Überschätzung des Ziels, wenn es in Proben mit niedriger Zielkonzentration nachgewiesen wird. Daher ist zu empfehlen, den Assay zu optimieren oder neu zu planen. Im Gegensatz dazu zeigt Abbildung 11.16C, dass das Schmelzprofil für das Produkt in der Negativkontrolle identisch mit dem Schmelzprofil für die Positivkontrolle und die Testprobe ist. Dies ist ein klarer Hinweis auf eine Kontamination der Negativkontrolle mit Template während des Versuchsaufbaus. Das letzte Beispiel zeigt die Erkennung der Amplifikation des Ziels aus gDNA, die in einer cDNA-Probe vorhanden ist (Abbildung 11.16D). Das aus gDNA gewonnene Amplikon ist länger und verfügt daher über einen höheren Tm-Wert als das aus cDNA.

Abbildung 11.16A.Eine Positivkontrolle, eine Testreaktion und ein NTC wurden amplifiziert und anschließend einer Post-PCR-Schmelzanalyse unterzogen. In der NTC ist ein Produkt erkennbar, das bei einer niedrigeren Temperatur und mit einem breiteren Schmelzpeak schmilzt, was mit der Bildung von Primer-Dimeren übereinstimmt.

Abbildung 11.16B.Primer-Dimere sind bei einer Gelauflösung dieser Proben (zusammen mit anderen) zu erkennen, wobei die Bildung von Primer-Dimeren umgekehrt proportional zur Konzentration der Eingabe-Template ist.

Abbildung 11.16C.Beispiel für die Anwendung der Schmelzkurvenanalyse zur Ermittlung von Reaktionsverunreinigungen in der NTC.
Abbildung 11.16D.Identifizierung eines größeren Amplikons, das durch PCR von gDNA entstanden ist.
| Problemstellung | Mehrere Tm-Peaks in Schmelzprofildaten | |
|---|---|---|
| Einzelprodukt auf einem Gel (Abbildung 11.17) | Mehrere Produkte auf einem Gel | |
| Mögliche Ursachen | Lokalisierte AT- oder GC-reiche Regionen oder kurze Wiederholungen im PCR-Produkt, die eine unvollständige erneute Hybridisierung des Amplikons mit offensichtlich unterschiedlichen Schmelzprofilen verursachen. | Die Primer sind unspezifisch und erzeugen mehrere Produkte.
|
| Diagnostischer Test | Überprüfen Sie die Sequenz des Amplikons auf AT- oder GC-reiche Regionen oder Wiederholungen. | Vergleichen Sie BLAST-Primersequenzen mit der Sequenz des Ausgangsorganismus, um das Einzelziel zu überprüfen. |
| Lösungen | Dies stellt kein Problem dar, wenn die Gelanalyse zeigt, dass das gesamte Produkt spezifisch ist. Verwenden Sie weiterhin die Primer. |
|

Abbildung 11.17. A)A) Schmelzprofil und B) Agarosegelanalyse einer SYBR Green I-Reaktion. Obwohl das Schmelzprofil Produkte mit unterschiedlichem Tm-Wert vermuten lässt, zeigt das Gelbild auf, dass ein einzelnes Amplikon vorhanden ist. Dies deutet auf eine Amplikonsequenz hin, die AT- oder GC-reiche Regionen oder ein sich wiederholendes Element enthält, was zu unregelmäßigem Schmelzen führt.
Serielle Verdünnung der Template/Standardkurven
Unabhängig davon, ob die Versuchsplanung eine Standardkurve für eine mögliche Quantifizierung vorsieht, ist die Ermittlung einer seriellen Verdünnung eines geeigneten Templates ein leistungsfähiger Ansatz für die Assay-Validierung und die Fehlerbehebung. Die Ermittlung einer seriellen Verdünnung ermöglicht es, den experimentellen linearen dynamischen Bereich des Assays zu definieren. In Abbildung 11.18A wird eine Standardkurve mit Datenpunkten niedriger Konzentrationen dargestellt, die nicht dem linearen Profil entsprechen. Der häufigste Grund für dieses Datenmuster liegt in der Bildung von Primer-Dimeren in Proben mit niedriger Konzentration (wie in Abbildung 11.18B dargestellt). Dies ist die Standardkurve, die anhand der in Abbildung 11.2 enthaltenen Daten erstellt wurde. Abbildung 11.18C zeigt eine Standardkurve mit hochkonzentrierten Proben, die aus dem linearen Bereich herausfallen. Die häufigsten Gründe hierfür sind die Hemmung der Reaktion durch das Template oder ungeeignete Grundlinieneinstellungen.

Abbildung 11.18. A) Die Datenpunkte, die sich auf die niedrigeren Konzentrationen des Ziels beziehen, liegen nicht auf der Standardkurve. B) Dies ist typisch für eine Reaktion, bei der Primer-Dimere entstehen, wie erläutert. In diesem Fall ist kein Anstieg von Cq in den Proben mit niedriger Konzentration zu beobachten.

Abbildung 11.18C.Die Proben mit hohen Konzentrationen des Templates liegen nicht auf der Standardkurve. Dies ist typisch für Reaktionen, die durch die Template-Konzentration oder eine falsche Grundlinieneinstellung gehemmt werden.
Die Standardkurve wird auch verwendet, um die Effizienz der Reaktion über den dynamischen Bereich der Verdünnungen zu messen. Es ist darauf zu achten, dass alle für die Effizienzberechnung verwendeten Punkte auf der Linie liegen. Die Reaktionen sollen so nahe wie möglich an 100 % Effizienz liegen, und diejenigen mit offensichtlich hoher (> 110 %) oder niedriger (< 85 %) Effizienz weiter untersucht werden.

Abbildung 11.19A.Eine Template-Nukleinsäure wurde in einer 10-fachen Serie verdünnt. Die Amplifikationskurven weisen einen ungewöhnlich flachen Gradienten auf und der ΔCq-Wert beträgt eher 4 Zyklen statt der erwarteten 3,3.

Abbildung 11.19B.Der Gradient einer Standardkurve, in welcher der Cq-Wert im Vergleich zur Menge dargestellt wird, wird zur Berechnung der Effizienz der Reaktion verwendet.
| Problemstellung | Die PCR-Effizienz ist größer als 120 % (Abbildung 11.20) |
|---|---|
| Mögliche Ursachen |
|
| Diagnostischer Test |
|
| Lösungen |
|

Abbildung 11.20A.Eine Template-Nukleinsäure wurde in einer 10-fachen Serie verdünnt. Der ΔCq-Wert zwischen den Amplifikationskurven beträgt 1,5 Zyklen statt 3,3

Abbildung 11.20B.Mithilfe des Gradienten einer Standardkurve, anhand der der Cq-Wert im Vergleich zur Menge dargestellt wird, wird die Effizienz der Reaktion berechnet, die nahe bei 140 % liegt.

Abbildung 11.20C.Eine Untersuchung des Schmelzkurvenprofils zeigt, dass die Proben mit niedrigerer Konzentration (gelbe und blaue Spuren) auch Signale von amplifizierten Primer-Dimeren enthalten (Peak bei niedrigem Tm-Wert).
Fallstudien zur Fehlerbehebung in der RT-PCR
Ein fehlgeschlagener Sonden-Assay
Ein sondenbasierter Assay wurde entwickelt, um EIFB1 in menschlichen cDNA-Proben nachzuweisen, es fand jedoch keine Amplifikation statt. Die ersten Reaktionen wurden auf einem ABi StepOne-Gerät mit kompatiblen Reagenzien durchgeführt. Es wurde ein Versuch unternommen, die Primer in einem Konzentrationsbereich von 200 nM bis 900 nM zu optimieren (Abbildung 11.21), was jedoch zu keiner Verbesserung führte. Die Assay-Planung wurde überprüft und als für das Ziel geeignet befunden. In silico wurde prognostiziert, dass es sich um einen qualitativ hochwertigen Assay handelt. Neue Primer wurden synthetisiert und zusammen mit einem Aliquot der ursprünglichen Synthese unter Verwendung eines anderen Operators, SYBR Green I-Reagenzien (daher andere Reagenzien) und eines Geräts (ein Eppendorf Realplex) durchgeführt (Abbildung 11.22). Bei der Wahl dieses Ansatzes haben wir darauf geachtet, dass das primäre Ziel darin besteht, das Problem zu lösen, während das sekundäre Ziel darin bestand, den Ausfall zu erklären. Diese Reaktion führte zu einer äquivalenten Amplifikation aus beiden Primerchargen. In dieser Stufe stellte sich heraus, dass das Reaktionsproblem bei der Sonde lag. Daher wurde eine neue Sonde synthetisiert und beide Chargen wurden vom zweiten Bediener auf dem Realplex-Gerät unter Verwendung von LuminoCt® Reagenzien (andere Reagenzien als die ursprünglich verwendeten) verglichen (Abbildung 11.23). Beide Sonden ergaben Amplifikationsdaten, wobei die neue Sonde etwas besser abschnitt als die ursprüngliche Sonde. Dabei ist jedoch zu beachten, dass die ursprüngliche Sonde zwischen den Prüflaboren hin- und hergeschickt wurde und daher mehrere Tage bei Raumtemperatur in Lösung lag. Zu diesem Zeitpunkt war klar, dass sowohl der ursprüngliche als auch der Ersatz-Assay funktionierten, wenn sie vom zweiten Bediener mit LuminoCt® Reagenzien auf dem Realplex-Gerät durchgeführt wurden.
Daher waren die verbleibenden Annahmen für das ursprüngliche Versagen wie folgt:
- Bediener: Der Versuch wurde von einem erfahrenen Wissenschaftler mehrmals wiederholt, sodass diese Erklärung als unwahrscheinlich gilt.
- Gerät: Könnte einige Probleme aufweisen, da einige andere Assays fehlgeschlagen sind.
- Reagenzien: Am einfachsten zu testende Erklärung. Die LuminoCt® Reagenzien wurden vom ersten Bediener mit den bestehenden Reagenzien verglichen, wobei beide Oligo-Chargen auf dem ABi StepOne verwendet wurden. Die Reaktion schlug mit den Originalreagenzien fehl, ergab aber eine gute Amplifikation mit LuminoCt® Reagenzien (Abbildung 11.24).

Abbildung 11.21.Die Primer für EIFB1 wurden in Konzentrationen zwischen 200 nM und 900 nM getestet. Unter keiner der Bedingungen (Oligos und ABi-Reagenzien auf dem ABi StepOne Plus) wurde eine Amplifikation beobachtet.

Abbildung 11.22.Zwei Chargen Primer für EIFB1 wurden in SYBR Green I-Reagenz verglichen: die ursprüngliche, nicht funktionierende Charge und eine neue Charge. (Oligos und Reagenzien auf dem Eppendorf Realplex-Gerät). Beide Primersätze haben die Amplifikation unterstützt.

Abbildung 11.23.Zwei Chargen Primer und Sonden für EIFB1 wurden im LuminoCt® Reagenz miteinander verglichen: die ursprüngliche fehlerhafte Charge und eine neue Charge. (Oligos und Reagenzien auf dem Eppendorf Realplex-Gerät). Beide Oligosätze haben die Amplifikation unterstützt.

Abbildung 11.24.Der EIFB1-Primer- und Sonden-Assay wurde mit zwei verschiedenen Reagenzien durchgeführt (Original ABi oder LuminoCt® Reagenz). Daten wurden mit diesem Assay nur bei Verwendung von LuminoCt® Reagenzien erlangt.
Der Wirkungsgrad der Reaktion war nicht korrekt und variabel
Ein Test wurde mit einer seriellen Verdünnung eines künstlichen Oligos unter Verwendung eines Standard-Primers und Sonden-Assays durchgeführt. Der Assay wurde ursprünglich auf einem anderen Gerät entwickelt und optimiert, aber der seltsame Verdünnungseffekt wurde nicht erwartet, als er an ein anderes Prüflabor übergeben und auf anderes Gerät übertragen wurde (Abbildung 11.25A). Alle Assaybedingungen wurden für das neue Labor neu optimiert, ohne dass die Daten geändert wurden. Der Bediener stellte fest, dass der Effekt stärker war, wenn der Assay innerhalb weniger Stunden mit der gleichen Verdünnungsreihe wiederholt wurde. Im Rahmen der Fehlerbehebung wurde der Assay auf einem anderen Gerät von einem anderen Bediener durchgeführt, der wiederum die erwartete Standardkurve generierte. Dies führte zu der Vermutung, dass das anfängliche Problem auf einen Bedienerfehler, einen Geräteausfall oder eine geringfügige Abweichung im Versuchsablauf zurückzuführen war. Da beide Bediener sehr erfahren sind und das Gerät bei anderen Experimenten gut funktionierte, wurde die Option der geringfügigen Unterschiede geprüft. Ein wichtiger Hinweis war die Beobachtung der Variabilität der Daten aus derselben Verdünnungsreihe nach einer Lagerung der Proben bei 4 °C (Abbildungen 11.25A und 11.25B). Dies führte zu einer Untersuchung der für die Verdünnungsreihen verwendeten Röhrchen und zu einem Test von Alternativen. Nach dem Wechsel zu Eppendorf 1,5 ml-Reaktionsröhrchen für die Verdünnungsreihe wurde die prognostizierte Standardkurve erstellt (Abbildung 11.25C). Dies macht deutlich, dass es wichtig ist, Kunststoffgefäße für die Mikrobiologie mit geringer Rückhaltung für die PCR zu wählen, und dass diese Assays empfindlich auf geringfügige Variationen im Protokoll reagieren.

Abbildung 11.25A.Ein künstliches Oligo-Template wurde 10-fach verdünnt und mit einem Assay auf Basis einer spezifischen Sonde nachgewiesen. Es gibt inkonsistente Unterschiede zwischen den Amplifikationskurven.

Abbildung 11.25B.Ein künstliches Oligo-Template wurde 10-fach verdünnt (dies sind die Verdünnungen, die in Abbildung 11.25A nachgewiesen wurden) und mehrere Stunden bei 4 °C gelagert, bevor es mit einem auf einer spezifischen Sonde basierenden Assay nachgewiesen wurde. Es gibt inkonsistente Unterschiede zwischen den Amplifikationskurven, die durch die Zeit zwischen Verdünnung und Test deutlicher werden.

Abbildung 11.25C.Ein künstliches Oligo-Template wurde 10-fach in Röhrchen für die Molekularbiologie verdünnt und mit einem spezifischen sondenbasierten Assay nachgewiesen. Wie erwartet, gibt es konsistente Unterschiede zwischen den Amplifikationskurven.
Zusammenfassung - PCR-Checkliste für die Fehlerbehebung
- Prüfen Sie die Qualität der Probe (minderwertiges Material führt zu fehlerhaften Ergebnissen).
- Prüfen Sie, ob das RT-Protokoll mit der Planung kompatibel ist (z. B. muss eine Oligo-dT-Primer-RT einen qPCR-Assay in den 3' 1 kb der Sequenz aufweisen).
- Überprüfen Sie die Assay-Planung.
- Prüfen Sie alle Kontrollen.
- Prüfen Sie die Primer mit dem SYBR Green I-Farbstoff / Lassen Sie ein Gel durchlaufen.
- Achten Sie auf korrekte Softwareeinstellungen (Grundlinie, Farbstoffnachweis, Konzentrationen für Standards).
- Vergewissern Sie sich, dass die ROX-Konzentration für das Gerät geeignet ist (und nicht mit dem Multiplex interferiert).
- Prüfen Sie den Grad der Hintergrundfluoreszenz.
- Überprüfen Sie die Sondenmarkierung mit einem DNase-I-Assay oder wiederholen Sie die Sondensynthese.
Um weiterzulesen, melden Sie sich bitte an oder erstellen ein Konto.
Sie haben kein Konto?