Niezawodna ekstrakcja DNA z kart Whatman® FTA®
W niniejszym badaniu zbadano wydajność i jakość DNA z próbek naniesionych na karty Whatman® FTA® przy użyciu pięciu popularnych metod ekstrakcji DNA. Dane pokazują, że wszystkie pięć metod daje oczyszczone DNA o wystarczającej ilości i jakości zarówno do ilościowego PCR, jak i analiz krótkich powtórzeń tandemowych. Ponadto wykazano, że urządzenie EasiCollect™ do pobierania próbek z policzka jest kompatybilne ze wszystkimi pięcioma metodami ekstrakcji DNA i zapewnia końcową wydajność DNA równą lub wyższą niż w przypadku oddzielnych wymazów z pianki.
Wprowadzenie
Rodzina produktów Whatman® FTA® (Rysunek 1) ułatwia pobieranie, transport, oczyszczanie i długoterminowe przechowywanie kwasów nukleinowych w temperaturze pokojowej w jednym urządzeniu. Technologia FTA ma zdolność do lizy komórek w kontakcie, denaturacji białek, usuwania zanieczyszczeń i ochrony DNA przed degradacją. W klasycznym scenariuszu dla kart FTA, oczyszczone DNA jest analizowane, gdy jest jeszcze związane z matrycą (tj. przy użyciu metody "punch-in"). W tym badaniu zbadano alternatywną opcję dla kart FTA - ekstrakcję DNA do roztworu.
Materiały i metody
Pobieranie próbek
Krew pełną i komórki policzkowe zebrano na kartach FTA.
Krew
Krew od dziesięciu niespokrewnionych osób została pobrana przez nakłucie żyły do próżniowych probówek zawierających dipotasowy EDTA. Krew (40 μl) została naniesiona na każdą kartę FTA Micro Card. Karty suszono przez co najmniej 3 godziny w temperaturze pokojowej.

Rysunek 1. Rodzina produktów Whatman® FTA®.
Próbki jamy ustnej
Próbki jamy ustnej pobrano od dziesięciu niespokrewnionych osób przy użyciu urządzeń EasiCollect firmy Cytiva (Rysunek 2) lub wacików piankowych. Próbki naniesiono na karty FTA zgodnie z instrukcjami producenta. Karty suszono przez co najmniej 2 godziny w temperaturze pokojowej.

Rysunek 2. EasiCollect™ to urządzenie typu "wszystko w jednym", które pobiera próbki z policzka i przenosi je na zintegrowaną kartę FTA.
Metody ekstrakcji DNA
DNA zostało wyekstrahowane lub wyizolowane z kart FTA przy użyciu pięciu różnych metod, dokładnie tak, jak opisano w poniższych protokołach. Metody te obejmują:
- Ekstrakcja organiczna
- Żywica Chelex 100 (142-1253, Bio-Rad Laboratories,vHercules, CA, USA)
- QIAamp™ DNA Investigator Kit (56504, Qiagen, Valencia, CA, USA)
- illustra™ tissue and cells genomicPrep Mini Spin Kit (Cytiva)
- DNA IQ™ Kit (TB297, Promega Corp, Madison, WI, USA)
Organiczna ekstrakcja DNA
Przygotować bufor ekstrakcyjny i roztwór podstawowy Proteinazy K. Bufor ekstrakcyjny: 10 mM Tris-HCl, pH 8,0; 10 mM EDTA, sól disodowa, pH 8,0; 100 mM chlorek sodu; i 2% v/v SDS. Roztwór podstawowy proteinazy K: 20 mg/ml w sterylnej wodzie destylowanej.
- Użyj jednorazowego stempla Harris Uni-Core (1,2, 2,0, 3,0 lub 6,0 mm), aby usunąć krążek ze środka wysuszonej plamki próbki. Umieść krążek w czystej, wolnej od RNazy/DNazy probówce do mikrowirówki o pojemności 1,5 ml.
- Dodaj 500 μL buforu ekstrakcyjnego i 20 μL podstawowego roztworu proteinazy K.
- Worteksować i inkubować w temperaturze 56°C przez noc z mieszaniem.
- Dodać taką samą objętość zbuforowanego fenolu (pH 8,0), krótko worteksować i wirować przez 10 minut z maksymalną prędkością w mikrowirówce.
- Przenieść górną, wodną fazę do nowej probówki mikrowirówki zawierającej 500 μL chloroformu.
- Dokładnie wymieszać i wirować przez 10 minut z maksymalną prędkością w mikrowirówce.
- Przenieść górną, wodną fazę do nowej probówki zawierającej 50 μL 3 M octanu sodu, pH 5.2.
- Dodać 800 μL 100% etanolu, wymieszać i wytrącać w temperaturze -20 °C przez co najmniej 1.5 godzin.
- Odzyskaj DNA przez wirowanie przez 30 minut z maksymalną prędkością w mikrowirówce i zdekantuj supernatant.
- Dodaj 1 ml 70% etanolu do osadu i wiruj przez 20 minut z maksymalną prędkością w mikrowirówce.
- Osusz osad w wirówce próżniowej przez 30 minut.
- Rozpuść osad w 50 μL buforu TE lub wody.
Izolacja DNA przy użyciu żywicy Chelex 100
Przygotuj 5% w/v zawiesinę żywicy Chelex 100 w sterylnej wodzie przed użyciem; mieszaj, aby utrzymać zawiesinę.
- Użyj jednorazowego stempla Harris Uni-Core (1,2, 2,0, 3,0 lub 6,0 mm), aby usunąć krążek próbki ze środka wysuszonej plamki próbki. Umieść krążek w czystej, wolnej od RNase/DNase probówce do mikrowirówki o pojemności 1,5 ml.
- Maceruj krążek FTA za pomocą igły o rozmiarze 20.
- Umyj krążek, dodając 1 ml sterylnej wody do każdej probówki z próbką i inkubując w temperaturze pokojowej przez 10 minut z okazjonalnym worteksowaniem. Usunąć wodę.
- Powtórzyć płukanie, dodając 1 ml sterylnej wody do każdej próbówki i inkubując w temperaturze pokojowej przez 10 minut z okazjonalnym worteksowaniem.
- Wirować próbki przez 3 minuty przy 20 000 x g.
- Usuń i wyrzuć supernatant.
- Dodaj 200 μL zawiesiny Chelex 100 za pomocą końcówki pipety o dużym otworze.
- Inkubuj próbkę w temperaturze 56 °C przez 20 min.
- Worteksować przez około 15 sekund.
- Inkubować w temperaturze 100 °C przez 8 minut.
- Worteksować przez około 15 sekund.
- Wirować próbkę przez 3 minuty z prędkością 20 000 x g.
- Przenieść supernatant do nowej sterylnej, wstępnie oznakowanej probówki do mikrowirówki, uważając, aby nie przenieść osadu żywicy Chelex 100.
- Przechowuj DNA w temperaturze 2 °C -8 °C przez krótki czas lub w temperaturze -20 °C przez dłuższy czas.
Izolacja DNA przy użyciu zestawu QIAamp DNA Investigator Kit
Odczynniki i kolumny są dostarczane z zestawem.
- Użyj jednorazowego stempla Harris Uni-Core (1,2, 2,0, 3,0 lub 6,0 mm), aby usunąć krążek próbki ze środka wysuszonej plamki próbki. Umieść krążek w czystej, wolnej od RNazy/DNazy probówce do mikrowirówki o pojemności 1,5 ml.
- Dodaj 280 μL buforu ATL do probówki.
- Dodaj 20 μL Proteinazy K i worteksuj przez 30 sekund.
- Ogrzewaj przez 60 minut w temperaturze 56°C, worteksując przez 30 sekund co 10 minut.
- Wirować z maksymalną prędkością w mikrowirówce przez 30 sekund.
- Dodać 300 μL buforu AL i worteksować przez 10 sekund.
- Inkubować w temperaturze 70°C przez 10 minut, worteksując przez 10 sekund co 3 minuty.
- Wirować z maksymalną prędkością przez 30 sekund i przenieść lizat na kolumnę minElute.
- Wirować z prędkością 6000 x g przez 1 min i odrzucić przesącz.
- Dodać 700 μL buforu AW2 i wirować przez 1 min z prędkością 6000 x g. Odrzucić przesącz.
- Dodać 700 μL 100% etanolu i wirować przez 1 min z prędkością 6000 x g. Odrzucić przesącz.
- Dodać 700 μL 100% etanolu i wirować przez 1 min z prędkością 6000 x g. Odrzucić przepływ.
- Wirować z prędkością 20 000 x g przez 3 minuty, a następnie otworzyć pokrywę kolumny na 10 minut w temperaturze pokojowej.
- Dodać 50 μL wody destylowanej i inkubować przez 5 minut w temperaturze pokojowej.
- Wirować z prędkością 20 000 x g przez 1 minutę. Przechowuj przepływ, który zawiera wyekstrahowane DNA, w temperaturze 4 °C, aż będzie potrzebny.
Izolacja DNA przy użyciu zestawu illustra tissue and cells genomicPrep Mini Spin Kit
Odczynniki i kolumny są dostarczane wraz z zestawem.
- Użyj jednorazowego dziurkacza Harris Uni-Core (1,2, 2,0, 3,0 lub 6,0 mm), aby usunąć krążek próbki ze środka wysuszonej plamki próbki. Umieść krążek w czystej, wolnej od RNase/DNase probówce do mikrowirówki o pojemności 1,5 ml.
- Dodaj 1 ml PBS i odwiruj przy 16 000 x g.
- Maceruj krążek za pomocą igły strzykawkowej o rozmiarze 20.
- Wiruj przez 10 sekund przy 2000 x g.
- Dodać 50 μL buforu 1 do każdej próbki.
- Dodać 10 μL proteinazy K do każdej próbki i worteksować przez 15 sekund.
- Inkubować przez 1 godzinę w temperaturze 56°C
- Wirować przez 10 sekund z prędkością 2000 x g. Dodać 5 μL RNazy A (20 mg/ml). Inkubować przez 15 minut w temperaturze pokojowej.
- Dodać 500 μL buforu 4 i worteksować przez 15 sekund.
- Inkubować przez 10 minut w temperaturze pokojowej.
- Nanieść pipetą próbkę do minikolumny (umieszczonej w probówce zbierającej) i wirować przez 1 min z prędkością 11 000 x g. Odrzucić przepływ.
- Dodać 500 μL buforu 4 do każdej kolumny. Wirować przez 1 minutę z prędkością 11 000 x g i odrzucić przepływ.
- Umieścić kolumnę w probówce zbierającej i dodać 500 μL buforu 6 do kolumny.
- Wirować przez 3 minuty z prędkością 11 000 x g. Przenieść kolumnę do nowej probówki o pojemności 1,5 ml.
- Dodać do kolumny 50 μL wstępnie ogrzanego buforu elucyjnego (bufor 6).
- Inkubować przez 1 minutę w temperaturze pokojowej.
- Wirować przez 1 minutę z prędkością 11 000 x g. Wyrzucić kolumnę i przechowywać DNA w temperaturze 4°C do czasu, aż będzie potrzebne.
Izolacja DNA przy użyciu zestawu DNA IQ
Odczynniki i kolumny są dostarczane wraz z zestawem.
- Użyj jednorazowego dziurkacza Harris Uni-Core (1.2, 2.0, 3.0 lub 6.0 mm), aby usunąć krążek próbki ze środka wysuszonej plamki próbki. Umieść krążek w czystej, wolnej od RNazy/DNazy probówce do mikrowirówki o pojemności 1,5 ml.
- Dodaj 100 μL buforu do lizy i inkubuj w temperaturze 70°C przez 30 minut.
- Przenieś zawartość do kolumny wirówkowej umieszczonej w probówce o pojemności 1,5 ml i wiruj z maksymalną prędkością przez 2 minuty. Wyrzucić kolumnę wirówkową.
- Worteksować żywicę wyjściową aż do dokładnego wymieszania i dodać 7 μL żywicy dla dysku 1,2 mm, 7 μL dla dysku 2,0 mm, 16 μL dla dysku 3,0 mm lub 28 μL dla dysku 6,0 mm.
- Worteksuj probówki przez 3 sekundy i inkubuj w temperaturze pokojowej przez 5 minut, worteksując przez 3 sekundy co minutę.
- Worteksuj probówki przez 2 sekundy i umieść na statywie magnetycznym.
- Odcedź supernatant bez naruszania osadu.
- Dodaj 100 μL buforu do lizy. Zdjąć probówkę ze statywu magnetycznego i worteksować przez 2 sekundy.
- Powrócić do statywu magnetycznego i zdekantować bufor do lizy.
- Dodać 100 μL 1× buforu do płukania. Zdejmij probówkę ze statywu i worteksuj przez 2 sekundy.
- Przywróć probówkę do statywu magnetycznego i zdekantuj bufor płuczący.
- Powtórz kroki płukania 10 i 11 jeszcze dwa razy, w sumie trzy płukania. Po ostatnim płukaniu zdekantować bufor płuczący.
- Z probówką w statywie magnetycznym otworzyć pokrywę i suszyć na powietrzu przez 10 minut.
- Dodać 100 μL buforu elucyjnego. Zamknąć pokrywkę i worteksować przez 2 minuty. Inkubować przez 5 minut w temperaturze 65°C.
- Wyjmij probówkę z ognia i worteksuj przez 2 sekundy, a następnie natychmiast umieść probówkę w statywie magnetycznym.
- Przenieś eluat do świeżej probówki i przechowuj eluowane DNA w temperaturze 4 °C do czasu, aż będzie potrzebne.
Analizy DNA
DNA wyekstrahowane z kart FTA zostało zmierzone przy użyciu ilościowego PCR (qPCR). DNA poddano analizie krótkich powtórzeń tandemowych (STR) w celu oceny jego jakości.
Ilość DNA za pomocą qPCR
Wyekstrahowane DNA mierzono za pomocą ilościowego PCR (qPCR) i systemu ABI™ 7900HT Fast Real-Time PCR (Applied Biosystems, Foster City, CA, USA). Reakcje przygotowano przy użyciu zestawu Quantifiler™ Human DNA Quantification Kit (4343895, Applied Biosystems). Test kwantyfikacji DNA łączy dwa testy nukleazy 5'. Pierwszym z nich jest specyficzny dla celu test ludzkiego DNA składający się z dwóch starterów do amplifikacji ludzkiego DNA i jednej sondy TaqMan™ MGB znakowanej barwnikiem FAM™ do wykrywania amplifikowanej sekwencji. Drugi test to test wewnętrznej kontroli PCR (IPC) składający się z dwóch starterów do amplifikacji matrycowego DNA IPC i jednej sondy TaqMan MGB znakowanej barwnikiem VIC™ do wykrywania amplifikowanego DNA IPC. Krzywe wzorcowe do oznaczania ilościowego DNA przygotowano przy użyciu kontrolnego DNA dostarczonego z zestawem.
Analiza krótkich powtórzeń tandemowych
Analizę STR wyekstrahowanego DNA przeprowadzono przy użyciu systemu Powerplex™ 16 (Promega) zgodnie z instrukcjami producenta. Amplifikację przeprowadzono w termocyklerze 9700 (Applied Biosystems), a produkty amplifikacji analizowano na analizatorze genetycznym ABI PRISM™ 3130XL (Applied Biosystems) zgodnie z instrukcjami producenta.
Wyniki i dyskusja
Ilość DNA metodą qPCR
DNA ekstrahowano z krążków FTA zawierających krew lub komórki policzkowe od dziesięciu niespokrewnionych osób. Dla każdej metody ekstrakcji użyto czterech rozmiarów krążków, a całkowitą ilość wyekstrahowanego DNA na krążek określono zgodnie z opisem w Materiałach i Metodach.
Dla każdej z pięciu metod ekstrakcji odzyskano co najmniej 10 ng genomowego DNA z próbek krwi na kartach FTA (Rysunek 3). Ilość DNA wyekstrahowanego przy użyciu każdej metody wzrastała wraz ze wzrostem rozmiaru dysku.
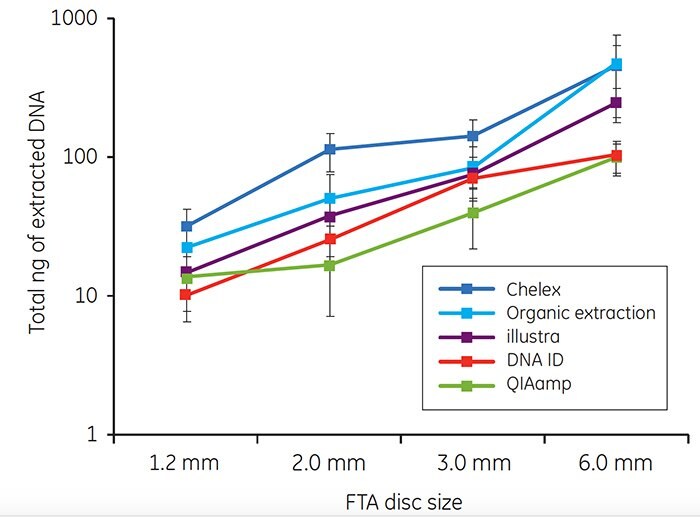
Rysunek 3. Całkowite DNA (ng) wyizolowane z krążków pobranych ze środka kart FTA nakrapianych 40 μL pełnej krwi ludzkiej. Każdy punkt na wykresie reprezentuje średni odzysk DNA z pojedynczego krążka od dziesięciu osób. Słupki błędów reprezentują odchylenia standardowe. Należy zauważyć, że oś y reprezentuje skalę logarytmiczną.
Próbki policzkowe historycznie dawały większą zmienność w wydajności DNA niż próbki krwi. Zmienność w wydajności policzkowego DNA jest najprawdopodobniej spowodowana zlepianiem się komórek na matrycy. Podobnie jak w przypadku próbek krwi, ilość DNA odzyskanego za pomocą każdej metody ekstrakcji miała tendencję do zwiększania się wraz ze wzrostem rozmiaru dysku. Należy zauważyć, że wyższą wydajność DNA często uzyskiwano z próbek policzkowych pobranych za pomocą urządzeń EasiCollect niż z próbek pobranych za pomocą wymazówek piankowych (Rysunek 4). Dane przedstawione na Ryc. 3 i Ryc. 4 ilustrują, że powszechnie stosowane metody i zestawy do ekstrakcji DNA dostarczają DNA o wystarczającej ilości i jakości do udanego qPCR.
DNA wyekstrahowane z pobranych próbek policzkowych

Rysunek 4. Całkowite DNA (ng) wyizolowane z krążków pobranych ze środka karty FTA zawierającej komórki policzkowe, nałożone za pomocą aplikatora piankowego lub urządzenia EasiCollect, jak wskazano,


Rysunek 5. A-O. Profile STR DNA wyekstrahowanego z kart FTA zawierających próbki krwi lub policzka od uczestnika testu 1.
STR w celu określenia dokładności wywołań alleli dla DNA ekstrahowanego z kart FTA. Dla każdej metody ekstrakcji DNA wykonano duplikaty DNA z kart FTA dla trzech próbek krwi, trzech próbek policzkowych pobranych za pomocą urządzeń EasiCollect i trzech próbek policzkowych pobranych za pomocą oddzielnych wymazówek piankowych, co daje łącznie 18 próbek na metodę ekstrakcji DNA. Należy zauważyć, że trzy próbki pochodziły od trzech badanych osób. Spośród 90 zebranych profili STR, 87 próbek dostarczyło wysokiej jakości danych z dokładnymi wywołaniami alleli przy pierwszej próbie; trzy próbki wymagały ponownego wstrzyknięcia w celu uzyskania 100% dokładnych wywołań alleli. Dane te ilustrują, że powszechnie stosowane metody i zestawy do ekstrakcji DNA zapewniają DNA o wystarczającej ilości i jakości, aby zapewnić dokładność wywoływania alleli na poziomie 100% w analizie STR.
Wnioski
Niniejsze badanie pokazuje, że pięć popularnych metod ekstrakcji DNA dało wystarczającą ilość DNA z kart FTA do dalszej analizy ilościowej. DNA to było wystarczającej jakości do określenia tożsamości człowieka za pomocą profilowania STR. Ponadto pobranie komórek policzkowych za pomocą urządzenia EasiCollect często skutkowało wyższą wydajnością DNA niż pobranie za pomocą wacika piankowego. Dane te pokazują, że urządzenie EasiCollect jest kompatybilne z aktualnymi zatwierdzonymi protokołami laboratoryjnymi do ekstrakcji DNA.
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?