C0383
3-Chlorobenzoic acid
About This Item
Prodotti consigliati
Punto di fusione
153-157 °C (lit.)
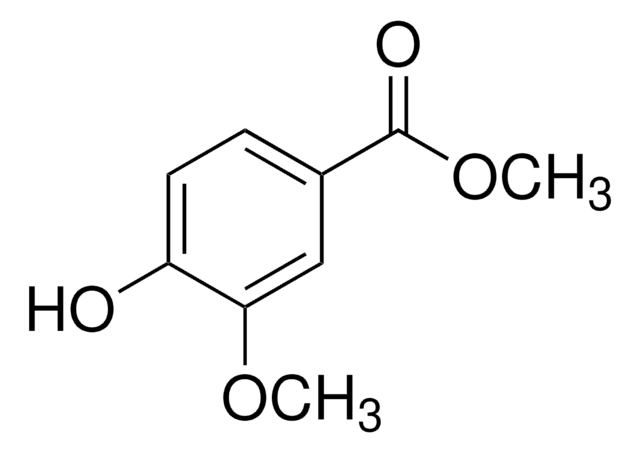
Stringa SMILE
OC(=O)c1cccc(Cl)c1
InChI
1S/C7H5ClO2/c8-6-3-1-2-5(4-6)7(9)10/h1-4H,(H,9,10)
LULAYUGMBFYYEX-UHFFFAOYSA-N
Cerchi prodotti simili? Visita Guida al confronto tra prodotti
Sostituito da
Avvertenze
Warning
Indicazioni di pericolo
Consigli di prudenza
Classi di pericolo
Eye Irrit. 2 - Skin Irrit. 2
Codice della classe di stoccaggio
11 - Combustible Solids
Classe di pericolosità dell'acqua (WGK)
WGK 3
Punto d’infiammabilità (°F)
Not applicable
Punto d’infiammabilità (°C)
Not applicable
Dispositivi di protezione individuale
dust mask type N95 (US), Eyeshields, Gloves
Certificati d'analisi (COA)
Cerca il Certificati d'analisi (COA) digitando il numero di lotto/batch corrispondente. I numeri di lotto o di batch sono stampati sull'etichetta dei prodotti dopo la parola ‘Lotto’ o ‘Batch’.
Possiedi già questo prodotto?
I documenti relativi ai prodotti acquistati recentemente sono disponibili nell’Archivio dei documenti.
Il team dei nostri ricercatori vanta grande esperienza in tutte le aree della ricerca quali Life Science, scienza dei materiali, sintesi chimica, cromatografia, discipline analitiche, ecc..
Contatta l'Assistenza Tecnica.