クロマチン免疫沈降(ChIP)法
ChIP法とは?
クロマチン免疫沈降(ChIP)法は、転写因子-DNA相互作用の評価に用いられ、遺伝子発現調節やエピジェネティック修飾の研究の発展にとって極めて重要です。ChIPを使って、in vivoの単一部位または複数部位での特異的なタンパク質-DNA相互作用やタンパク質-タンパク質相互作用を検出し、相対的に定量することができます。ChIPでは、タンパク質とDNA配列を化学的に架橋した後、架橋された複合体の免疫沈降を行い(図1)、得られたDNAをエンドポイントまたは定量ポリメラーゼ連鎖反応(qPCR)(図2~4)、マイクロアレイ(ChIP-chip)、あるいは次世代シーケンシング(ChIP-seq)(図5、6)によって解析します。
クロマチン免疫沈降(ChIP)法ワークフロー

図1.ChIP手順のワークフロー
Song et al., 2015
生体細胞または組織内のタンパク質とそれに関連するクロマチンを、ホルムアルデヒドを用いて架橋します。架橋されたDNA-タンパク質複合体(クロマチン-タンパク質)を、酵素消化または超音波処理による物理的せん断を用いて約500 bpのDNA断片にせん断します。次に、DNA-タンパク質複合体を、適切なタンパク質特異的抗体によって免疫沈降させます。架橋を反転させた後、結合したDNA断片を溶出し、続いて架橋された複合体の免疫沈降を行い、得られたDNAをエンドポイントまたは定量ポリメラーゼ連鎖反応(qPCR)、マイクロアレイ(ChIP-chip)、あるいは次世代シーケンシング(ChIP-seq)によって解析します。
ChIP法が利用可能な研究
- 複数の特異的タンパク質標的が占有するDNA配列
- 特定の細胞条件下における、ゲノム全体にわたる転写因子、転写補因子、DNA複製因子、DNA修復タンパク質などの特定のタンパク質の結合部位および分布
- 遺伝子の転写とポリメラーゼ活性
- 疾患表現型の基礎となる複雑なDNA/タンパク質相互作用
- クロマチンの構造や遺伝子発現に影響を及ぼす可能性のあるヒストンなどのタンパク質の修飾
- ヌクレオソームの構築と染色体維持の調節
細胞に由来するDNA配列のChIP濃縮

図2.細胞株サンプルを用いたChIP
組織サンプルを用いた効果的なChIP法

図3.組織サンプルを用いたChIP
上段:凍結切片組織と1 mmのマイクロダイセクション法によって切り抜かれ単離された領域特異的な組織サンプル。領域特異的な組織単離の一例を画像内に示します。海馬(A)および大脳皮質(B)を領域特異的にマイクロダイセクションした300 µmのマウス冠状断脳切片。単離された組織を切り出された領域の上に置きました(a:海馬、b:大脳皮質)。次に、精製した組織を組織安定化溶液で分散させた後、ホルムアルデヒドで処理しました。ホルムアルデヒドはタンパク質とDNAを架橋し、共沈させます。
下段:Magna ChIP® G組織キット(製品番号17-20000)を用いて、さまざまな細胞株に由来するクロマチンを表示した抗体で免疫沈降を行いました。相対比較に用いたIgGには、ChIP抗体に応じてウサギ精製IgG(製品番号PP64B)またはマウス精製IgG(製品番号12- 371B)のいずれかを使用しました。定量PCRデータを独立した実験によるIgGに対する相対濃縮倍率またはインプット率(%)として示します。生物学的ネガティブコントロールとして、Dhfr遺伝子上流のプライマー(UpSt (-))を用いてqPCRを評価しました。使用した抗体とプライマーは以下の通りです。抗RNAポリメラーゼIIクローンCTD4H8(製品番号05-623B)、アフィニティー精製済みマウスモノクローナル抗体1 µgをさまざまなマウス組織のクロマチンと免疫沈降させ、マウスGAPDHプロモーターに特異的なプライマーを用いて行ったqPCRと抗SP1抗体(製品番号07-645)、アフィニティー精製済みマウスモノクローナル抗体1 µgをさまざまなマウス組織のクロマチンと免疫沈降させ、マウスDhfrプロモーターに特異的なプライマーを用いて行ったqPCR。
少量のクロマチンによる高い濃縮倍率/ChIPアッセイ処理時間の短縮

図4.ChIPキットは、他メーカーのキットと比べて少量のクロマチンでより高い濃縮倍率、もしくは処理時間の短縮を実現する
上段:HeLa細胞から調製した10,000細胞相当の超音波処理クロマチンを、精製IgG(ウサギIgG、製品番号12-370)または特異的抗体(H3K4Me3、製品番号17-614、Phospho-CREB、製品番号17-10131)1 µgを用いてクロマチン免疫沈降しました。各アッセイでは、Magna ChIP® HiSensキット(製品番号17-10460)付属の試薬およびプロトコルのほか、サプライヤーAまたはサプライヤーDの低インプットChIPキットの試薬およびプロトコルを使用しました。ポジティブコントロールプライマー(H3K4me3に対してGAPDHプロモータープライマー、pCREBに対してc-Fosプロモータープライマー)とネガティブコントロールプライマー(ヒトβ-グロビンプロモータープライマー)を用いたqPCRにより抗体に結合したDNA断片の免疫沈降を検証しました。結果は、qPCR反応あたりの総DNA 50 µL中2 µLの分析結果を表しています。
下段:Imprint®クロマチン免疫沈降キット(製品番号ChIP1)用のフレキシブルなストリップウェルフォーマットでは、ChIP反応が6時間以内に完了します。さまざまなクロマチン免疫沈降キットを用いたときの固定から精製までのプロトコル完了に要する時間の比較。各ChIP実験は、それぞれのサプライヤーが推奨するプロトコルを用いて実施し、総所要時間を比較しました。Imprint® CHP1のプロトコルに従って、オプションの高感度法により、HeLa細胞を計数、固定、免疫沈降しました。抗H3K9ac抗体(H9286)とキット付属のヒトRNAポリメラーゼIIに対する抗体および非特異的マウスIgGを用いて、ChIP法を行いました。ChIP濃縮を評価するために、GAPDHプロモーター領域(高発現のハウスキーピング遺伝子)を標的としてSYBR® qPCRを行いました。インプット率(%)は、抗体選択の有無によるDNAの量を表します。これらの抗体については、非特異的なプルダウンの減少による反応あたりの細胞数の減少とともに、見かけの濃縮度が増加しました。
次世代シーケシングによるChIP DNAの解析(ChIP-seq)


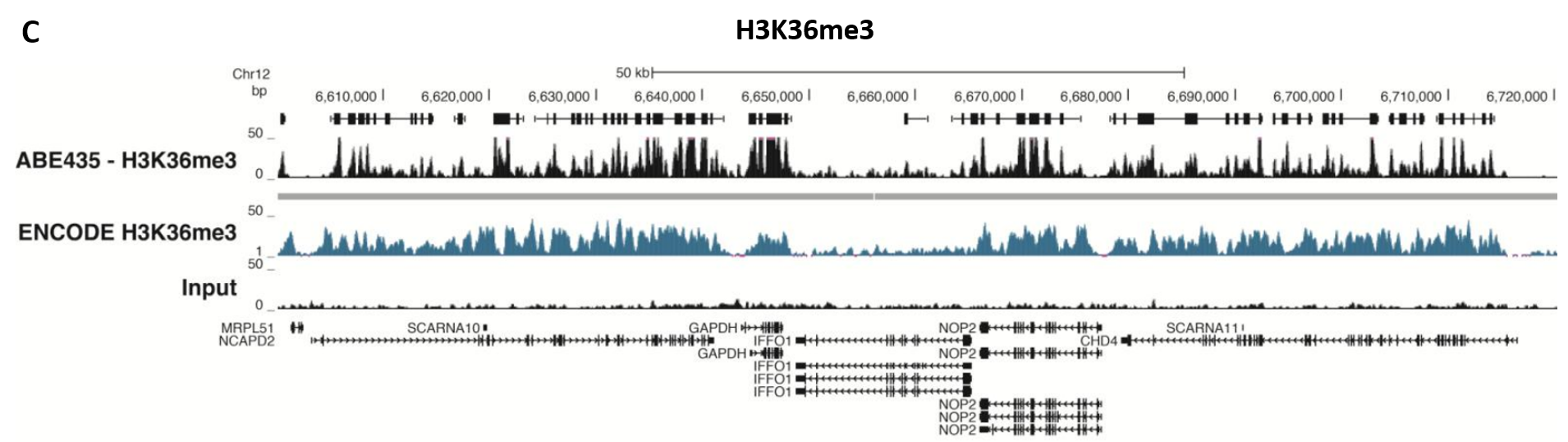
図5.ChIP-Seq解析
Magna ChIP® HiSensキット(製品番号17-10460)、抗H3K4me2抗体2 µL、2 µgまたは5 µg(製品番号04-790、05-1338またはABE250)、抗H3K4me3抗体1µL 、3 µLまたは4 µg(製品番号05-745R、07-473または05-1339)、抗H3K36me3抗体2 µg(製品番号ABE435)、プロテインA/G磁気ビーズ20 µL、および1e6、4e6または5e6個の架橋されたHeLa細胞クロマチンを用いて、クロマチン免疫沈降を行った後、DNAを精製しました。標準プロトコルに従い、Illumina®バーコード付きアダプターを用いて、インプットサンプルとChIP DNAサンプルからライブラリを作製し、Illumina® HiSeq®装置で解析しました。TagDust(http://genome.gsc.riken.jp/osc/english/dataresource/)タグ除去に続いて、Bowtie(http://bowtiebio.sourceforge.net/manual.shtml)を用いて、FastQファイルから1,200万を超えるリードをマッピングしました。MACS(http://luelab.dfci.harvard.edu/MACS)を用いてピークを同定し、BigWigおよびBEDファイルから、ピークとリードをUCSC Genome Browser (http://genome.ucsc.edu)におけるカスタムトラックとして可視化しました。データにおいて同定されたピークの上位25%は、HeLa S3のENCODE H3K4me2またはH3K4me3またはH3K36me3 BROADヒストントラックで同定されたピークと、90~99%重複していました。転写活性のあるハウスキーピング遺伝子TUBA1A(チューブリンα1A)、HOXB、ACTBまたはGAPDHの領域のデータを示します。
少量のDNAを用いたChIP法と次世代シーケンシング

図6.限られた量のDNAからの効果的なChIPと信頼性の高い次世代シーケンシングライブラリ構築
Sp1 ChIP DNAから、定量された免疫沈降DNA 10 ng(上段)または1 ng(中段)を用いて、次世代シーケンシングライブラリを構築しました。参照ライブラリは、インプットクロマチンに由来する精製DNA 10 ngを用いて作製しました。Magna ChIP®-Seqキット(製品番号17-1010)とChIPab+Sp1抗体/プライマーセット(製品番号17-601)を用いて、サンプルを調製しました。得られたライブラリはIllumina® Genome Analyzerで配列決定を行った後、ヒトhg18参照ゲノムにアラインメントおよびマッピングしました。上に示したのは、DNAnexus®ソフトウェアで読んだ、明確にマッピングされたリードから得られた、Sp1遺伝子座のピーク分析(QuESTを用いて導出)です。10 ngおよび1 ngのライブラリからシーケンシングを反復して比較すると、Sp1遺伝子のプロモーターにおけるピーク同定の重複が認められます。三角は、このゲノム範囲において、主に転写開始部位近傍で発生する、高確率のSp1結合イベントを表します。
ChIPの実験ガイド、FAQ、トラブルシューティングのヒントおよび補足プロトコル
ChIPは、最も経験豊富な研究者にとってさえ困難であることがあります。ChIPは、信頼性が高く再現性のある結果を生み出すために、質の高いクロマチン、強固な抗体、最適化された試薬とプロトコルを必要とするマルチステップの手法です(図1)。これらは通常、市販の製品とプロトコルを用いるか、複数の検証実験を行うことによって達成されます。どちらの戦略を選ぶにしても、以下にサンプル調製から下流のDNA解析まで、ChIPにおいてよく起こる問題のいくつかに対処するための包括的なChIP実験ガイドを示します。
- ChIP・エンドポイント解析のための細胞数ガイド
- アガロースビーズと磁気ビーズの比較
- タンパク質とDNAの架橋・細胞溶解
- 免疫沈降・洗浄・溶出
- ChIP-qPCRとデータ解析(インプット率[%]および濃縮倍率)
- ChIP-Seqにおけるピークコールのガイド
- FAQ(抗体、融合タグ、架橋とビーズ、クロマチン断片化、データ解析)
- トラブルシューティングのヒント(バックグラウンドが高い、DNA回収率が低い、DNA増幅がない、大きなDNAしかプルダウンされない、非特異的なDNAが沈殿してしまう)
- 補足プロトコル
おすすめのChIPキット
標準ChIPキット
特殊なChIPキット
ChIP付属製品
続きを確認するには、ログインするか、新規登録が必要です。
アカウントをお持ちではありませんか?