T1660
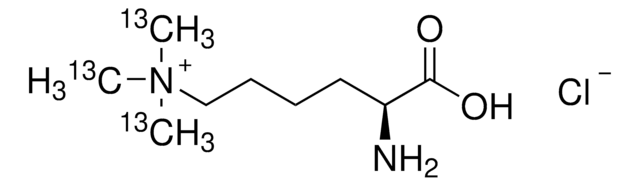
Nε,Nε,Nε-Trimethyllysine hydrochloride
≥97% (TLC)
Sinónimos:
1-Pentanaminium, 5-amino-5-carboxy-N,N,N-trimethyl-, chloride (1:1), (5S)-
About This Item
Productos recomendados
Nivel de calidad
Ensayo
≥97% (TLC)
Formulario
powder
contiene
salts and water as balance
composición
Amino acid content, ~75%
técnicas
LC/MS: suitable
color
white
temp. de almacenamiento
−20°C
cadena SMILES
[Cl-].C[N+](C)(C)CCCC[C@H](N)C(O)=O
InChI
1S/C9H20N2O2.ClH/c1-11(2,3)7-5-4-6-8(10)9(12)13;/h8H,4-7,10H2,1-3H3;1H/t8-;/m0./s1
Clave InChI
ZKIJKCHCMFGMCM-QRPNPIFTSA-N
Categorías relacionadas
Código de clase de almacenamiento
11 - Combustible Solids
Clase de riesgo para el agua (WGK)
WGK 3
Punto de inflamabilidad (°F)
Not applicable
Punto de inflamabilidad (°C)
Not applicable
Equipo de protección personal
Eyeshields, Gloves, type N95 (US)
Elija entre una de las versiones más recientes:
¿Ya tiene este producto?
Encuentre la documentación para los productos que ha comprado recientemente en la Biblioteca de documentos.
Los clientes también vieron
Nuestro equipo de científicos tiene experiencia en todas las áreas de investigación: Ciencias de la vida, Ciencia de los materiales, Síntesis química, Cromatografía, Analítica y muchas otras.
Póngase en contacto con el Servicio técnico