推荐产品
生物源
rabbit
品質等級
抗體表格
purified antibody
抗體產品種類
primary antibodies
無性繁殖
RM172, monoclonal
物種活性
human
技術
multiplexing: suitable
western blot: suitable
同型
IgG
UniProt登錄號
運輸包裝
wet ice
目標翻譯後修改
acetylation (Lys27)
基因資訊
human ... H3F3B(3021)
一般說明
组蛋白H3,也称为组蛋白H3.1t (H3/t)、H3t、H3/g,由基因名称HIST3H3/H3FT编码,是核小体的核心组分。核小体将DNA包裹并压缩到染色质中,从而限制了DNA进入需要DNA作为模板的细胞机制的可及性。因此,组蛋白在转录调控、DNA修复、DNA复制和染色体稳定性中起着核心作用。H3具有一个主要的球状结构域和一个长N末端的尾巴,与串珠′结构上的′珠核小体结构有关。组蛋白H3的N末端尾巴从球状核小体核心突出,并且可以经历影响细胞过程的几种不同类型的表观遗传修饰。这些修饰包括甲基或乙酰基与赖氨酸和精氨酸氨基酸的共价连接以及丝氨酸或苏氨酸的磷酸化。组蛋白H3的高水平磷酸化与有丝分裂有关。
特異性
该抗体可与赖氨酸27乙酰化的组蛋白H3(K27ac)反应。与组蛋白H3中的其他乙酰化赖氨酸无交叉反应性。
免疫原
对应于在Lys27乙酰化的人组蛋白H3的线性肽。
應用
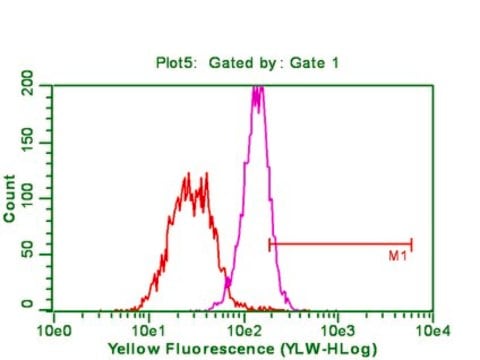
多重分析(Mplex):使用一个代表性批次以0.1-0.5 μg/mL进行多重分析。
研究子类别
染色质生物学
染色质生物学
研究类别
表观遗传学&核功能
表观遗传学&核功能
经验证,该抗乙酰基组蛋白H3(Lys27)抗体(克隆RM172)可用于蛋白质印迹法(多重检测乙酰基组蛋白H3)。
品質
通过蛋白质印迹法在未处理和丁酸钠处理的HeLa酸提取物中进行评价。
蛋白质印迹分析(WB):1 μg/mL该抗体可在丁酸钠处理的HeLa酸性提取物中检测到乙酰基组蛋白H3(Lys27)。
蛋白质印迹分析(WB):1 μg/mL该抗体可在丁酸钠处理的HeLa酸性提取物中检测到乙酰基组蛋白H3(Lys27)。
標靶描述
观测分子量〜17 kDa。 在某些裂解物中可以观察到未鉴定的条带。
外觀
兔单克隆抗体,溶于含1% BSA和0.09%叠氮化钠的PBS中
形式:纯化
纯化蛋白A
儲存和穩定性
自接收之日起,在2-8°C下可稳定保存1年。
注意:冷冻箱温度变化至低于-20°C时可能导致含甘油的溶液在储存过程中冻结。
注意:冷冻箱温度变化至低于-20°C时可能导致含甘油的溶液在储存过程中冻结。
其他說明
浓度:请参考特定批次的数据表。
免責聲明
除非我们的目录或产品随附的其他公司文件中另有说明,否则我们的产品预期仅用于研究用途,不得用于任何其他目的,包括但不限于未经授权的商业用途、体外诊断用途、离体或体内治疗用途或对人类或动物的任何类型的消费或应用。
Not finding the right product?
Try our 产品选型工具.
儲存類別代碼
12 - Non Combustible Liquids
水污染物質分類(WGK)
WGK 2
閃點(°F)
does not flash
閃點(°C)
does not flash
Michael S Hoetker et al.
Nature cell biology, 25(8), 1121-1134 (2023-07-18)
The epigenetic mechanisms that maintain differentiated cell states remain incompletely understood. Here we employed histone mutants to uncover a crucial role for H3K36 methylation in the maintenance of cell identities across diverse developmental contexts. Focusing on the experimental induction of
Steven J Wu et al.
Nature biotechnology, 39(7), 819-824 (2021-04-14)
Methods for quantifying gene expression1 and chromatin accessibility2 in single cells are well established, but single-cell analysis of chromatin regions with specific histone modifications has been technically challenging. In this study, we adapted the CUT&Tag method3 to scalable nanowell and
Chi Sun et al.
Scientific reports, 13(1), 12899-12899 (2023-08-10)
Enhancers function with a basal promoter to control the transcription of target genes. Enhancer regulatory activity is often studied using reporter-based transgene assays. However, unmatched results have been reported when selected enhancers are silenced in situ. In this study, using
Derek H Janssens et al.
Epigenetics & chromatin, 11(1), 74-74 (2018-12-24)
Our understanding of eukaryotic gene regulation is limited by the complexity of protein-DNA interactions that comprise the chromatin landscape and by inefficient methods for characterizing these interactions. We recently introduced CUT&RUN, an antibody-targeted nuclease cleavage method that profiles DNA-binding proteins
Hatice S Kaya-Okur et al.
Nature communications, 10(1), 1930-1930 (2019-05-01)
Many chromatin features play critical roles in regulating gene expression. A complete understanding of gene regulation will require the mapping of specific chromatin features in small samples of cells at high resolution. Here we describe Cleavage Under Targets and Tagmentation
我们的科学家团队拥有各种研究领域经验,包括生命科学、材料科学、化学合成、色谱、分析及许多其他领域.
联系技术服务部门