简介
荧光染料或探针包含在PCR混合物中,用以监测在反应进行过程中DNA扩增子浓度的变化。本章将讨论几种常用的检测方法,这些方法适用于间接测量qPCR中的模板。此外,双标记探针和一些DNA结合染料已被证明在数字PCR(dPCR)中有效。
dsDNA结合染料
双链DNA(dsDNA)结合染料起着嵌入和/或小沟结合剂的作用,当与dsDNA结合时发出可检测的荧光,但当在溶液中游离时则具有非常低的背景。因此,荧光信号强度与存在的扩增子的量成比例地增加。双链DNA结合染料很受欢迎,因为它们是低成本检测选择,不需要额外的设计考虑。
SYBR®Green I 染料
该染料是最受欢迎的dsDNA结合染料,在分子生物学中具有悠久的应用历史。当在溶液中游离、且仅存在单链DNA(ssDNA)时,SYBR Green I 染料发出低强度信号(图5.1,表5.1)。随着PCR的进行和dsDNA数量的增加,更多的染料与扩增子结合,因此信号强度增加(参见以下网页上的动画:sigma.com/sybr-animation)。然而,由于染料不加选择地与所有扩增产物结合,因此诸如由引物二聚体或引物的非特异性结合产生的那些假象也提高了整体荧光水平。这可能使得难以获得准确的定量,尤其是在低模板浓度下。然而,PCR后的解链曲线分析有助于确定反应特异性1(图5.2)。

图 5.1.随着反应的进行,SYBR Green I 染料在未结合(变性)和已结合(延伸退火)状态之间循环,随着扩增子数量的增加,信号强度增加。
a当与dsDNA复合时

图 5.2.解链曲线分析示例。dsDNA结合染料可逆地结合,因此随着反应温度升高至高于解链温度(Tm),荧光强度降低。与对照相结合,这种类型的分析能够检测在不同于特异性产物的温度下熔化的非特异产物。
探针
在qPCR的所有应用中,扩增反应由特异性正向和反向引物驱动。然而,与依赖于使用dsDNA结合染料的检测的测定不同,探针检测系统不包括游离染料,而是包含与报告染料和/或淬灭基团缀合的第三寡核苷酸(有时是第四寡核苷酸)。
双标记探针
双标记探针(亦称为水解或TaqMan®探针)用于5’核酸酶测定2,3,这是最流行的探针检测化学(图5.3;亦请参见以下网页的动画:sigma.com/probe-animation)。双标记探针是单链寡核苷酸,它用报告染料和淬灭剂标记。报告基团位于5'末端,淬灭基团位于3'末端。淬灭基团通常通过福斯特型能量转移(更常称为荧光共振能量转移(FRET))吸收报告基团的天然荧光发射。从正向引物扩增后,Taq DNA聚合酶遇到探针。然后Taq DNA聚合酶中固有的5’外切核酸酶活性将5’报告基团与3’淬灭基团分开(表5.2,图5.3),从而产生与扩增子产量成比例的荧光信号。
水解探针测定对于低拷贝数靶标的定量是特异且准确的。通过在探针中包含修饰的核苷酸如锁核酸(如下所述),可以进一步提高特异性。锁核酸修饰的探针特别适用于区分单核苷酸多态性(SNP)或其他类似序列。双标记探针需要仔细设计(参见PCR/qPCR/dPCR 测定设计),并且通常比dsDNA结合染料更昂贵。此外,虽然非特异性产物的扩增可能仍未检测到,但是副反应会导致整体反应效率较低,因此含有探针的测定仍然可以从优化中获益(参见样本纯化和质量评估)。

图 5.3.双标记探针的机理。Taq DNA聚合酶延伸位于与探针同一链上的引物,直至其到达探针位置。固有的外切核酸酶活性将探针从5'水解至3',将报告染料释放到溶液中,从而造成荧光增加。测得的荧光信号与靶 DNA 的量成正比。
大多数实时PCR热循环仪具有多个检测通道,可灵活选择探针标记。选择与仪器检测通道相容的报告染料、配置正确的滤光器、以及进行校准,都至关重要。当多重测定时,需要尽可能彼此不同的报告基团组合,以最大限度地减小光学串扰。典型报告基因包括:FAM、HEX、TxRd (硫代罗丹明101-X) 和花青素 5。在相同条件下,通常观察来自不同报告基团的发射强度的差异。因此,建议单独分析每个报告基团组合的数据(使用适合探针发射的不同阈值设置)。
分子信标
Molecular Beacon(亦称为杂交探针)是单链探针,其通过每个末端处的互补茎序列(4-6个核苷酸)保持发夹环构象(20-25个核苷酸)4(图5.4)。发夹环与模板互补,并且当探针在溶液中游离时,氢键结合的茎序列允许3'淬灭基团抑制5'报告基团的荧光(表5.3)。
图 5.4.Molecular Beacon的机理。Molecular Beacon与其特定靶序列杂交,导致发夹环结构打开,将5'报告基团与3'淬灭基团分开。由于猝灭分子不再靠近报告分子,荧光发射发生。与双标记探针不同,Molecular Beacon的检测机理不依赖于反应过程中的降解。测得的荧光信号与靶 DNA 的量成正比。
LightCycler® 探针
LightCycler探针或FRET系统(亦称为双杂交探针)由一对单链荧光标记的寡核苷酸组成5,6(图5.5)。寡核苷酸探针1在3'末端用供体荧光团染料标记,寡核苷酸探针2在5'末端用少数可用的受体荧光团染料中的一种标记(表5.4)。寡核苷酸探针2的游离3'羟基必须用磷酸基团封闭,以防止DNA聚合酶延伸。

图 5.5.LightCycler FRET探针的机理。在退火步骤中,引物和两种探针与它们的特定靶区域杂交,使供体染料紧密接近受体染料(探针通常间隔1至5个核苷酸)。当供体被来自实时PCR仪的光激发时,能量通过FRET从供体转移到受体。检测受体探针上染料的发射波长。荧光信号的增加与靶DNA的量成正比。
Scorpions® 探针
Scorpions®探针有两种形式:单探针和双探针。单探针由一个茎环结构组成,类似于Molecular Beacon,但它与正向引物连接,PCR阻断基团位于两个寡核苷酸区段之间7(阻断基团阻止Taq DNA聚合酶延伸引物)(图5.6A,表5.5)。
双探针结构是双链体,在一条链上有5'报告基团、探针序列、PCR阻断基团和正向引物,在另一条链上有3'淬灭基团,淬灭基团与报告基团链双链化(图5.6B)。

图 5.6.Scorpions®探针的机理。单探针 A)在一条链上具有所有探针组分,而双探针 B)在两条链上具有探针组分。两种形式的Scorpions®探针都包括正向PCR引物。该正向引物延伸成为新形成的扩增子的一部分。在退火/延伸期间,Scorpions®中的探针序列与模板杂交,将报告基团与猝灭基团分开,从而产生荧光信号。由于Scorpions®的尾部和扩增子都是同一条链的一部分,因此检测相互作用是分子内的,也因此比其他探针检测系统更快。通常选择距离Scorpions®引物的3’末端5至50个碱基的模板。Scorpions®单探针和双探针都需要单独的反向引物。
淬灭基团
大多数探针检测系统需要淬灭剂。在原始探针结构中使用的那些淬灭剂中,有些是受体荧光染料,例如TAMRA,其与FAM配合良好,但不适用于其他染料。作为报告染料本身,TAMRA产生荧光,因此可导致较差的信噪比。出于这个原因,Biosearch Technologies开发了暗淬火剂,它们发热而不发光,例如Black Hole Quencher®(BHQ®)。选择暗淬灭剂作为染料分子的常用替代品,可在很宽的波长范围内进行淬灭,使含有大量靶 / 探针组合的多重反应成为可能。
Onyx Quencher™(OQ™)是Sigma-Aldrich的专有暗淬灭剂。有四种衍生版本(OQA、OQB、OQC和OQD)可供选择。如表5.6所示,四种Onyx淬灭剂与各种流行的报告染料相容。
图5.7中给出的数据显示了人工模板的扩增,该模板来自合成寡核苷酸,用于优化曼氏血吸虫靶标测定8。检测使用FAM标记的探针以及相当的暗淬灭剂:CDQ(A)或OQA(B)。从这些数据可以看出,CDQ和OQA具有相同的性能、相似的背景荧光、和相似的研究数据Cq值,由相同浓度的模板得来。
总之,OQ在性能上与CDQ相当,重要的是,对任何应用,许可证和版税都是免费的,不受限制。这使得Onyx淬灭剂成为开发含有qPCR探针的商用分子诊断试剂盒和试剂的一种性能卓越、且具成本效益的选择。
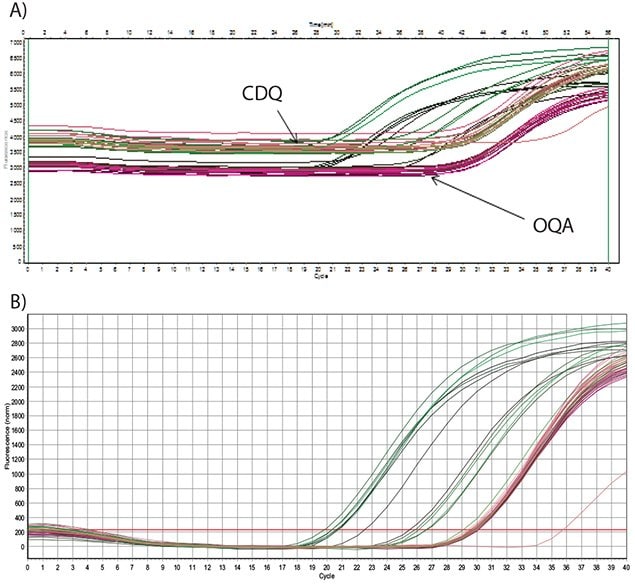
图 5.7.使用250 nM引物扩增人工寡核苷酸的稀释液,并用200 nM的双标记探针检测扩增子。A)探针用FAM标记,并用CDQ或OQA淬灭(如图所示),图中显示的是原始荧光数据。B)探针用FAM标记,并用CDQ或OQA淬灭,图中显示的是基线校正数据。两种探针的性能没有显著差异。
核酸类似物
许多修饰可用来产生具有改变的生化性质的寡核苷酸。这些修饰通常提供更高的Tm,因此对其加以控制可改善特异性。这使得能够设计测定用于具有挑战性的序列区域,或用于要求用单个寡核苷酸检测几种序列(例如一个病毒的所有血清型)时。
锁核酸
锁核酸是一种RNA类似物碱基(图5.8),当掺入探针时可提高灵敏度和特异性9。具有锁核酸碱基的探针具有更高的热稳定性,因此与模板产生更强的杂交。每种锁核酸碱基可以将探针的Tm提高8°C10,这使得锁核酸成为一个功能强大的工具,用于SNP鉴别测定11(单个碱基错配对双链体形成具有比没有锁核酸时更大的去稳定作用)、多重测定(使Tm优化更简单)、以及有问题的靶序列(锁核酸探针可以更短,这使它们可以围绕具体问题加以设计,这些问题包括富含AT或GC的区域、重复序列或具有显著二级结构的序列)。还发现,含有锁核酸的PCR引物在诸如SNP基因分型的应用中是有益的12。
锁核酸还提供核酸酶消化保护,使其适合体内使用13。

图 5.8.锁核酸与DNA核苷酸结构的比较。锁核酸与DNA的不同之处在于,锁核酸含有核糖,在2’氧和4’碳之间具有亚甲基桥,其在3’内构象中“锁住”核糖,而DNA含有2’-脱氧核糖,没有亚甲基桥。
空格表示不推荐检测方法;X= 检测方法性能良好;XX= 检测方法性能更好。
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?