寡核苷酸是一种短片段的核酸单链或双链聚合物。当暴露于260 nm紫外光(UV)时,溶液中核酸的浓度很容易在分光光度计中定量(图1)。该技术的工作原理是,碱基缀合到具有不同UV吸光度的双键上。

图 1.分光光度计中的吸光度。样品池中溶液中的核酸由260 nm处最大强度的UV入射光照射。由于核酸碱基吸收一些UV光,因此UV透射光的强度降低。吸光度由以下公式确定:
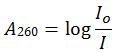
式中:A260 = 260 nm处的吸光度;Io = 入射光强度;I = 透射光强度。
在读取吸光度之后,即可使用比尔-朗伯定律,该定律将吸光度与吸收核酸的浓度联系起来,如下所示:
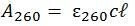
式中:
A260 = 260 nm处的吸光度
260 = 寡核苷酸在260 nm处的消光系数,以L ⋅ mol-1 ⋅ cm-1为单位
c = 浓度,以mol L-1为单位
ℓ = 光程,以cm为单位
比尔-朗伯定律的关键是消光系数,它测量吸收碱基从UV的入射光到透射光减少了多少光强。由于每个碱基的吸光度不同,碱基组成、碱基顺序、和序列长度都会影响定量中特定核酸的最终吸光度。因此,如果计算出每种寡核苷酸的消光系数,就可实现最高准确度。最邻近模型是测定寡核苷酸的理想选择,公式如下:

表1和2显示了在计算寡核苷酸的最终总消光系数时,在最邻近公式中使用的最邻近碱基和个体碱基的消光系数。
已知样品池的路径长度为1.0 cm,比尔-朗伯定律每个输入的含义也已明确,现将公式重新排列,即可计算所讨论的寡核苷酸的浓度。
为计算浓度,现将比尔-朗伯定律重新排列如下:

举例:5'-ACGT-3'的计算方法
第一步:计算消光系数
260 = (AC+CG+GT)-(C+G) L ⋅ mol-1 ⋅ cm-1
260 = (21,200+18,000+20,000)-(7,400+11,500) L ⋅ mol-1 ⋅ cm-1
260 = 40,300 L ⋅ mol-1 ⋅ cm-1
在所有网上技术信息和印刷的文件中,上述值40,300显示为40.3。这是因为单位是L ⋅ mmol-1 ⋅ cm-1(是毫摩尔而不是摩尔)。
第二步:计算浓度
A260设定为1.0光密度(OD)单位



第三步:计算摩尔质量(MM)



通常用“分子量”代替“摩尔质量”,但由于这些计算需要摩尔质量单位,因此“摩尔质量”是最正确的术语。而且,为了简化显示,每个碱基的上述摩尔质量已经四舍五入到最接近的整数。因此,计算值1,170略低于真实值1,173.83。
第四步:将浓度换算为常用单位

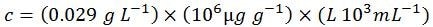

因此,对于该特定序列:5'-ACGT-3',1 OD = 29.0 μg mL-1(一个OD是1 mL溶液中寡核苷酸的量,其在1.0 cm的光程长度中显示出A260为1.0)。作为比较,这是由我们的在线寡核苷酸序列计算器OligoEvaluator™确定的值:

手动计算的29.0值与OligoEvaluator的计算值29.1非常吻和(差异是因为在手动计算中,每个碱基摩尔质量被四舍五入,如上述第三步所述)。
除了OligoEvaluator之外,29.1也是这种寡核苷酸所附的技术数据表中给出的值。
5'-ACGT-3'的快捷计算方法
上述详细计算步骤太繁琐。下列公式将所有步骤合并为一个快捷的计算方式:



修饰
标准和修饰寡核苷酸的浓度计算之间的唯一差异,在于后考虑了修饰的摩尔质量,并将其加到寡核苷酸的总摩尔质量中。例如,如果我们的上述序列:
● 5'-ACGT-3'
被改为:
● 5'-6-FAM™-ACGT-3'
将新的摩尔质量1,711.3 g mol-1代入详细步骤计算或快捷公式,即可得到新浓度为:
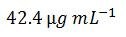
该值与OligoEvaluator确定的值高度吻合:

快速换算
实验已经验证了如下快速换算,可以作为经验法则(ds =双链;ss =单链):
● A260 dsDNA = 50 µg mL-1
● A260 ssDNA = 37 µg mL-1
● A260 ssRNA = 40 µg mL-1
在不进行任何计算的情况下,这些换算值为具有比较均匀的A、C、G和T碱基分布的较长序列提供了合理的估算值,例如通过剪切基因组DNA产生的序列(对于dsDNA估算)。然而,对于寡核苷酸,碱基组成更可能倾斜,因此,这些估计值是不可靠的(通常高估浓度)。因此,必须使用上述的详细步骤计算或快捷公式计算来确定每个寡核苷酸的浓度。详细步骤计算或快捷公式计算可用于验证技术数据表中提供的定量值。
如要继续阅读,请登录或创建帐户。
暂无帐户?