643440
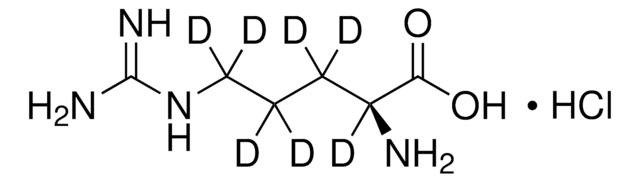
L-Arginine-13C6 hydrochloride
99 atom % 13C, 95% (CP)
Synonyme(s) :
SILAC Amino Acid, (S)-(+)-2-Amino-5-[(aminomethyl)amino]pentanoic acid-13C6 hydrochloride, 13C Labeled arginine, 13C Labeled arginine hydrochloride
About This Item
Produits recommandés
Pureté isotopique
99 atom % 13C
Niveau de qualité
Pureté
95% (CP)
Forme
solid
Activité optique
[α]20/D +22°, c = 12 in 10% HCl
Technique(s)
protein expression: suitable
Pf
226-230 °C (dec.) (lit.)
Changement de masse
M+6
Chaîne SMILES
Cl[H].N[13C@@H]([13CH2][13CH2][13CH2]N[13C](N)=N)[13C](O)=O
InChI
1S/C6H14N4O2.ClH/c7-4(5(11)12)2-1-3-10-6(8)9;/h4H,1-3,7H2,(H,11,12)(H4,8,9,10);1H/t4-;/m0./s1/i1+1,2+1,3+1,4+1,5+1,6+1;
Clé InChI
KWTQSFXGGICVPE-BJPSCUOKSA-N
Vous recherchez des produits similaires ? Visite Guide de comparaison des produits
Application
Conditionnement
Produit(s) apparenté(s)
Code de la classe de stockage
11 - Combustible Solids
Classe de danger pour l'eau (WGK)
WGK 3
Point d'éclair (°F)
Not applicable
Point d'éclair (°C)
Not applicable
Faites votre choix parmi les versions les plus récentes :
Déjà en possession de ce produit ?
Retrouvez la documentation relative aux produits que vous avez récemment achetés dans la Bibliothèque de documents.
Les clients ont également consulté
Contenu apparenté
La structure des protéines fournit des informations utiles qui peuvent être exploitées pour déduire leur fonction. L'étude de la structure des protéines et la cartographie des interactions entre protéines, de leur niveau d'expression et de leur localisation permettent d'identifier des biomarqueurs de maladies ainsi que des cibles thérapeutiques potentielles.
La structure des protéines fournit des informations utiles qui peuvent être exploitées pour déduire leur fonction. L'étude de la structure des protéines et la cartographie des interactions entre protéines, de leur niveau d'expression et de leur localisation permettent d'identifier des biomarqueurs de maladies ainsi que des cibles thérapeutiques potentielles.
La structure des protéines fournit des informations utiles qui peuvent être exploitées pour déduire leur fonction. L'étude de la structure des protéines et la cartographie des interactions entre protéines, de leur niveau d'expression et de leur localisation permettent d'identifier des biomarqueurs de maladies ainsi que des cibles thérapeutiques potentielles.
Protein structure analysis aids in identifying disease biomarkers and drug targets crucial for therapeutic treatments.
Notre équipe de scientifiques dispose d'une expérience dans tous les secteurs de la recherche, notamment en sciences de la vie, science des matériaux, synthèse chimique, chromatographie, analyse et dans de nombreux autres domaines..
Contacter notre Service technique