MISSION® esiRNA-Spezifikationen
Kundenspezifische & vordefinierte esiRNA
Seit Ende Oktober/Anfang November 2020 werden esiRNA-Produkte, die in Röhrchen geliefert werden – einzelne esiRNA, esiOPEN und esiSEC – nicht mehr in Lösung geliefert, sondern lyophilisiert (esiRNA-Produkte in Platten – esiLibrary und esiFLEX – werden weiterhin in Lösung geliefert). Wir nehmen diese Änderung bei Produkten in Röhrchen (die meisten esiRNA werden in Röhrchen geliefert) aus folgenden Gründen vor:
- Lyophilisierte Materialien können bei Umgebungstemperatur versandt werden, statt gefroren auf Trockeneis. Die Reduzierung der Versandkosten unterstützt uns dabei, die Produktpreise so niedrig wie möglich zu halten.
- Aufgrund der Tatsache, dass wird das Trockeneis aus den Sendungen entfernen, benötigen wir weniger Verpackungsmaterial, was wiederum dazu beiträgt, der Verpflichtung unseres Unternehmens zur Verringerung der Umweltbelastung zu entsprechen. Als RNA ist esiRNA bei Raumtemperatur mindestens 3 Wochen haltbar. Nach Erhalt von esiRNA ist es wichtig, diese bei -20 °C bis zur Verwendung zu lagern. Anhang 2 des technischen Merkblatts zum Produkt enthält ein Resuspensionsprotokoll. Es wird eine Übergangsphase geben, in der ein Teil der esiRNA-Lieferungen weiterhin in Lösung und manche in trockener Form erfolgen. Wir werden die Umstellung auf alle Einheiten in trockener Form so schnell wie möglich voranbringen.
Alle Bestell- und Angebotsoptionen sind in der Tabelle mit den esiRNA-Spezifikationen enthalten
MISSION® esiRNA – von Eupheria Biotech – bietet RNAi-Forschern eine bewährte, kostengünstige und einfache Methode für die posttranskriptionelle Stilllegung proteincodierender Gene und lncRNA (lange, nicht-codierende RNA). Biologisch hergestellte esiRNA besteht aus einem heterogenen Pool siRNA (natürliche RNA, keine Modifikationen), die alle auf die gleiche mRNA-Sequenz abzielen. Diese multiplen Auslöser für die Genstilllegung bewirken hochspezifische und effektive Genstummschaltungen mit geringeren Off-Target-Effekten als einzelne, chemisch synthetisierte siRNA (Abbildung 1).

Abbildung 1.Der Knockdown der Ziel-mRNA kann durch chemisch-synthetisierte siRNA oder enzymatisch hergestellte siRNA (esiRNA) erreicht werden. A) Chemisch synthetisierte siRNA besteht aus einem einzelnen Auslöser für die Stilllegung von 21 bp, der komplementär zur Ziel-mRNA ist. Die hohe Konzentration der siRNA in der Transfektionsreaktion führt zu ausgeprägten Off-Target-Effekten. B) Im Gegensatz dazu besteht die esiRNA aus einem Pool Hunderter siRNA (21 bp), die einen Bereich von 300 – 600 bp der Ziel-mRNA abdecken. Jede einzelne siRNA hat eine geringere Konzentration im Pool, was zu geringeren Off-Target-Effekten führt und gleichzeitig einen effizienten Knockdown bewirkt.
Produktvorteile
- Garantierte Genstilllegung
- Geringere Off-Target-Effekte als bei einer einzelnen, chemisch synthetisierten siRNA
- Hohe On-Target-Spezifität bewirkt ein effektives primäres Screening-Tool (Abbildung 2)
- Erschwingliches RNAi-Screening-Tool im Genom-Maßstab

Abbildung 2.Bewertung des Gen-Knockdowns in HeLa-Zellen, die mit esiRNA transfiziert wurden, die gegen Renilla-Luciferase (Negativkontrolle) und verschiedene exprimierte Targets auf mRNA- und Proteinebene gerichtet ist. A) qPCR-Daten bestätigen die Knockdowns 24 Stunden nach der Transfektion. Die mRNA-Abundanz wurde in allen Transfektionen um > 90% reduziert. B) Der effiziente Knockdown wird durch quantitative Western-Blot-Daten 72 Stunden nach der Transfektion bestätigt. Der Proteingehalt wurde um 45 bis 90 % reduziert.
Produktmerkmale
Die folgenden Angaben gelten für alle in der esiRNA-Spezifikationstabelle aufgeführten Produktoptionen:
- Aufreinigung: Q-Sepharose-Trennung, Isopropanol-Ausfällung und Waschen mit Ethanol
- Sequenzform: Pool Hunderter siRNA mit einer durchschnittlichen Duplexlänge von 21 bp
- Qualitätskontrolle: Durchgeführt in zwei Stufen
o PCR-Produkt des cDNA-Klons wird durch Gelelektrophorese und DNA-Sequenzierung analysiert
o Verdauungsreaktion wird durch Gelelektrophorese analysiert - Haltbarkeit: Lebensfähig über einen Zeitraum von 2 Jahren bei Lagerung bei -20 °C
Für alle Belange, die nachstehend mit "Inquire" (Anfragen) gekennzeichnet sind, oder bei spezifischen Anforderungen, die von den anderen dargestellten allgemeinen Spezifikationen abweichen, möchten wir Sie bitten, eine Anfrage an [email protected] zu senden.
Die vollständige Liste vordefinierter esiRNA entdecken.
| esiRNA-Produktoptionen & Spezifikationen | |
|---|---|
| Individuelle esiRNA |
|
esiBibliothek |
|
esiFLEX |
|
esiOPEN |
|
esiSEC |
|
esiRNA-Kontrollen
Es gibt eine Reihe von Positiv- und Negativkontrollen (Abbildung 3):
- Positivkontrollen: Humane Eg5 (KIF11) & murine Eg5 (Kif11) esiRNA sowie alle validierten esiRNA (siehe nachstehende Tabelle) zur Sicherstellung von Transfektion und Optimierung des Versuchsaufbaus
- Negativkontrollen: RLUC, FLUC & EGFP (EGFP kann auch als Positivkontrolle in Zellen verwendet werden, die EGFP exprimieren) esiRNA zur Unterscheidung zwischen sequenzspezifischer Genstilllegung und unspezifischen Effekten

Abbildung 3.Phänotypische Analyse von HeLa-Zellen, die mit esiRNA gegen A) RLUC (Negativkontrolle) und B) Eg5/KIF11 (Positivkontrolle) transfiziert wurden. RLUC löst keine phänotypischen Veränderungen aus, während Eg5/KIF11 einen mitotischen Stillstand bewirkt, der sich in runden Zellen zeigt.
Validierte esiRNA
Viele gängige Gentargets wurden mit einem mRNA-Knockdown von ≥ 70 % validiert (qPCR- und Western-Blot-Validierungsdaten). In der Tabelle ist eine nach Gensymbolen geordnete Liste der validierten esiRNA aufgeführt. Validierte siRNA eignen sich sowohl für die Transfektionsoptimierung als auch für Positivkontrollen.
lncRNA esiRNA
Transkriptomanalysen zeigen, dass bis zu 90 % des Genoms in nicht-codierende RNA transkribiert werden, zu denen unter anderen lncRNA gehören. lncRNA werden mit verschiedenen Funktionen in Verbindung gebracht, darunter Chromatinmodifikation, Co-Aktivierung von Transkriptionsfaktoren, Transkription, Interaktion mit RNA-bindenden Proteinen und Unterdrückung von Promotern. Darüber hinaus wurden lncRNA bei Krankheiten wie Krebs impliziert. Die Einzelheiten der Funktionsweise von lncRNA müssen noch weiter erforscht werden. esiRNA sind ein wirksames Screening-Instrument zur Untersuchung der Funktion von lncRNA (Abbildung 4).
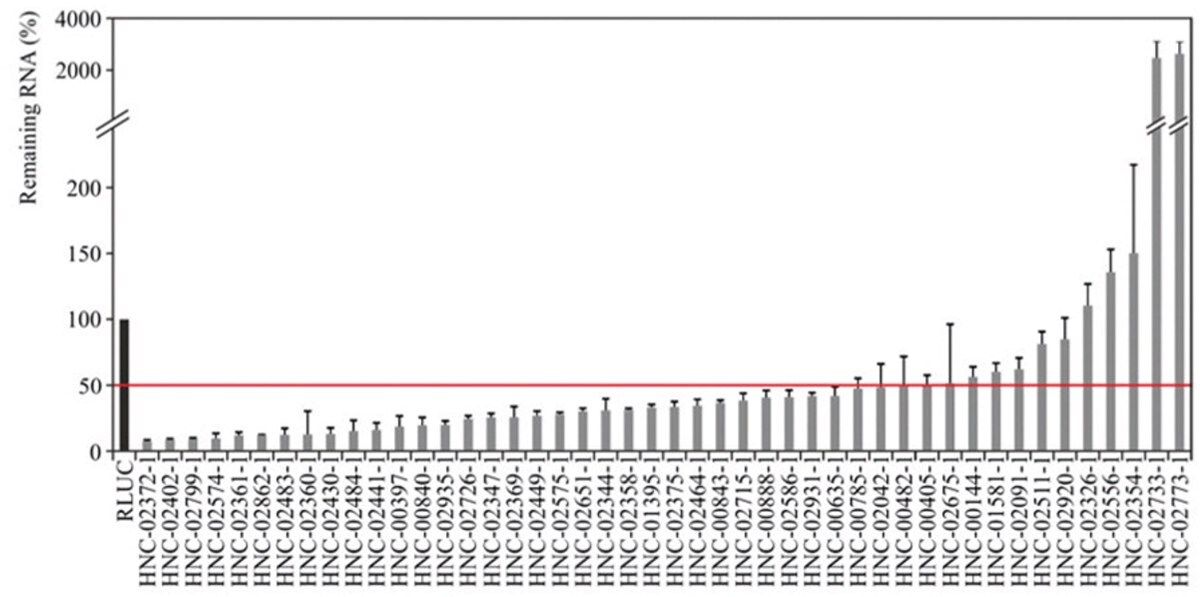
Abbildung 4.qPCR-Daten zur Validierung des Knockdowns von lncRNA in HeLa-Zellen 24 Stunden nach der Transfektion mit der entsprechenden esiRNA. Die Kontrollen wurden mit esiRNA gegen RLUC transfiziert und in der Normalisierung eingesetzt.
Produktherstellung
Biologisch hergestellte esiRNA (Abbildung 5) wird durch Spaltung langer dsRNA (doppelsträngiger RNA) mit der Endoribonuklease E. coli RNase III hergestellt.

Abbildung 5.Überblick über die Herstellung von esiRNA für einzelne Gene. A) DEQOR wird verwendet, um die mRNA-Zielregion auszuwählen, welche die größte Anzahl hochwirksamer siRNAs produziert, alle bekannten Transkriptvarianten abdeckt und Off-Target-Effekte minimiert. B) Die Zielregion wird aus dem cDNA-Klon extrahiert und mittels PCR amplifiziert (beide Primer werden zur Einführung von T7-Promotern verwendet). C) Das PCR-Produkt wird in vitro mit Hilfe von RNA-Polymerasen transkribiert. D) Die hybridisierte und lange, doppelsträngige RNA (dsRNA) wird mit RNase III verdaut und durch Aufreinigung werden die DNA-Template, verbleibende NTP und unvollständig verdaute dsRNA entfernt. Schließlich ist die esiRNA als ein Pool Hunderter siRNA mit einer durchschnittlichen Duplexlänge von 21 bp fertiggestellt.
esiRNA-Garantie
Durch MISSION® esiRNA wird die Ziel-mRNA in kultivierten Zellen um 70 % reduziert, wenn sie mit einer Konzentration von ≥ 30 nM transfiziert wird. Wenn die esiRNA das Zielgen nicht zu 70 % unterbindet (Knockdown), stellen wir kostenfrei eine weitere esiRNA für dieses Gen zur Verfügung. Wenn es keine esiRNA mehr für dieses Gen gibt, erstatten wir den Kaufpreis zurück.
Für die Garantie ist der Erhalt geeigneter Daten zur Transfektionseffizienz erforderlich. Geeignete unterstützende Daten für die Transfektionseffizienz wären qPCR-Daten, durch welche die Ziel-mRNA-Niveaus einer validierten MISSION® esiRNA, die mit ≥ 30 nM transfiziert wurde, mit einer geeigneten Negativkontrolle (z. B. mock-Transfektion, RLUC-, FLUC- oder eGFP-esiRNA) verglichen werden, die einen Knockdown der Ziel-mRNA von 70 % belegen.
Aufgrund der Variabilität von Antikörpern und Protein-Halbwertszeiten können wir keine Daten aus proteinbasierten Nachweisverfahren akzeptieren.
Häufig gestellte Fragen
Weitere Informationen über esiRNA sind im FAQ-Bereich mit häufig gestellten Fragen aufgeführt.
Video-Tutorial
Screening in Säugerzellen mit esiRNA.
Falls Sie weitere Hilfe benötigen, wenden Sie sich bitte unter [email protected] an unseren technischen Dienst.
Erfahren Sie mehr über RNA-Interferenz: von Einzelgenstudien bis zu Ganzgenomscreens – Dr. Julia Krüger, Senior Scientist bei Eupheria Biotech GmbH
Zitationen auswählen
Einzelgen-esiRNA
Ganzgenom- und Teilbibliotheken-Screens
lncRNA esiRNA
Um weiterzulesen, melden Sie sich bitte an oder erstellen ein Konto.
Sie haben kein Konto?