07-355
Anticorps anti-acétyl-histone H3 (Lys23)
serum, Upstate®
Synonyme(s) :
H3K23Ac, Histone H3 (acetyl K23)
About This Item
Produits recommandés
Source biologique
rabbit
Niveau de qualité
Forme d'anticorps
serum
Type de produit anticorps
primary antibodies
Clone
polyclonal
Espèces réactives
Saccharomyces cerevisiae, human, vertebrates
Fabricant/nom de marque
Upstate®
Technique(s)
ChIP: suitable
dot blot: suitable
western blot: suitable
Numéro d'accès NCBI
Numéro d'accès UniProt
Conditions d'expédition
wet ice
Modification post-traductionnelle de la cible
acetylation (Lys23)
Informations sur le gène
human ... H3C1(8350)
Description générale
La queue N-terminale de l'histone H3 dépasse de la structure globulaire centrale du nucléosome et peut subir différentes modifications épigénétiques qui affectent les processus cellulaires. Ces modifications comprennent la fixation covalente de groupements méthyle ou acétyle sur les acides aminés lysine et arginine, et la phosphorylation de la sérine en thréonine.
Spécificité
Immunogène
Application
Épigénétique et fonction nucléaire
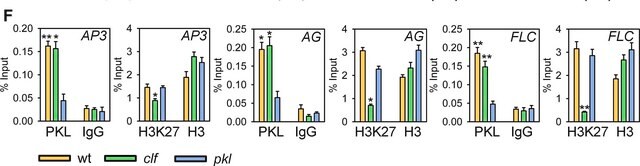
données d'un lot représentatif.
De la chromatine préparée à partir de cellules HeLa (1 × 10e6 équivalents cellulaires par IP) et passée aux ultrasons a été soumise à une immunoprécipitation de la chromatine avec soit 2 µl de sérum de lapin normal, soit 2 µl d'anticorps anti-acétyl-histone H3 (Lys23), et le kit Magna ChIP A (réf. 17-610). Le succès de l'immunoprécipitation de fragments d'ADN associés à l'acétyl-histone H3 (Lys23) a été vérifié par qPCR en utilisant des amorces témoins de la région promotrice du gène GAPDH comme locus positif, et des amorces MyoD comme locus négatif. Les données sont exprimées en pourcentage de chaque échantillon immunoprécipité par rapport à la chromatine de départ ("input") pour chacun des amplicons et des échantillons de ChIP indiqués.
Se référer au protocole du kit EZ-Magna ChIP A (réf. 17-408) ou du kit EZ-ChIP (réf. 17-371) pour une description détaillée du protocole expérimental.
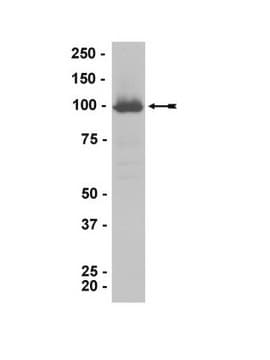
Analyse par western blotting :
données d'un lot représentatif.
Des extraits acides de cellules HeLa traitées au butyrate de sodium (Lane 1, réf. 17-305) et de l'histone H3 recombinante (Lane 2, réf. 14-494) ont été testés avec l'anticorps anti-acétyl-histone H3 (Lys23) (dilution au 1/100 000e).
La flèche indique l'acétyl-histone H3 (env. 17 kDa)
Analyse par dot blot :
données d'un lot représentatif.
40 ng et 4 ng de peptides d'histone portant diverses modifications (voir Tableau 1) ont été transférés sur une membrane en PVDF et testés avec l'anticorps anti-acétyl-histone H3 (Lys23) (dilution au 1/2 000e). Les protéines ont été visualisées à l'aide d'une IgG de chèvre anti-lapin conjuguée à de la HRP et d'un système de détection par chimiluminescence. L'image présentée correspond à une exposition de 60 secondes.
Histones
Qualité
Description de la cible
Forme physique
Stockage et stabilité
Remarque sur l'analyse
Extraits acides de cellules HeLa traitées au butyrate de sodium
Informations légales
Clause de non-responsabilité
Vous ne trouvez pas le bon produit ?
Essayez notre Outil de sélection de produits.
Code de la classe de stockage
10 - Combustible liquids
Classe de danger pour l'eau (WGK)
WGK 1
Certificats d'analyse (COA)
Recherchez un Certificats d'analyse (COA) en saisissant le numéro de lot du produit. Les numéros de lot figurent sur l'étiquette du produit après les mots "Lot" ou "Batch".
Déjà en possession de ce produit ?
Retrouvez la documentation relative aux produits que vous avez récemment achetés dans la Bibliothèque de documents.
Notre équipe de scientifiques dispose d'une expérience dans tous les secteurs de la recherche, notamment en sciences de la vie, science des matériaux, synthèse chimique, chromatographie, analyse et dans de nombreux autres domaines..
Contacter notre Service technique