Resolução de problemas de RT-PCR / RT-qPCR
Nesta página
- Desenvolvimento de um protocolo de resolução de problemas
- Otimização do design de oligonucleotídeo
- Otimização do ensaio de PCR
- Modelo de ensaio de RT-PCR e qPCR
- Qualidade do molde de RNA ou DNA
- Programa de ensaio PCR
- Mau funcionamento do termociclador
- Exemplos de resolução de problemas: Ferramentas de diagnóstico
- Curvas de dissociação/fusão
- Estudos de caso de resolução de problemas de RT-PCR
- Resumo - Lista de verificação de resolução de problemas de PCR
Desenvolvimento de um protocolo de resolução de problemas de PCR ou RT-PCR
Possíveis fontes de erro e/ou problemas de erro do operador
Há muitas possibilidades de erro do operador. As fontes desses erros geralmente não são identificadas. A primeira etapa de qualquer procedimento de resolução de problemas é verificar o protocolo e repetir o experimento. É importante verificar o protocolo (consulte o Apêndice A, Protocolos, deste guia) e pedir a um biólogo molecular experiente que revise o plano experimental. A história de advertência do pós-doutorando que executou várias PCRs com falhas antes de perceber que os dNTPs estavam faltando na mistura-mestre de PCR é um lembrete de que até mesmo os melhores cientistas, que trabalham demais, são vulneráveis a erros simples.
Mistura-mestre
Erros ou problemas com a mistura-mestre dos componentes da reação podem ser a fonte de uma falha catastrófica de amplificação em todas as amostras e controles positivos. Antes de repetir o experimento, verifique todos os componentes e suas concentrações. Se um novo lote de reagente estiver sendo usado, é uma precaução útil comparar o novo com o antigo antes de iniciar uma grande série de experimentos.
Ao trocar os produtos da mistura-mestre, é fundamental reconhecer que alguns ensaios são particularmente sensíveis às combinações de composição do tampão/temperatura de hibridização (Ta)/concentração do primer. A alteração de qualquer um desses itens pode resultar em um desempenho diferente. Portanto, verifique todos os ensaios em misturas-mestre selecionadas e em todos os instrumentos desejados antes de fazer alterações radicais. Também é essencial revisar as instruções fornecidas com cada mistura-mestre, pois elas especificam as condições recomendadas que são otimizadas para a enzima em questão, o mecanismo Hot Start e os componentes do tampão.
É uma boa prática de laboratório garantir que seja preparada uma mistura-mestre de reação suficiente para todas as amostras que serão executadas juntas. Certifique-se de que todos os componentes estejam cuidadosamente descongelados e bem misturados e que a mistura-mestre do experimento esteja muito bem misturada antes de aliquotar as amostras. Isso é particularmente relevante para alguns dos tampões 2×, como o KiCqStart® , que são mais viscosos do que os tampões normais de PCR.
Otimização de oligonucleotídeos
Os oligonucleotídeos podem causar problemas se tiverem uma sequência incorreta ou forem mal projetados, se forem executados em uma concentração abaixo do ideal, em uma Ta abaixo do ideal ou se forem marcados ou atenuados de forma inadequada (para sondas). Um ensaio executado em condições abaixo do ideal para o oligo, ou usando um design inadequado, pode produzir alguns dados, mas isso pode não refletir a biologia genuína em consideração. Ao receber um oligonucleotídeo liofilizado, é fundamental:
- Verificar a sequência
- Garantir que todo o DNA seja novamente suspendido antes do uso
- Confirmar se a resolução tem a concentração esperada
Suspender novamente os oligonucleotídeos aquecendo-os a 90 °C por 5 minutos e misturando-os bem. Ciclos repetidos de congelamento e descongelamento também podem afetar o desempenho dos oligonucleotídeos e, portanto, todos os oligonucleotídeos em uma concentração de estoque (geralmente 100 μM) devem ser aliquotados e armazenados a -20 °C ou -80 °C a longo prazo.
Durante a fase de resolução de problemas, é fundamental verificar se a sequência correta foi solicitada, retornando à sequência-alvo e confirmando que as sequências de oligonucleotídeos estão de fato presentes. Verifique se a qualidade do oligonucleotídeo estava correta entrando em contato com o fornecedor do oligonucleotídeo. Meça a concentração de trabalho do oligonucleotídeo e inspecione visualmente as moléculas fluorescentes para confirmar que elas estão marcadas. Teste os primers dos ensaios de sonda em uma mistura de qPCR SYBR® Green I para verificar a amplificação. Considere otimizar as concentrações de primers ou Ta (consulte Otimização e validação de ensaios). Ao usar uma sonda pela primeira vez, colete dados fluorescentes para o maior número possível de comprimentos de onda, de modo que qualquer possível vazamento de sinal entre os canais seja observado e erros na marcação possam ser detectados.
Otimização inadequada de PCR
O efeito da otimização do ensaio foi descrito e demonstrado em Otimização e validação de ensaios. Quando um ensaio falha ou tem desempenho subótimo, mas não há erros no design ou nos procedimentos operacionais, ele pode se beneficiar da otimização das condições experimentais. Ao solucionar problemas, teste os primers em concentrações finais de 100 nM, 500 nM e 900 nM e/ou Ta entre 55 °C e 70 °C (usando um gradiente de temperatura) para identificar se o ensaio melhorará com otimização adicional.
Modelo de ensaio de RT-PCR e qPCR
O modelo do ensaio foi descrito em Modelo de ensaio PCR/qPCR/dPCR. Ao solucionar problemas de um ensaio, certifique-se de que o design tenha sido verificado. Confirme se o primer de PCR/qPCR e a posição do amplicon estão de acordo com o protocolo de primer de RT. Por exemplo, certifique-se de que os ensaios aplicados ao cDNA que foi preparado após a preparação do oligo-dT estejam situados na direção do ponto 3' da transcrição. Certifique-se de que as informações da sequência sejam confiáveis e que as variantes de emenda e os SNPs apropriados tenham sido considerados.

Figura 11.1A.O ensaio tem um perfil de gráfico de amplificação incomum com um desvio pronunciado da linha de base.

Figura 11.1B.A sequência da sonda que foi incluída no ensaio foi inserida no software de previsão de dobragem mfold. Está claro que a sonda pode adotar uma estrutura dobrada estável em resolução e é provável que isso resulte no problema observado.
Qualidade do molde de RNA ou DNA
O efeito da qualidade do modelo no desempenho do ensaio foi descrito em Purificação de amostras e avaliação da qualidade. A qualidade do modelo abrange a consideração da quantidade, da integridade e da presença de inibidores. É fundamental garantir que a qualidade do RNA seja compatível com o protocolo de preparação de RT mais adequado (consulte Transcrição reversa) e usar o modelo da melhor qualidade possível. Da mesma forma, a quantidade de RNA que é adicionada às reações de RT deve estar dentro do escopo do protocolo e, em muitos casos, deve ser a mesma para todas as reações. O ReadyScript® é uma exceção notável a essa diretriz porque a adoção desse reagente e do protocolo produz uma concentração linear de cDNA que é proporcional à quantidade de RNA de entrada. Ao solucionar problemas de uma amostra que esteja produzindo um Cq maior do que o esperado, execute o ensaio SPUD ou dilua a amostra em uma série de diluições de 1:5 ou 1:10 e repita o ensaio (Figura 11.2) para identificar amostras que contenham inibidores.


Figura 11,2.Amplificação de uma diluição em série de 10 vezes de um molde de DNA. As réplicas são precisas, mas o ΔCq é inconsistente, diminuindo com o aumento das diluições. Os dados também mostram um sinal positivo no controle sem modelo (NTC), indicando contaminação ou formação de dímero de primer, e que as diluições para menos de 105 cópias têm dados idênticos aos do NTC.
A quantidade de modelos também é uma consideração importante. A inclusão de um modelo muito grande ou muito pequeno na PCR resultará em reações fracassadas e gráficos de amplificação de qPCR que parecem anormais. A Figura 11.3A mostra uma reação contendo uma diluição em série de 10 vezes do modelo de oligonucleotídeo artificial. As diluições mais baixas são muito concentradas para que a reação seja eficiente ou para que o instrumento processe efetivamente os dados da linha de base (Figura 11.3B), resultando em gráficos de amplificação anormais e dados não confiáveis.

Figura 11.3. A)Amplificação de uma diluição em série de 10 vezes de um modelo artificial com primers específicos e uma sonda marcada com FAM. O Cq é muito baixo para as amostras concentradas, os gráficos de amplificação não são espaçados regularmente e são anormais. B) Mostra os dados brutos para esses gráficos de amplificação. As reações que contêm a maior concentração de alvo também apresentam uma fluorescência de fundo significativamente maior e um rendimento mínimo de fluorescência durante a reação.
Programa de ensaio PCR
As condições de ciclos da PCR devem ser adequadas tanto para o experimento quanto para os reagentes (por exemplo, consulte Mistura-mestre). Não é aconselhável aceitar as configurações padrão do instrumento sem verificação.
Mau funcionamento do termociclador
As falhas do instrumento podem ter um início insidioso e, portanto, podem ser difíceis de diagnosticar. Para evitar custos de reparo elevados, certifique-se de que todos os operadores de instrumentos sejam totalmente treinados e inicialmente supervisionados. Algumas falhas de instrumentos causam falhas catastróficas, resultando na ausência de amplificação ou de dados fluorescentes, enquanto outras distorcem os dados ou tratam as amostras de maneira não uniforme, criando diferenças artificiais entre amostras biológicas idênticas. O uso de amostras de controle com ensaios de controle é inestimável para a resolução de problemas. Quando se suspeita de uma falha no instrumento, um ensaio confiável e otimizado deve ser executado em todos os poços. Essa verificação de uniformidade revelará problemas específicos de regiões do instrumento, bem como problemas separados do ensaio e do instrumento.
Exemplos de resolução de problemas de PCR que demonstram o uso das ferramentas de diagnóstico
Depois de executar uma PCR bem planejada, há várias ferramentas de diagnóstico disponíveis para a resolução de problemas:
- Amostras de controle e ensaios
- Gel de ponto final/Reagente de corante SYBR Green I
- Gráficos de amplificação (verifique as réplicas e o perfil do gráfico de amplificação)
- Curvas padrão (gradiente e R2)/série de diluição
- Gráficos de fusão/dissociação (corante SYBR Green I, Molecular Beacons (“faróis” moleculares), sondas Scorpions®)
- Visualizações de dados brutos/multicomponentes
Amostras/Reações de controle
O uso de controles é altamente recomendado. É quase impossível resolver um ensaio com falha sem informações de um conjunto apropriado de controles.

Figura 11.4. A)O modelo não diluído não consegue amplificar, enquanto as diluições mostram uma maior eficiência de amplificação. B) A adição de 0,3% de BSA à mistura de qPCR favorece a amplificação a partir do modelo não diluído.
As investigações sobre um ensaio completamente com falha podem ser difíceis porque há poucas informações para trabalhar na resolução de problemas. Como muitas falhas de ensaio são resultado de algum erro catastrófico, a primeira verificação deve ser a configuração do experimento e, em seguida, repetir a PCR. Se isso falhar, o processo de resolução de problemas dependerá das informações sobre cada componente do experimento (Figura 11-5).

Figura 11.5.O processo básico de resolução de problemas para PCR.
Quando um experimento de qPCR falha completamente, a primeira etapa é verificar o design do ensaio, as sequências de oligonucleotídeos e os dados de CQ do fabricante dos oligo. Embora o ensaio possa ter falhado, os dados de qPCR multicomponentes/brutos podem ser usados para fornecer mais informações. A Figura 11.6A mostra o gráfico de dados brutos para dois ensaios contendo uma sonda marcada com 6-FAM™ ou HEX™ (VIC®). Embora ambos os ensaios mostrem amplificação, o sinal HEX é aproximadamente a metade do sinal FAM. Como esse é um corante inerentemente mais fraco, essa é uma observação normal. A análise do gel de agarose (Figura 11.6B) mostra que ambas as reações geram uma concentração semelhante de produtos, apoiando a observação de que os valores de Cq da qPCR são semelhantes.

Figura 11.6. A)Os gráficos de dados brutos de um ensaio duplex contendo uma sonda FAM e uma sonda marcada com HEX. A sonda FAM produz naturalmente maior fluorescência. B) O gel de agarose mostra que quantidades iguais de produto foram produzidas em cada reação e confirma a observação do qPCR Cq.
O exame dos dados brutos é uma verificação útil para verificar se a sonda está rotulada corretamente e se foi adicionada à reação. A Figura 11.7 mostra os dados brutos da amplificação de três alvos em um experimento triplex. As sondas específicas para cada alvo são marcadas com FAM, HEX e TAMRA. As sondas HEX e TAMRA mostram um fundo baixo e uma amplificação eficiente, mas o sinal FAM é consistentemente alto durante todo o experimento e não há evidência de amplificação. Isso é consistente com uma concentração muito alta da sonda na reação ou com uma falha na sonda, de modo que não há extinção inicial do sinal. Nesses casos, a concentração da sonda e o modelo do ensaio devem ser verificados, garantindo que a sonda tenha um rótulo e um redutor compatíveis e, se necessário, que uma nova sonda seja testada.
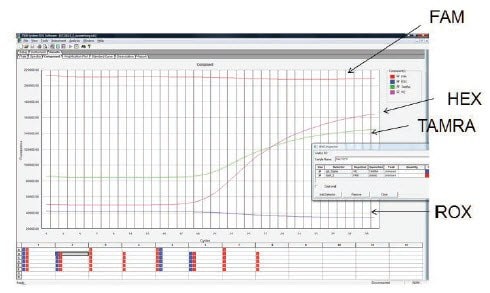
Figura 11.7.Uma reação triplex foi usada para detectar três alvos usando sondas marcadas com FAM, HEX e TAMRA. As sondas HEX e TAMRA produziram amplificação dos alvos, mas a sonda FAM não apresentou amplificação. O exame dos dados brutos revelou que a fluorescência de fundo era excepcionalmente alta e nenhuma diferença foi observada durante a reação. Isso é consistente com uma concentração muito alta da sonda na reação ou com uma sonda defeituosa com extinção inadequada.
Se o experimento original se baseou na detecção de sonda, o ensaio deve ser repetido usando reagentes SYBR Green I, incluindo um controle positivo e um negativo (mas não amostras preciosas). Como alternativa, os produtos de uma reação com falha podem ser verificados em um gel de agarose corado com brometo de etídio. A adoção da abordagem SYBR Green I para a repetição do experimento é preferível porque evita o risco de contaminação e fornece um experimento repetido para verificar a falha inicial. Se o experimento SYBR Green I fornecer dados, é possível que a falha da sonda original tenha sido causada por um erro técnico ou por uma falha na sonda. Para diferenciar entre um erro experimental ou uma falha na sonda, repita o experimento com a sonda; se a reação falhar novamente, substitua a sonda. Essa abordagem pode ser adotada para investigar reações que estejam produzindo dados ruins. No exemplo mostrado na Figura 11.8, a reação da sonda estava abaixo do ideal e, quando comparada com a reação executada usando SYBR Green I, pode-se ver que o sinal da sonda não reflete o experimento. Nesses casos, o modelo do ensaio deve ser verificado e uma nova sonda deve ser testada.

Figura 11.8.Reações idênticas foram executadas com uma sonda qPCR ou corante SYBR Green I (conforme indicado). A reação SYBR Green I foi aproximadamente onze ciclos mais sensível e produziu uma fluorescência de ponto final muito maior. Isso indica uma falha na sonda ou um problema com o design da sonda (dados gentilmente fornecidos pelo Prof. Stephen Bustin, Reino Unido).
Validação da marcação da sonda
Os dados brutos ou o gráfico de multicomponentes são uma ferramenta de diagnóstico útil para investigar se a concentração adequada da sonda foi incluída na reação e se a sonda foi adequadamente marcada e extinta. A Figura 11.9 mostra o gráfico multicomponente para uma reação contendo três sondas. Os dois primeiros geram gráficos de amplificação e a fluorescência de fundo é evidente. Não há dados da terceira sonda e um exame dos dados brutos revela que a fluorescência de fundo é equivalente ao controle em branco da água, que não contém nenhuma sonda. Portanto, esses dados são o resultado da ausência de fluorescência na reação. Isso pode ser devido a um erro durante a configuração em que a sonda não foi incluída ou em que a sonda não foi etiquetada.

Figura 11.9.Três genes foram detectados na mesma amostra modelo. Duas reações resultaram em amplificação (1 e 2), mas a terceira foi negativa. Um exame da visualização de multicomponentes revela que a fluorescência de fundo para a terceira reação é equivalente ao controle de água, indicando uma ausência de sinal.
Uma verificação adicional da marcação da sonda pode ser realizada usando uma digestão com DNase I. Isso deve ser feito com extremo cuidado para garantir que os estoques de sondas e primers não sejam contaminados com enzimas, o que levaria a resultados catastróficos. Uma alíquota de uma sonda com falha (Figura 11.10A) equivalente à incluída em uma reação, por exemplo, 300 nM, é incubada com e sem DNase I. Isso pode ser feito em tempo real (Figura 11.10B), de modo que o rendimento fluorescente seja medido em relação ao tempo ou, alternativamente, a leitura do ponto inicial e final (após 10 minutos) fornece informações suficientes. Ao realizar esse teste, é importante comparar os dados com uma sonda que esteja funcionando bem e que tenha o mesmo marcador fluorescente e redutor (Figura 11.10B).

Figura 11.10. A)Dois modelos foram detectados usando sondas diferentes, ambas marcadas com FAM. Enquanto a detecção usando uma sonda resultou em um sinal fluorescente alto, a segunda foi muito mais fraca. B) Um controle e uma sonda de teste (300 nM) foram incubados a 37 °C em um instrumento em tempo real no tampão DNase I na presença ou ausência da enzima DNase I. A liberação fluorescente da sonda 1 foi aproximadamente o dobro da sonda 2, demonstrando que a marcação da sonda 2 era inadequada.
Gráficos de amplificação
A estrutura dos gráficos de amplificação e a reprodutibilidade das réplicas técnicas fornecem uma grande quantidade de informações sobre a qualidade do ensaio de qPCR e também podem fornecer as primeiras indicações de alerta de que nem tudo está como deveria estar. Os gráficos de amplificação na Figura 11.11A não são típicos, são muito ruidosos e seriam difíceis de interpretar com precisão. Um exame mais aprofundado dos valores de fluorescência dR revela que o rendimento fluorescente do ponto final é de apenas 400 unidades, indicando que a reação é inadequada, mas os gráficos de amplificação foram gerados pelo software do instrumento e dimensionados automaticamente. Da mesma forma, os dados da Figura 11.11B têm uma cauda de raposa pronunciada (curva decrescente) no início do perfil, antes de aumentar novamente após uma seção de linha de base. A aparência da cauda de raposa é consistente em duas reações, mas uma reação tem um ponto final muito mais baixo (Figura 11.11C), resultando em uma cauda de raposa amplificada e relativa.
Figura 11.11. A) Gráficos de amplificação com ruído devido ao escalonamento automático pelo software do instrumento de dados ruins com baixa fluorescência. B) As reações que produzem um ponto final baixo dR têm uma cauda de raposa inicial pronunciada. C) O rabo de raposa é visto como um efeito normal quando comparado ao ensaio de alta qualidade.
Da mesma forma, os gráficos de amplificação na Figura 11.12A são claramente anormais e não podem ser usados da forma como são apresentados. Um gráfico de amplificação que fica abaixo de zero dR (Figura 11.12A) é uma indicação clássica de que foram aplicadas configurações de linha de base inadequadas. O exame dos dados brutos dessa reação (Figura 11.12B) mostra que os gráficos de amplificação reais têm um perfil normal, confirmando que os dados analisados são o resultado de um problema de software do instrumento. A linha de base apropriada pode ser deduzida dos dados brutos e aplicada no software. Nesse caso, os ciclos 6 a 16 representam a fase inicial linear e de linha de base da reação e, quando aplicados, resultam em gráficos de amplificação normais (Figura 11.12C).

Figura 11.12. A)Os gráficos de amplificação eram claramente anormais, com uma seção do perfil caindo abaixo da linha de base. B) O exame do gráfico de dados brutos revela que os dados da reação estão de acordo com o esperado. C) A configuração da linha de base do instrumento de acordo com os ciclos apropriados restaura o perfil normal dos dados dos gráficos de amplificação analisados.
O perfil do gráfico de amplificação também pode ser interpretado para fornecer informações sobre a qualidade do ensaio e a otimização. A Figura 11.13 mostra a tentativa de amplificação de uma diluição serial de 10 vezes do modelo com cada concentração executada em qPCR duplicada. A reprodutibilidade entre réplicas é ruim, a diferença de ciclo (ΔCq) entre os dados não é constante e não é de 3,323 ciclos, como esperado para uma diluição em série de 10 vezes. O exame dos gráficos de amplificação, considerando que se trata de uma curva padrão, revela que o ensaio está abaixo do padrão e não pode ser usado para análise. Os motivos precisariam de uma investigação mais aprofundada, mas poderiam ser o resultado de: modelo de ensaio inadequado (consulte Modelo de ensaio PCR/qPCR/dPCR), condições de ensaio abaixo do ideal (consulte Otimização e validação do ensaio) ou pipetagem inadequada (ensaio repetido).

Figura 11.13.Uma amostra de cDNA foi diluída por meio de uma diluição em série de 10 vezes e o modelo específico foi detectado usando qPCR duplicado para cada diluição. As réplicas são ruins, indicando um problema com a pipetagem ou com a otimização do ensaio.

Figura 11.14.Durante uma qPCR padrão, os dados aumentam repentinamente com um perfil não típico.

Figura 11.15.No caso de gráficos de fluorescência decrescentes ou com gancho, a possível causa pode ser o fato de a fita complementar competir com o primer e/ou a sonda para o hibridização do modelo. Ignorar, desde que o Ct não seja afetado
Curvas de dissociação/fusão
A análise de dissociação ou curva de fusão é executada após a qPCR e é uma ferramenta de análise usada em conjunto com corantes de ligação ao DNA (como SYBR Green I) ou sondas não degradantes, como Molecular Beacons (“faróis” moleculares) ou sondas Scorpions®, para verificar se um único produto foi amplificado. Após a amplificação da PCR, o amplicon resultante é incubado em temperaturas mais altas e as alterações no sinal fluorescente são detectadas conforme as transições do DNA entre os estados de fita dupla e fita simples. Quando a reação contém um único amplicon, ele se funde uniformemente e o gráfico de dF/dT (taxa de alteração da fluorescência em relação à temperatura) mostra um único pico. O exame da curva de fusão é particularmente eficaz quando combinado com dados de controles. A Figura 11.16A mostra o perfil de fusão pós qPCR para uma série de amostras de teste experimentais, o controle positivo e o controle sem modelo. O perfil de fusão das amostras de teste é idêntico ao do controle positivo e cada uma delas apresenta um único pico para a dF/dT. O perfil de fusão para o controle sem modelo tem um perfil mais amplo e uma Tm mais baixa. Essas observações são consistentes com a presença de dímeros de primers aparentes no controle negativo. Isso é confirmado com o uso de um gel de agarose corado com brometo de etídio (Figura 11.16B), que também mostra que os dímeros de primer se tornam aparentes quando o modelo está presente em baixa concentração. Isso causa uma superestimativa do alvo quando detectado em amostras de baixa concentração de alvo. Portanto, o ensaio deve ser otimizado ou reprojetado. Em contraste, a Figura 11.16C mostra que o perfil de fusão do produto no controle sem modelo é idêntico ao perfil de fusão do controle positivo e da amostra de teste. Essa é uma indicação clara de contaminação do controle sem modelo com modelo durante a configuração do experimento. O exemplo final demonstra o reconhecimento da amplificação do alvo do gDNA que está presente em uma amostra de cDNA (Figura 11.16D). O amplicon derivado do gDNA é mais longo e, portanto, tem uma
Tm mais alta do que a do cDNA.

Figura 11.16A.Um controle positivo, a reação de teste e o NTC foram amplificados e, em seguida, submetidos à análise de fusão pós-PCR. Há um produto evidente no NTC que derrete em uma temperatura mais baixa e com um pico de fusão mais amplo, consistente com a formação do dímero do primer.

Figura 11.16B.Os dímeros de primers são evidentes em uma resolução de gel dessas amostras (juntamente com outras), com a formação de dímeros de primers sendo inversamente proporcional à concentração do modelo de entrada.

Figura 11.16C.Um exemplo de aplicação da análise da curva de fusão para identificar a contaminação da reação no NTC.
Figura 11.16D.Identificação de um amplicon maior resultante da PCR do gDNA.

Figura 11.17. A)A) perfil de fusão e B) análise em gel de agarose de uma reação SYBR green I. Embora o perfil de fusão sugira produtos de Tm variável, a imagem do gel indica que um único amplicon está presente. Isso é indicativo de uma sequência de amplicon que contém regiões ricas em AT ou GC ou um elemento repetitivo que resulta em fusão irregular
Diluição em série do modelo/curvas padrão
Independentemente de o design experimental incluir a exigência de uma curva padrão para eventual quantificação, a detecção de uma diluição em série do modelo adequado é uma abordagem eficiente para a validação do ensaio e a resolução de problemas. A detecção de uma diluição em série permite que o intervalo dinâmico linear experimental do ensaio seja definido. A Figura 11.18A mostra uma curva padrão com pontos de dados de baixa concentração que não se ajustam ao perfil linear. O motivo mais comum para esse padrão de dados é a formação de dímeros de primers em amostras de baixa concentração (como mostrado na Figura 11.18B). Essa é a curva padrão gerada a partir dos dados mostrados na Figura 11.2. A Figura 11.18C mostra uma curva padrão com amostras altamente concentradas que caem fora da faixa linear. Os motivos mais comuns para isso são a inibição do modelo da reação ou o fato de as configurações da linha de base serem inadequadas.

Figura 11.18. A) Os pontos de dados relativos às concentrações mais baixas do alvo não se encontram na curva padrão. B) Isso é típico de uma reação que resulta em dímeros de primers, conforme ilustrado. Nesse caso, não há aumento observado no Cq para as amostras em baixa concentração.

Figura 11.18C.As amostras com altas concentrações de modelo não se encontram na curva padrão. Isso é típico de reações que são inibidas pela concentração do modelo ou devido à configuração incorreta da linha de base.
A curva padrão também é usada para medir a eficiência da reação em toda a faixa dinâmica das diluições. Deve-se tomar cuidado para garantir que todos os pontos usados nos cálculos de eficiência estejam sobre a linha. As reações devem ser tão próximas de 100% de eficiência quanto possível, e aquelas com eficiência aparentemente alta (>110%) ou baixa (<85%) devem ser investigadas mais a fundo.

Figura 11.19A.Um ácido nucleico modelo foi diluído em uma série de 10 vezes. Os gráficos de amplificação têm um gradiente anormalmente raso e o ΔCq é de 4 ciclos, em vez dos 3,3 esperados.

Figura 11.19B.O gradiente de um gráfico de curva padrão de Cq em relação à quantidade é usado para calcular a eficiência da reação.

Figura 11.20A.Um ácido nucleico modelo foi diluído em uma série de 10 vezes. O ΔCq entre os gráficos de amplificação é de 1,5 ciclos em vez de 3,3.

Figura 11.20B.O gradiente de um gráfico de curva padrão de Cq em relação à quantidade é usado para calcular a eficiência da reação, que está próxima de 140%.

Figura 11.20C.Um exame do perfil da curva de fusão revela que as amostras de menor concentração (traços amarelo e azul) também contêm sinal de dímeros de primers amplificados (pico em Tm inferior).
Estudos de caso de resolução de problemas de RT-PCR
Um ensaio de sonda com falha
Um ensaio baseado em sonda foi projetado para detectar EIFB1 em amostras de cDNA humano, mas não mostra amplificação. As reações iniciais foram executadas em um instrumento ABi StepOne usando reagentes compatíveis. Foi feita uma tentativa de otimizar os primers usando uma faixa de concentrações de 200 nM a 900 nM (Figura 11.21), mas não houve melhora. O design do ensaio foi verificado e considerado apropriado para o alvo e, in silico, foi previsto que se tratava de um ensaio de alta qualidade. Novos primers foram sintetizados e executados juntamente com uma alíquota da síntese original usando um operador diferente, reagentes SYBR Green I (portanto, reagentes diferentes) e instrumento (um Eppendorf Realplex) (Figura 11.22). Ao adotar essa abordagem, tivemos em mente que o objetivo principal era resolver o problema, enquanto o objetivo secundário era explicar a falha. Essa reação produziu amplificação equivalente de ambos os lotes de primers. Nesse estágio, parecia que o problema da reação estava na sonda e, portanto, uma nova sonda foi sintetizada e os dois lotes foram comparados pelo segundo operador no instrumento Realplex usando reagentes LuminoCt® (reagentes diferentes dos originalmente testados) (Figura 11.23). Ambas as sondas produziram dados de amplificação, sendo que a nova sonda pareceu um pouco melhor do que a original, embora seja importante observar que a sonda original foi enviada entre laboratórios de teste e, portanto, estava em temperatura ambiente, em solução, por vários dias. Nesse estágio, ficou claro que tanto o ensaio original quanto o de substituição funcionaram quando executados pelo segundo operador com reagentes LuminoCt® no instrumento Realplex.
Portanto, os motivos restantes para a falha original foram considerados:
- Operador: o experimento foi repetido várias vezes por um cientista experiente e, portanto, essa foi considerada uma explicação improvável.
- Instrumento: poderia ter alguns problemas, pois alguns outros ensaios estavam falhando.
- Reagentes: explicação mais fácil de testar. Os reagentes LuminoCt® foram comparados com os reagentes existentes usando ambos os lotes de oligonucleotídeo no instrumento ABi StepOne, pelo primeiro operador. A reação falhou com os reagentes originais, mas apresentou boa amplificação com os reagentes LuminoCt® (Figura 11.24).

Figura 11.21.Os primers para EIFB1 foram testados em concentrações entre 200 nM e 900 nM. Não foi observada amplificação em nenhuma condição (oligonucleotídeos e reagentes ABi no ABi StepOne Plus).

Figura 11.22.Dois lotes de primers para EIFB1 foram comparados no reagente SYBR Green I; o lote original com falha e um novo lote. (oligonucleotídeos e reagentes no instrumento Eppendorf Realplex). Ambos os conjuntos de primers suportaram a amplificação.

Figura 11.23.Dois lotes de primers e sondas para EIFB1 foram comparados no reagente LuminoCt®; o lote original com falha e um novo lote. (oligonucleotídeos e reagentes no instrumento Eppendorf Realplex). Ambos os conjuntos de oligonucleotídeos suportaram a amplificação.

Figura 11.24.O ensaio do primer e da sonda EIFB1 foi executado em dois reagentes diferentes (reagente Original ABi ou LuminoCt®). Os dados só foram adquiridos desse ensaio quando executados com os reagentes LuminoCt®.
A eficiência da reação foi incorreta e variável
Um teste foi realizado em uma diluição em série de um oligonucleotídeo artificial, usando um primer padrão e um ensaio de sonda. O ensaio havia sido originalmente desenvolvido e otimizado em um instrumento diferente, mas o efeito de diluição estranho não era esperado quando transferido para um laboratório e instrumento de teste diferentes (Figura 11.25A). Todas as condições do ensaio foram otimizadas novamente para serem específicas do novo laboratório, mas sem alteração dos dados. O operador observou que o efeito era mais pronunciado quando o ensaio era repetido em poucas horas, usando a mesma série de diluição. Como parte do processo de resolução de problemas, o ensaio foi executado em um instrumento diferente por um operador diferente que, novamente, gerou a curva padrão esperada. Isso levou à sugestão de que o problema inicial era devido a erro do operador, falha do instrumento ou alguma variação sutil no procedimento experimental. Como os dois operadores são altamente experientes e o instrumento estava funcionando bem em outros experimentos, a opção de diferenças sutis foi examinada. Uma pista importante foi a observação da variabilidade nos dados da mesma série de diluição após um período de armazenamento das amostras a 4 °C (Figuras 11.25A e 11.25B). Isso levou a um exame dos tubos usados para a série de diluição e a um teste de alternativas. Depois de mudar para tubos de reação Eppendorf de 1,5 ml para a série de diluição, a curva padrão prevista foi gerada (Figura 11.25C), demonstrando que é essencial que a biologia molecular, os materiais plásticos de baixa retenção sejam selecionados para PCR e que esses ensaios são sensíveis a variações sutis no protocolo.

Figura 11.25A.Um modelo de oligonucleotídeo artificial foi diluído 10 vezes e detectado usando um ensaio específico baseado em sonda. Há diferenças inconsistentes entre os gráficos de amplificação.

Figura 11.25B.Um modelo de oligonucleotídeo artificial foi diluído 10 vezes (essas são as diluições detectadas na Figura 11.25A) e deixado a 4 °C por várias horas antes de ser detectado usando um ensaio específico baseado em sonda. Há diferenças inconsistentes entre os gráficos de amplificação, que são exacerbadas pelo tempo entre a diluição e o teste.

Figura 11.25C.Um modelo de oligonucleotídeo artificial foi diluído 10 vezes em tubos de grau e detectado usando um ensaio específico baseado em sonda. Há diferenças consistentes entre os gráficos de amplificação, como esperado.
Resumo - Lista de verificação de resolução de problemas de PCR
- Verifique a qualidade da amostra (material degradado causará resultados errôneos).
- Verifique se o protocolo de RT é compatível com o design (por exemplo, um RT com primer Oligo-dT deve ter um ensaio de qPCR no 3' 1 kb da sequência).
- Verifique o modelo do ensaio.
- Verifique todos os controles.
- Verifique os primers usando corante SYBR green I/execute um gel.
- Verifique se as configurações do software estão corretas (linha de base, detecção de corante, concentrações para padrões).
- Certifique-se de que a concentração de ROX seja aplicável ao instrumento (e não interfira com o multiplex).
- Verifique os níveis de fluorescência de fundo.
- Verifique a marcação da sonda com o ensaio de DNase I ou repita a síntese da sonda.
Para continuar lendo, faça login ou crie uma conta.
Ainda não tem uma conta?