S9192
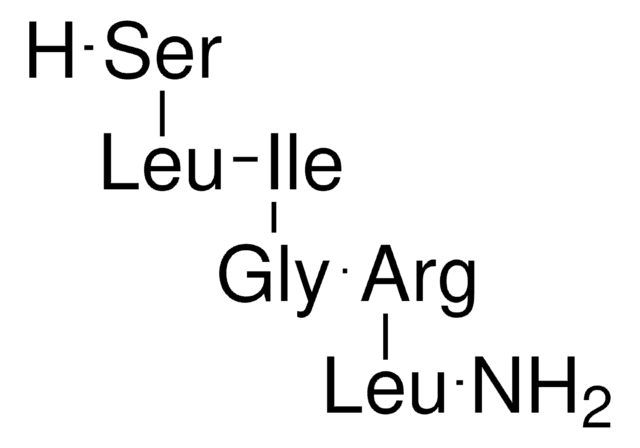
Ser-Leu-Ile-Gly-Lys-Val-amide
≥95% (HPLC), solid
Synonyme(s) :
PAR-2 (1-6) Human, PAR2 activating peptide, PAR2-AP, SLIGKV-NH2
About This Item
Produits recommandés
Niveau de qualité
Pureté
≥95% (HPLC)
Forme
solid
Numéro d'accès UniProt
Température de stockage
−20°C
Chaîne SMILES
CC[C@H](C)[C@H](NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CO)C(=O)NCC(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(N)=O
InChI
1S/C28H54N8O7/c1-7-17(6)23(36-27(42)20(12-15(2)3)34-25(40)18(30)14-37)28(43)32-13-21(38)33-19(10-8-9-11-29)26(41)35-22(16(4)5)24(31)39/h15-20,22-23,37H,7-14,29-30H2,1-6H3,(H2,31,39)(H,32,43)(H,33,38)(H,34,40)(H,35,41)(H,36,42)/t17-,18-,19-,20-,22-,23-/m0/s1
Clé InChI
HOWDUIVVWDUEED-WAUHAFJUSA-N
Informations sur le gène
mouse ... Insrr(23920)
Amino Acid Sequence
Actions biochimiques/physiologiques
Conditionnement
Autres remarques
Code de la classe de stockage
11 - Combustible Solids
Classe de danger pour l'eau (WGK)
WGK 3
Point d'éclair (°F)
Not applicable
Point d'éclair (°C)
Not applicable
Équipement de protection individuelle
Eyeshields, Gloves, type N95 (US)
Certificats d'analyse (COA)
Recherchez un Certificats d'analyse (COA) en saisissant le numéro de lot du produit. Les numéros de lot figurent sur l'étiquette du produit après les mots "Lot" ou "Batch".
Déjà en possession de ce produit ?
Retrouvez la documentation relative aux produits que vous avez récemment achetés dans la Bibliothèque de documents.
Notre équipe de scientifiques dispose d'une expérience dans tous les secteurs de la recherche, notamment en sciences de la vie, science des matériaux, synthèse chimique, chromatographie, analyse et dans de nombreux autres domaines..
Contacter notre Service technique



![[D-Lys3]-GHRP-6 synthetic, lyophilized powder](/deepweb/assets/sigmaaldrich/product/structures/386/044/971c33d0-e117-4aad-bf07-27172c088df1/640/971c33d0-e117-4aad-bf07-27172c088df1.png)



