39441
Nitrate Reagent B
suitable for microbiology
Synonyme(s) :
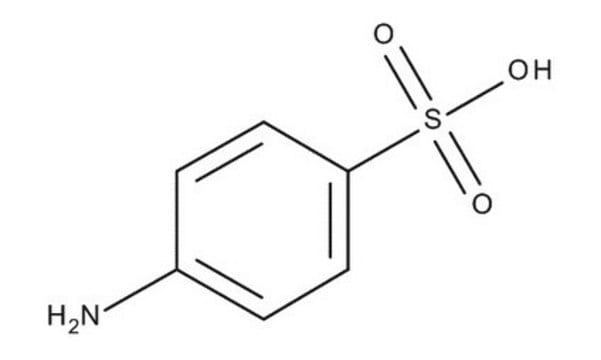
Sulfanilic acid solution, 4-Aminobenzenesulfonic acid
About This Item
Produits recommandés
Durée de conservation
limited shelf life, expiry date on the label
Niveau de qualité
Composition
acetic acid (30%), 1000 mL
sulfanilic acid, 8.0 g
Technique(s)
microbe id | metabolite detection: suitable
Application(s)
agriculture
clinical testing
environmental
food and beverages
pharmaceutical
microbiology
Adéquation
Enterobacter spp.
Escherichia coli
Neisseria spp.
anaerobic bacteria
coliforms
Chaîne SMILES
Nc1ccc(cc1)S(O)(=O)=O
InChI
1S/C6H7NO3S/c7-5-1-3-6(4-2-5)11(8,9)10/h1-4H,7H2,(H,8,9,10)
Clé InChI
HVBSAKJJOYLTQU-UHFFFAOYSA-N
Vous recherchez des produits similaires ? Visite Guide de comparaison des produits
Application
Point d'éclair (°F)
Not applicable
Point d'éclair (°C)
Not applicable
Équipement de protection individuelle
Faceshields, Gloves, Goggles, type ABEK (EN14387) respirator filter
Faites votre choix parmi les versions les plus récentes :
Déjà en possession de ce produit ?
Retrouvez la documentation relative aux produits que vous avez récemment achetés dans la Bibliothèque de documents.
Les clients ont également consulté
Notre équipe de scientifiques dispose d'une expérience dans tous les secteurs de la recherche, notamment en sciences de la vie, science des matériaux, synthèse chimique, chromatographie, analyse et dans de nombreux autres domaines..
Contacter notre Service technique