靶向蛋白降解
靶向蛋白降解是一種新穎的策略,利用小分子來劫持內源蛋白分解系統,以降解疾病相關蛋白。降低細胞中蛋白質的豐度,可實現與基因編輯方法(如 CRISPR-Cas9)類似的表型,但具有小分子帶來的優勢。這不僅是研究選擇性蛋白質敲除影響的研究工具,也因其優點而迅速被全球藥物發現界所採用,其優點在於佔位抑制和藥物治療 ~80% 傳統上對小分子藥物難以治療的蛋白質。用於這些方法的製劑稱為蛋白質降解劑,例如蛋白質分解靶向嵌合體 (PROTAC® degraders) 或分子膠。
異種功能蛋白質降解器一端含有目標結合彈頭,另一端含有E3泛素配體酶靶向配體,中間以連結器連接(圖1)。當使用降解器時,它會將目標物募集到 E3 配接酶。1-3
PROTAC® is a registered trademark of Arvinas Operations, Inc、的註冊商標,並經授權使用。

圖 1.透過蛋白質分解靶向嵌合體 (PROTAC) 進行靶向蛋白質降解
蛋白降解器合成的挑戰
降解器的設計具有挑戰性,因為結構的輕微改變會改變三元複合物的形成和隨後的降解。4圖 2中基於 PDB 5T35 的 3D 模型說明了精心設計以實現與兩個不同蛋白質(靶蛋白和 E3 配接酶)結合以及建立蛋白質-蛋白質介面的重要性。即使隨著計算化學的進步,降解器的設計在很大程度上仍然是一個經驗性的過程,研究人員會生成降解器的文庫,採用模組化的方法來改變配體、連結物和出口載體,這是一個高强度的前期化學工作。

圖 2.a)具有代表性的 PROTAC MZ1 的化學結構,突出了目標配體、E3 配體、連結器和出口載體。 b)降解器結構影響三元複合物的形成,以 MZ16 為例。 c)經驗設計需要生成文庫,以逐案進行測試。
簡化合成異多功能降解劑庫
我們的蛋白質降解劑構成區塊是從單一起始目標配體產生異多功能降解劑篩選庫的最簡單方法,可加速降解劑的發現。在這個包含了所有構建降解器元件的組合砌塊系列中,我們的配體-連接器共轭物省去了前期合成步驟,只需要在終端官能基上連接目標配體的化學反應(圖 3)。此外,如果選擇相同的末端化學,化學家可以同時將 50+ 配體連結結合物與一個起始目標配體平行反應,以產生一個初始篩選庫。

圖 3.配體連結劑共結物
配體 - 連結劑結合物的多樣性
我們的配體 - 連結劑結合物套件包含 E3 配體、出口載體、連結劑和末端化學的策略性組合:7我們的結合物包括配體和經驗證的 E3 配體 CRBN、VHL、IAP 和 RNF4 的各種出口載體(圖 4)。
連結劑:烷基和 PEG 連結劑是極佳的開端,可提供一系列疏水性、彈性和長度的樣品。此外,我們提供許多「混合」8 和剛性9-11連結劑,讓您的連結劑庫達到多樣化的連結劑特性 (圖 3)。
終端化學:有多種常用的官能基團可用於連結目標彈頭;我們最大的官能基團包括末端胺(圖 3)。
優勢
- 節省合成時間:配體連結結合物簡化了單一降解物的合成和平行合成的文庫建構
- 分子設計:最有興趣的 E3 配體、出口向量和連結劑在共結物中的組合可簡化前期組合庫設計
- 相容性:連結劑可與目標配體上的常見官能基團結合
- SAR:配體-連結劑共結物內建的策略性元件變異,可預先窺視 SAR,以進行有根據的最佳化
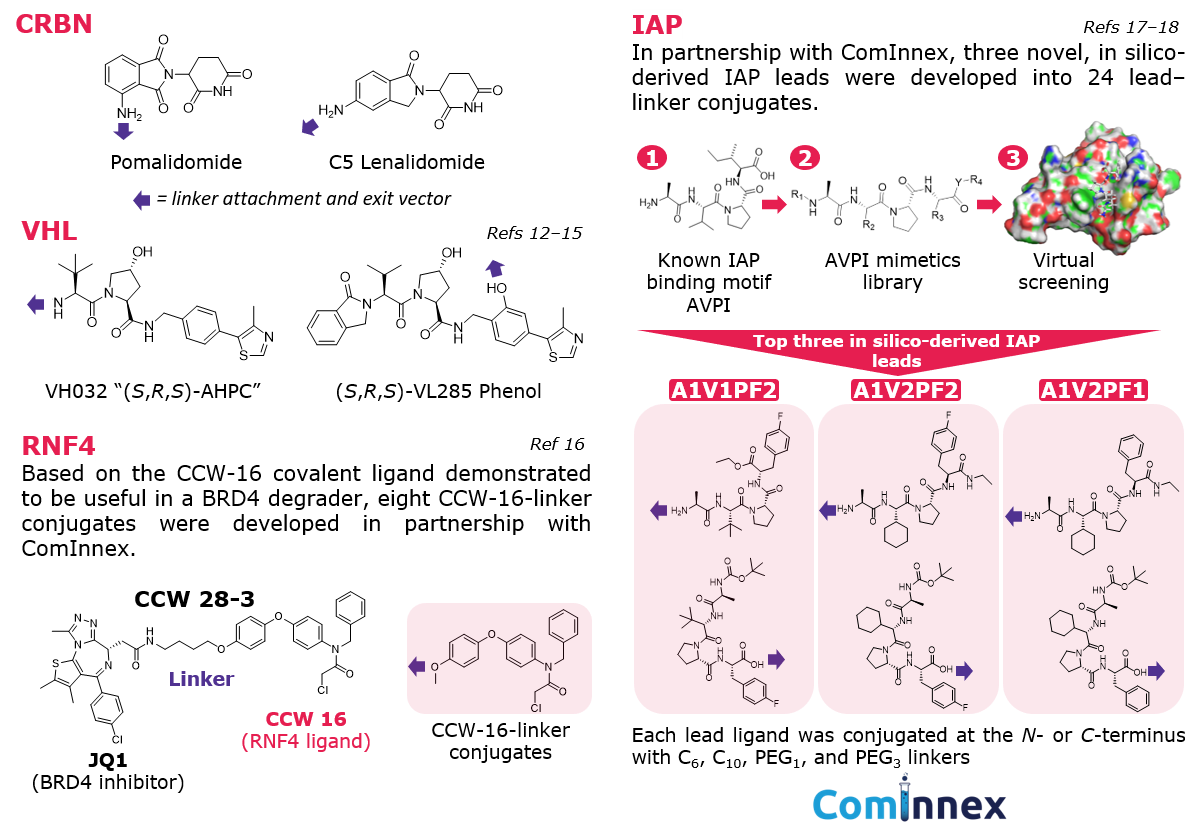
圖 4.結合物中的 E3 配體酶配體
Let us help you build your DEGRADER Library based on what is most important to you.
請聯絡您的 Merck 技術專家或 SigmaAldrich.com/techservice,取得所有合成產品的可排序清單或結構資料檔案,包括 150+ 配體連結結合物、250+ 異功能連結物、20+ 配體及相關探針化合物。
相關文章
- Degrader Building Blocks with IAP In Silico-Derived Ligands
- Accelerating IAP-Based Protein Degrader Discovery with Novel Ligand Library
網路研討會
。參考資料
若要繼續閱讀,請登入或建立帳戶。
還沒有帳戶?