分析基因表达的方法之一是测量基因的mRNA浓度。这种分析存在一些挑战,例如不同转录物之间的半衰期差异,转录的时间模式以及mRNA与蛋白质之间缺乏相关性。为利用PCR方法分析RNA,必须使用逆转录(RT)产生cDNA。该过程采用了逆转录酶和dNTP。
RT步骤可针对总RNA进行,从而能够全局表示许多转录物(通常通过两步实验方案),或者在基因特异性方法中,只有目标RNA被转化为cDNA(通常通过一步实验方案)。
目前已经确证两步RT反应的输入RNA与cDNA产率并不总是线性的1,因此确定和控制提取的RNA总量以及包括在RT反应中的RNA总量是很重要的。测量RNA浓度存在不确定性,绝对值取决于用于进行测量的仪器或系统。从图8.1中可以看出,当使用Nanodrop、常规紫外分光光度法、Ribogreen、Agilent 2100 BioAnalyzer或者Bio-Rad Experion时,5个RNA样品(A-E)的浓度测量值之间存在很大差异(参见样品纯化和质量评估)。值得注意的是,取决于使用哪种系统,EDTA(其抑制下游RT和PCR)的存在(样品D和E)和降解(样品C),对浓度测量值具有不同的影响。该观察结果说明了在下游反应中使用样品之前执行额外质量控制步骤的重要性(参见样品纯化和质量评估)。

图 8.1.使用Nanodrop、常规UV分光光度法、Bio-Rad Experion、Agilent 2100 BioAnalyzer(所有次数的重复测量)或Ribogreen(单次测量)测量五种总RNA样品(A-E)的浓度。可以看出,样品之间的绝对浓度以及相对浓度是不同的。这是因为样品C降解,样品D和E含有EDTA,这导致Experion和BioAnalyzer系统的测量结果不准确。

图 8.2.将总RNA稀释100倍,并使用两步随机引发进行逆转录;共进行了两次独立的RT反应。对于每个RT反应,在重复的qPCR中检测到β-肌动蛋白。RT是可再现的,但cDNA产量与输入RNA浓度不成比例。因此,如果实验限制要求在RT中包含可变浓度的RNA,则必须验证实验方案和试剂组合能够导致线性响应。
在图8.3所示的例子中,ReadyScript® RT试剂(RDRT)用于逆转录2倍和10倍连续稀释模板中的总RNA,该反应使用的是两步骤实验方案,而且组合使用了oligo-dT(O4387)引发和随机引发(如下所述)。在两个稀释系列中检测到CANX基因,其与输入RNA浓度成正比。

图 8.3.ReadyScript® RT试剂(RDRT)用于逆转录2倍和10倍连续稀释模板中的总RNA。在两个稀释系列中检测到CANX基因,其与输入RNA浓度成正比(数据来自参加EMBL Advanced qPCR Workshop的学生组)。
逆转录的引发
选择用于启动逆转录的引物可极大地影响RT-qPCR结果。对于一步法
RT-qPCR,使用基因特异性引物。对于两步法测定,则可以使用反向基因特异性引物、oligo-dT(O4387)、随机六聚体、九聚体、十聚体、十二聚体、十五聚体2、或者oligo-dT(O4387)与随机引物的组合。基因特异性引发通常在针对每种靶RNA的单独反应中进行。这些单独的反应可能具有非常不同的效率,因此使RNA浓度之间的比较复杂化。另一方面,当使用基因特异性引物时,所有RT产物将编码目标基因,并且可以对使用非特异性RT引物无法检测的极低丰度mRNA进行定量。为了避免在使用基因特异性引物时可能在不同测定之间产生较高的RT差异,可以使用非特异性引物来产生cDNA库。随后可以用来自cDNA库的等分试样对每个靶标进行单独的qPCR测定。如果所有qPCR靶标都在多聚腺苷酸化mRNA的3'末端附近,则oligo-dT(O4387)是合适的引物选择。另一方面,如果qPCR靶标距离3'末端超过几千碱基,或者如果RNA不是多聚腺苷酸化的,则随机引物将产生更可靠的检测。如果qPCR靶标相对于3'的位置是变化的,或所需的转录物同时含有多聚腺苷酸化和非多聚腺苷酸化的RNA,则oligo-dT(O4387)和随机寡聚物的混合物将产生最佳结果。
两步法RT反应中的逆转录引发
两步法RT引发常用两种方法:oligo-dT(O4387)和随机引发。oligo-dT(O4387)方法依赖于oligodT(通常为15聚合体)与存在于大多数mRNA分子的3'末端的聚-A尾的杂交,以引发和选择性地逆转录mRNA。
这种方法虽然在概念上非常简单,但也存在相关的困难:oligo-dT(O4387)引物结合在RT反应温度下对mRNA不具有特异性,因此oligo-dT(O4387)将非特异性地结合至RNA的其他区域。另外,还检测到延伸的rRNA,因为这些分子中的ATrich区域由oligo-dT(O4387)引发。有些mRNA,例如编码组蛋白的mRNA,不含聚A尾,可能不会在所得到的cDNA中体现。
第二种方法使用随机引发。随机引物由随机序列组成,通常为六聚体或九聚体。这些用于引发RT反应,导致合成不同长度的cDNA片段,其代表原始RNA。随机引物沿着转录物的长度方向杂交,并且倾向于比oligo-dT(O4387)或基因特异性引发更耐受二级结构。
为了从相应的技术优势中受益,一些实验方案采用两种引物类型的组合。靶序列的特异性引物也可以用于两步RT方案,但更常用于一步法(见下文)。
一步法RT反应中的逆转录引发
在一步法RT方案中,基因特异性引物用于逆转录单个靶标。基因特异性引物的设计至关重要,当在RT反应的温度下预测时,它必须位于mRNA靶标的开放的可接近区域内。在这些条件下,输入RNA与cDNA之间存在线性关系(图8.4)。该引物可以是(并且通常是)常用的PCR引物。

图 8.4.对总RNA进行10倍连续稀释,并进行一步RT-qPCR,以检测GAPDH。每个反应一式三份进行。这在RNA浓度与cDNA产量之间产生了线性关系(图片由英国安格利亚鲁斯金大学的Stephen Bustin教授友情提供)。
逆转录是一个高度可变的过程,必须考虑所有环节以使反应组分尽可能保持恒定。执行一步法RT-qPCR反应可减少所需的移液步骤数量并减少错误。因此,在需要测量小差异并且准确性至关重要的情况下,它可能是首选方法。然而,相反的考虑是,目标基因(GOI)与参考基因的比率的确定(参见数据分析)需要两个单独的一步法RT反应,而不是来自两步反应的单个cDNA,这两个目标不能使用多重qPCR方法检测。
逆转录效率
通常假设RT反应中的所有RNA / mRNA都转化为cDNA,并且所有转录物以1:1的比例转化,或者与起始RNA浓度成比例地转化。最近已经开始研究这些假设。很明显,转化成cDNA的RNA量是高度可变的。两步法RT过程是可变的,并且大大取决于RNA浓度、酶、缓冲液组成和引发方案。由于该过程是可变的,因此保持尽可能多的恒定条件非常重要1,3,5。
对于两步法RT反应,通常有必要使用相同的输入RNA浓度,并保持引发条件、RT酶和缓冲液恒定。当无法确定恒定的输入浓度时,建议使用一步法,包括聚乙二醇(PEG)等载体6,或选择经过验证可产生线性响应的商购试剂盒,例如ReadyScript® RT。
引发策略的选择影响绝对产量和来自总RNA样品的不同cDNA靶标的比例。图8.5显示了RNA样品中三种不同基因(通过直方图的不同颜色显示)的cDNA拷贝数产量,反应中样品还经受了受控的酶促降解。很明显,不同的引发方法对不同的样品所产生的绝对拷贝数和比例不同。此外,基因特异性引发似乎比oligo-dT引发更能耐受RNA降解。

图 8.5.将总RNA在裸露的人手上孵育指定的时间(1、2.5、5和7.5分钟)。cDNA是用基因特异的、随机的或oligo-dT(O4387)引发制备的。确定了三个基因的拷贝数,如直方图中紫色、绿松石和橙色条所示。很明显,不同的引发策略影响每个基因的检测,用oligo-dT(O4387)引发的结果是没检测到基因3(橙色),而用基因特异性引发则明显地在RNA样本中检测到(数据由英国安格里亚鲁斯金大学的Stephen Bustin教授提供)。
RT反应所使用的温度可能影响特异性,尤其是在杂交基因特异性引物时。可形成强3'-双链体的引物在较低温度下更容易杂交。由于RT酶可以从DNA模板上的DNA引物延伸,因此引物二聚体形成可以在RT步骤期间开始。 将RT孵育温度提高至酶完全活化的最高温度,或者使用高温酶,可以降低引物二聚体产物的量。当使用莫洛尼鼠白血病病毒-逆转录酶(MMLV-RT)在45°C进行RT(图8.6B)时,图8.6中使用的引物在一步RT-qPCR后,相对于非特异性产物,所产生的特异性产物比在37°C下进行RT(图8.6A)所产生的特异性产物要多得多。类似地,使用RT非特异性引物和qPCR热启动Taq聚合酶进行两步RT-qPCR(图8.6D)时,所产生的引物二聚体产物比使用可以形成3'-双链体的基因特异性引物进行一步RT-qPCR(图8.6B)所产生的引物二聚体产物少。
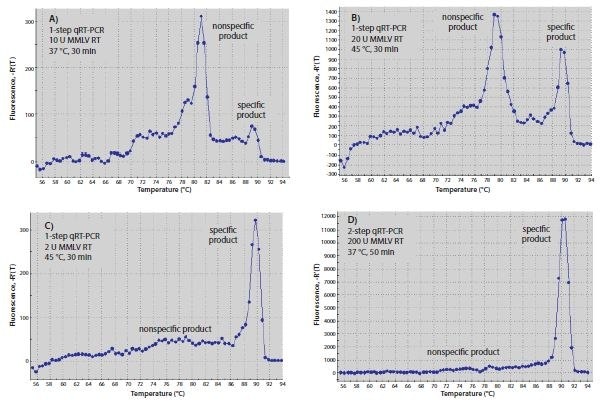
图 8.6.RT的优化。一步法(A-C)或两步法(D)RT-qPCR产生的RT-qPCR产物的解链曲线。反应A-C各自含有10μL SYBR® Green JumpStartTM Taq ReadyMixTM,0.02μL参比染料,两种0.4μM基因特异性引物和10ng人总RNA,终体积为20μL。基因特异性引物是5’-CGGGCTTCAACGCAGACTA-3´和 5´ -CTGGTCGAGATGGCAGTGA-3´,用于c-fos(登记号:NM_005252)。反应A和B还含有20单位的MMLV-RT,而反应C含有2单位。反应A在qPCR之前在37℃下孵育30分钟,而反应B和C则在qPCR之前在45℃下孵育30分钟。在D中,RT反应含有1x MMLV缓冲液(产品号D8559),0.5mM dNTP,1μM oligo-dT(O4387),0.8单位/μL RNase抑制剂,200单位MMLV-RT,和10ng人总RNA,终体积为20μL。将反应在25℃下孵育10分钟,在37℃下孵育50分钟,然后在80℃下孵育10分钟。将2μL RT反应产物添加到含有10μL SYBR® Green JumpStart™ Taq ReadyMix™、0.02μL参比染料和两种0.4μM基因特异性引物的qPCR中进行一步反应(A-C)。将所有qPCR反应物在94℃下孵育3分钟以使其变性,然后进行40个94℃、15秒和60℃、1分钟孵育循环。
每个反应的RT酶的量也会影响RT-qPCR结果。如图8.6所示,用2单位MMLV-RT进行的一步反应(图8.6C)比用20单位进行的反应(图8.6B)更具特异性。对RT使用oligo-dT(O4387)或随机引物的两步法RT-PCR,比一步法RT-PCR(图8.6D)产生更高的特异性。这可归因于在低温RT反应期间不存在基因特异性引物,从而阻止了非特异性产物的形成。较高浓度的RT在两步反应中可能会产生更好的结果,但由于RT酶可干扰Taq DNA聚合酶活性7,转移至qPCR的RT产物的量应限制在不超过最终反应体积的10%。该建议的例外是当使用ReadyScript时,在这种情况下,RT反应物可以是PCR体积的25%,而不会影响PCR效率。
对RT过程中固有变量的描述,表明了对来自RT的基因
表达的确定取决于所用的RT方法、样品的数量和质量、以及模板数量的考虑,应仔细予以报告,如MIQE指南8中所述(参见定量PCR)。
参考文献
如要继续阅读,请登录或创建帐户。
暂无帐户?