05-1336
Anticorps anti-phospho-histone H3 (Ser10), clone CMA312
clone CMA312, from mouse
Synonyme(s) :
H3S10P, Histone H3 (phospho S10), H3 histone, family 3A
About This Item
Produits recommandés
Source biologique
mouse
Niveau de qualité
Forme d'anticorps
purified antibody
Type de produit anticorps
primary antibodies
Clone
CMA312, monoclonal
Espèces réactives
vertebrates, human
Technique(s)
ChIP: suitable
ELISA: suitable
immunocytochemistry: suitable
multiplexing: suitable
western blot: suitable
Isotype
IgG1
Numéro d'accès NCBI
Numéro d'accès UniProt
Conditions d'expédition
wet ice
Modification post-traductionnelle de la cible
phosphorylation (pSer10)
Informations sur le gène
human ... H3C1(8350)
Description générale
Spécificité
Application
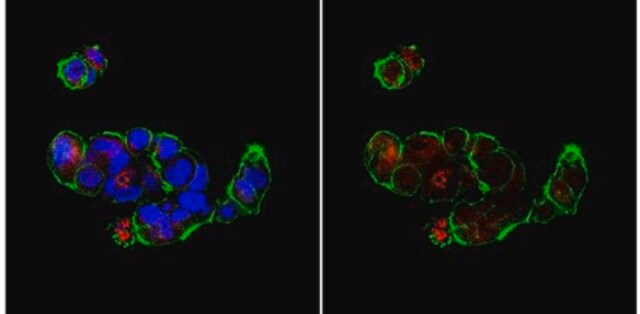
Immunocytochimie : Un laboratoire extérieur a montré que cet anticorps convient à l'immunocytochimie.1
Multiplexage :
Cet anticorps reconnaît spécifiquement l'histone H3 phosphorylée au niveau du résidu Ser10 dans un essai Luminex®.
Qualité
Description de la cible
Liaison
Forme physique
Autres remarques
Informations légales
Vous ne trouvez pas le bon produit ?
Essayez notre Outil de sélection de produits.
En option
Code de la classe de stockage
10 - Combustible liquids
Classe de danger pour l'eau (WGK)
WGK 2
Point d'éclair (°F)
Not applicable
Point d'éclair (°C)
Not applicable
Certificats d'analyse (COA)
Recherchez un Certificats d'analyse (COA) en saisissant le numéro de lot du produit. Les numéros de lot figurent sur l'étiquette du produit après les mots "Lot" ou "Batch".
Déjà en possession de ce produit ?
Retrouvez la documentation relative aux produits que vous avez récemment achetés dans la Bibliothèque de documents.
Notre équipe de scientifiques dispose d'une expérience dans tous les secteurs de la recherche, notamment en sciences de la vie, science des matériaux, synthèse chimique, chromatographie, analyse et dans de nombreux autres domaines..
Contacter notre Service technique