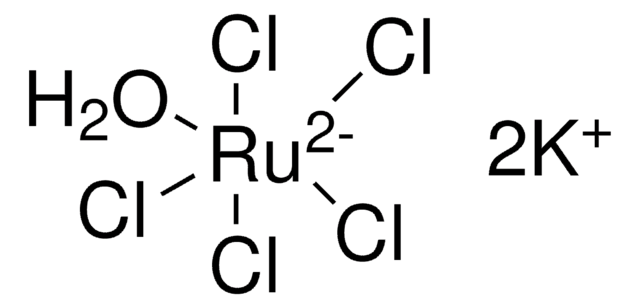
334537
Potassium perruthenate
Synonyme(s) :
Potassium ruthenium oxide, Potassium ruthenium tetraoxide, Potassium tetraoxoruthenium
About This Item
Produits recommandés
Forme
solid
Niveau de qualité
Pertinence de la réaction
reagent type: oxidant
Concentration
≥49.0% (gravimetric)
Solubilité
H2O: slightly soluble(lit.)
Chaîne SMILES
[K+].[O-][Ru](=O)(=O)=O
InChI
1S/K.4O.Ru/q+1;;;;-1;
Clé InChI
RDFCRPAXZNYVJH-UHFFFAOYSA-N
Vous recherchez des produits similaires ? Visite Guide de comparaison des produits
Mention d'avertissement
Danger
Mentions de danger
Conseils de prudence
Classification des risques
Eye Irrit. 2 - Ox. Sol. 2 - Skin Irrit. 2 - STOT SE 3
Organes cibles
Respiratory system
Code de la classe de stockage
5.1B - Oxidizing hazardous materials
Classe de danger pour l'eau (WGK)
WGK 3
Point d'éclair (°F)
Not applicable
Point d'éclair (°C)
Not applicable
Équipement de protection individuelle
dust mask type N95 (US), Eyeshields, Gloves, type P3 (EN 143) respirator cartridges
Faites votre choix parmi les versions les plus récentes :
Déjà en possession de ce produit ?
Retrouvez la documentation relative aux produits que vous avez récemment achetés dans la Bibliothèque de documents.
Notre équipe de scientifiques dispose d'une expérience dans tous les secteurs de la recherche, notamment en sciences de la vie, science des matériaux, synthèse chimique, chromatographie, analyse et dans de nombreux autres domaines..
Contacter notre Service technique