334537
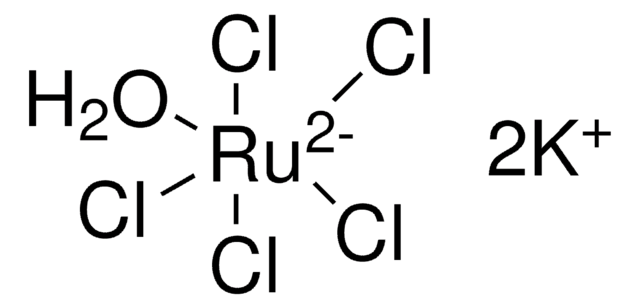
Potassium perruthenate
Sinónimos:
Potassium ruthenium oxide, Potassium ruthenium tetraoxide, Potassium tetraoxoruthenium
About This Item
Productos recomendados
formulario
solid
idoneidad de la reacción
reagent type: oxidant
concentración
≥49.0% (gravimetric)
solubilidad
H2O: slightly soluble(lit.)
cadena SMILES
[K+].[O-][Ru](=O)(=O)=O
InChI
1S/K.4O.Ru/q+1;;;;-1;
Clave InChI
RDFCRPAXZNYVJH-UHFFFAOYSA-N
¿Está buscando productos similares? Visita Guía de comparación de productos
Palabra de señalización
Danger
Frases de peligro
Consejos de prudencia
Clasificaciones de peligro
Eye Irrit. 2 - Ox. Sol. 2 - Skin Irrit. 2 - STOT SE 3
Órganos de actuación
Respiratory system
Código de clase de almacenamiento
5.1B - Oxidizing hazardous materials
Clase de riesgo para el agua (WGK)
WGK 3
Punto de inflamabilidad (°F)
Not applicable
Punto de inflamabilidad (°C)
Not applicable
Equipo de protección personal
dust mask type N95 (US), Eyeshields, Gloves, type P3 (EN 143) respirator cartridges
Certificados de análisis (COA)
Busque Certificados de análisis (COA) introduciendo el número de lote del producto. Los números de lote se encuentran en la etiqueta del producto después de las palabras «Lot» o «Batch»
¿Ya tiene este producto?
Encuentre la documentación para los productos que ha comprado recientemente en la Biblioteca de documentos.
Nuestro equipo de científicos tiene experiencia en todas las áreas de investigación: Ciencias de la vida, Ciencia de los materiales, Síntesis química, Cromatografía, Analítica y muchas otras.
Póngase en contacto con el Servicio técnico