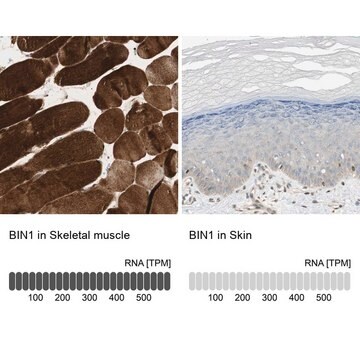
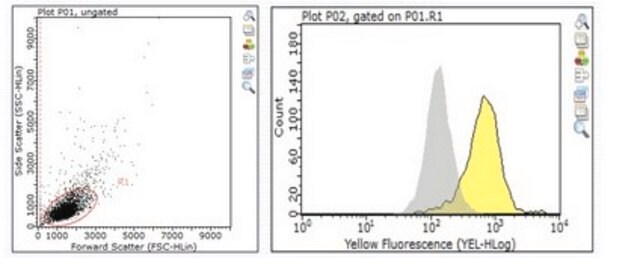
SAB1408547
Anti-BIN1 antibody produced in rabbit
purified immunoglobulin, buffered aqueous solution
Synonyme(s) :
AMPH2, AMPHL, DKFZp547F068, MGC10367, SH3P9
About This Item
Produits recommandés
Source biologique
rabbit
Conjugué
unconjugated
Forme d'anticorps
purified immunoglobulin
Type de produit anticorps
primary antibodies
Clone
polyclonal
Forme
buffered aqueous solution
Poids mol.
antigen ~53 kDa
Espèces réactives
human
Technique(s)
immunofluorescence: suitable
western blot: 1 μg/mL
Numéro d'accès NCBI
Numéro d'accès UniProt
Conditions d'expédition
dry ice
Température de stockage
−20°C
Modification post-traductionnelle de la cible
unmodified
Informations sur le gène
human ... BIN1(274)
Catégories apparentées
Description générale
Immunogène
Sequence
MAEMGSKGVTAGKIASNVQKKLTRAQEKVLQKLGKADETKDEQFEQCVQNFNKQLTEGTRLQKDLRTYLASVKAMHEASKKLNECLQEVYEPDWPGRDEANKIAENNDLLWMDYHQKLVDQALLTMDTYLGQFPDIKSRIAKRGRKLVDYDSARHHYESLQTAKKKDEAKIAKAEEELIKAQKVFEEMNVDLQEELPSLWNSRVGFYVNTFQSIAGLEENFHKEMSKLNQNLNDVLVGLEKQHGSNTFTVKAQPSDNAPAKGNKSPSPPDGSPAATPEIRVNHEPEPAGGATPGATLPKSPSQLRKGPPVPPPPKHTPSKEVKQEQILSLFEDTFVPEISVTTPSQPAEASEVAGGTQPAAGAQEPGETAASEAASSSLPAVVVETFPATVNGTVEGGSGAGRLDLPPGFMFKVQAQHDYTATDTDELQLKAGDVVLVIPFQNPEEQDEGWLMGVKESDWNQHKELEKCRGVFPENFTERVP
Forme physique
Clause de non-responsabilité
Not finding the right product?
Try our Outil de sélection de produits.
En option
Code de la classe de stockage
10 - Combustible liquids
Classe de danger pour l'eau (WGK)
WGK 1
Point d'éclair (°F)
Not applicable
Point d'éclair (°C)
Not applicable
Certificats d'analyse (COA)
Recherchez un Certificats d'analyse (COA) en saisissant le numéro de lot du produit. Les numéros de lot figurent sur l'étiquette du produit après les mots "Lot" ou "Batch".
Déjà en possession de ce produit ?
Retrouvez la documentation relative aux produits que vous avez récemment achetés dans la Bibliothèque de documents.
Notre équipe de scientifiques dispose d'une expérience dans tous les secteurs de la recherche, notamment en sciences de la vie, science des matériaux, synthèse chimique, chromatographie, analyse et dans de nombreux autres domaines..
Contacter notre Service technique