Quantifizierung von Oligonukleotiden
Oligonukleotide sind kurze, einzel- oder doppelsträngige Polymere aus Nukleinsäuren. Die Konzentration von Nukleinsäuren in Lösung lässt sich in einem Spektralphotometer leicht quantifizieren, wenn sie ultraviolettem (UV) Licht bei 260 nm ausgesetzt wird (Abbildung 1). Diese Technik funktioniert, weil die Basen konjugierte Doppelbindungen haben, die UV-Licht unterschiedlich absorbieren.

Abbildung 1. Absorbanz in einem Spektralphotometer.Die Nukleinsäure in Lösung in der Probenzelle wird mit einfallendem UV-Licht bei 260 nm und maximaler Intensität beleuchtet. Da die Nukleinsäurebasen einen Teil des UV-Lichts absorbieren, wird die Intensität des übertragenen UV-Lichts verringert. Die Absorbanz wird anhand der folgenden Formel bestimmt:
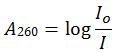
Wobei: A260 = Absorbanz bei 260 nm; Io = Intensität des einfallenden Lichts; und I = Intensität des durchgelassenen Lichts.
Nach einer Absorbanzmessung kommt das Lambert Beer‘sche Gesetz ins Spiel, das die Höhe der Absorbanz mit der Konzentration der absorbierenden Nukleinsäure wie folgt in Beziehung setzt:
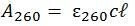
Wobei:
A260 = Absorbanz bei 260 nm
260 = Extinktionskoeffizient des Oligonukleotids bei 260 nm in l ⋅ mol-1 ⋅ cm-1
c = Konzentration in mol/l-1
ℓ = Weglänge in cm
Der Schlüssel zum Lambert Beer‘schen Gesetz ist der Extinktionskoeffizient, der angibt, wie stark die absorbierenden Basen die Intensität des UV-Lichts vom Einfall bis zur Transmission reduzieren. Da die Absorbanz jeder Base unterschiedlich ist, beeinflussen Basenzusammensetzung, Basenanordnung und Sequenzlänge die endgültige Absorbanz der jeweiligen Nukleinsäure, die quantifiziert wird. Die größte Genauigkeit wird daher erreicht, wenn der Extinktionskoeffizient für jedes Oligonukleotid berechnet wird. Das Modell des nächsten Nachbarn ist ideal für Oligonukleotide. Die Formel lautet wie folgt:

In Tabellen 1 und 2 sind die Extinktionskoeffizienten der nächsten Nachbarn und der einzelnen Basen aufgeführt, die in der Formel für die nächsten Nachbarn bei der Berechnung des finalen Gesamtextinktionskoeffizienten eines Oligonukleotids verwendet werden.
Da die Weglänge der Probenzelle 1,0 cm beträgt, sind nun alle Eingaben in das Lambert Beer‘sche Gesetz berücksichtigt, sodass die Gleichung umgestellt werden kann, um die Konzentration des betreffenden Oligonukleotids zu berechnen.
Zur Berechnung der Konzentration wird das Lambert Beer‘sche Gesetz wie folgt umgestellt:

Beispiel: Berechnung für 5'-ACGT-3'
Erster Schritt: Berechnung des Extinktionskoeffizienten
260 = (AC+CG+GT)-(C+G) L ⋅ mol-1 ⋅ cm-1
260 = (21.200+18.000+20.000)-(7.400+11.500) l ⋅ mol-1 ⋅ cm-1
260 = 40.300 l ⋅ mol-1 ⋅ cm-1
In allen technischen Online-Anzeigen und Druckunterlagen wird der vorgenannte Wert von 40.300 als 40,3 angezeigt. Das liegt daran, dass die Einheiten l ⋅ mmol-1 ⋅ cm-1 (Millimol statt Mol) sind.
Schritt zwei: Berechnung der Konzentration
A260 wird auf 1,0 Einheiten der optischen Dichte (OD) eingestellt



Schritt drei: Berechnung der molaren Masse (MM)



Normalerweise wird "Molekulargewicht" anstelle von "molare Masse" verwendet, aber da die Masseneinheiten eines Mols für diese Berechnungen benötigt werden, ist "molare Masse" der korrekteste Begriff. Zur Vereinfachung der Darstellung wurden die vorgenannten molaren Massen der einzelnen Basen auf die nächste ganze Zahl gerundet. Daher liegt der berechnete Wert von 1.170 leicht unter dem tatsächlichen Wert von 1.173,83.
Schritt vier: Umrechnung der Konzentration in gebräuchliche Einheiten

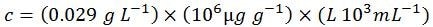

Daher gilt für diese spezielle Sequenz: 5'-ACGT-3', 1 OD = 29,0 µg ml-1 (ein OD ist die Menge an Oligonukleotid in 1 ml Lösung, die ein A260 von 1,0 bei einer Lichtweglänge von 1,0 cm aufweist). Zum Vergleich sehen Sie hier den Wert, der von OligoEvaluator™, unserem Online-Rechner für Oligonukleotidsequenzen, ermittelt wurde:

Es besteht eine ausgezeichnete Übereinstimmung zwischen dem manuell berechneten Wert von 29,0 und dem von OligoEvaluator berechneten Wert von 29,1 (die Diskrepanz ist darauf zurückzuführen, dass bei der manuellen Berechnung gerundete molare Massen der einzelnen Basen verwendet wurden, wie weiter oben in Schritt 3 erläutert).
Zusätzlich zu OligoEvaluator ist 29,1 der Wert, der auf dem technischen Datenblatt dieses Oligonukleotids angezeigt wird.
Berechnung in Kurzform für 5'-ACGT-3'
Die obigen Schritte der Berechnung in Langform sind mühsam. In der folgenden Formel werden alle Schritte zu einem Schritt zusammengefasst. Sie kann daher als Kurzform verwendet werden:



Modifikationen
Der einzige Unterschied zwischen der Berechnung der Konzentration für ein Standard-Oligonukleotid und ein modifiziertes Oligonukleotid besteht darin, dass die molare Masse der Modifikation berücksichtigt und zur gesamten molaren Masse des Oligonukleotids addiert wird. Zum Beispiel, wenn unsere obige Sequenz:
● 5'-ACGT-3'
verändert wird in
● 5'-6-FAM™ ACGT-3'
und setzt man die neue molare Masse von 1.711,3 g mol-1 entweder in die Langform oder in die Kurzform ein, resultiert daraus die neue Konzentration
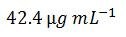
Dieser Wert stimmt gut mit dem von OligoEvaluator ermittelten Wert überein:

Schnellumrechnungen
Als Faustregel wurden in Versuchen die folgenden Schnellumrechnungen bestätigt (ds = doppelsträngig; ss = einzelsträngig):
● A260 dsDNA = 50 µg ml-1
● A260 ssDNA = 37 µg ml-1
● A260 ssRNA = 40 µg ml-1
Ohne weitere Berechnungen liefern diese Umrechnungsfaktoren fundierte Schätzungen für längere Sequenzen mit einer gleichmäßigeren Verteilung von A-, C-, G- und T-Basen, wie sie bei der Scherung genomischer DNA (für die dsDNA-Schätzung) entstehen. Bei Oligonukleotiden ist es jedoch wahrscheinlicher, dass die Basenzusammensetzung verzerrt ist, sodass diese Schätzungen unzuverlässig sind (oft wird die Konzentration überschätzt). Die Konzentration muss für jedes Oligonukleotid wie weiter oben beschrieben bestimmt werden, wobei entweder die Langform oder die Kurzform für die Berechnung verwendet werden kann. Zur Überprüfung der in den technischen Datenblättern angegebenen Quantifizierungswerte kann entweder die Langform- oder die Kurzformberechnung verwendet werden.
Um weiterzulesen, melden Sie sich bitte an oder erstellen ein Konto.
Sie haben kein Konto?