RT-PCR/RT-qPCRトラブルシューティング
PCRまたはRT-PCRのトラブルシューティングプロトコルの作成
オペレーターのミスによる問題の潜在的原因
オペレーターがミスする可能性は多々あります。こうしたミスの原因はなかなか特定できません。トラブルシューティング手順の最初のステップは、プロトコルを確認して実験を繰り返すことです。プロトコルを確認し(本書のプロトコル付録Aを参照)、経験豊富な分子生物学者に実験計画の見直しを依頼することが重要です。あるポスドク研究者がPCRに何度も失敗した後、PCRマスターミックスにdNTPが欠けていることに気づいたという経験談がありますが、多くの仕事をこなす優れた科学者でさえ単純なミスに陥りやすいことを示す例といえます。
マスターミックス
マスターミックス用の成分を混合時の間違いや問題は、すべてのサンプルおよびポジティブコントロールで増幅が失敗する原因となる可能性があります。実験を繰り返す前に、全成分とその濃度を確認してください。新ロットの試薬を使って一連の大規模実験を始める前に、旧ロットと新ロットを比べる対照実験をするのは有用な予防策です。
マスターミックス製品の切替えを行う際には、バッファー組成/アニール温度(Ta)/プライマー濃度の組み合わせに特に敏感な試験法があることを認識することが重要です。パラメータのいずれかを変更すると、性能や結果が変わる場合があります。このため、大幅な変更を行う前に、選択したマスターミックスと使用するすべての装置を使って、分析法をすべて検証してください。酵素、ホットスタート機構、およびバッファー成分には推奨の至適条件があるので、各マスターミックスに付属の説明書を確認することも重要です。
同時測定サンプルすべてに対し、反応用マスターミックスが十分量調製されていることを確認してください。実験用マスターミックスの全成分を注意して解凍し、よく混ぜ、混合が十分であることを確認してからサンプルに分注してください。これは特に、通常のPCRバッファーよりも粘性の高いKiCqStart®などの一部の2倍濃縮バッファーが該当します。
オリゴの最適化
オリゴは、配列が不適切であったり、設計不良であったり、至適濃度未満、至適Ta未満であったり、あるいは(プローブの)標識・消光が不適切であったりすると、問題を引き起こす可能性があります。オリゴに最適とは言えない条件で分析を行ったり、設計不良のオリゴで分析を行ったりすると、データが得られたとしても、検討対象の真のバイオロジーを反映していない可能性があります。凍結乾燥オリゴ製品を受けとったら、以下の実施が重要です:
- 配列を確認する
- すべてのDNAが再懸濁されていることを確認してから使用する
- 正しい濃度であることを確認する
オリゴを90℃で5分間加熱し、十分に混合して懸濁させてください。凍結融解の反復もオリゴの性能に影響を及ぼす可能性があるため、長期保存の場合は原液濃度(通常100 µM)のオリゴをすべて分注し、−20℃または-80℃で保存する必要があります。
トラブルシューティングでは、標的の配列に立ち戻ってオリゴ配列が実存することを確認し、正しい配列を注文したかどうかを検証することが重要です。製造業者に連絡し、オリゴの品質に間違いがないか確認しましょう。オリゴの使用濃度を測定し、蛍光分子が標識されていることを目で確認してください。SYBR®グリーンI qPCRミックスでプローブアッセイのプライマーをテストし、増幅を検証してください。プライマー濃度またはTaの最適化をご検討ください(アッセイの最適化および検証を参照)。プローブの初回使用時は、チャンネル間でのシグナルの漏れを見つけ標識の間違いを検出できるよう、できるだけ多くの波長で蛍光データを集めてください。
PCR最適化が不十分
分析の最適化の効果については、分析の最適化とバリデーションに記載しており、実証済みです。アッセイが失敗したり、性能が至適未満であるにもかかわらず、アッセイのデザインや操作手順に誤りがない場合には、実験条件の最適化が有益かもしれません。トラブルシューティングでは、100 nM、500 nM、900 nMの最終濃度や55~70℃(温度勾配を使用)のTaで、プライマーをテストし、最適化を進めることでアッセイが改善するかどうかを確認してください。
RT-PCR法・qPCR法のデザイン
分析法のデザインはPCR/qPCR/dPCR分析法のデザインに記載しました。分析法の不良診断を行うときは、分析法のデザインが検証済みであることを確認してください。PCR/qPCRプライマーおよび増幅産物の位置がRTプライミングプロトコルと一致していることを確認してください。例えば、オリゴ-dTプライミング後に調製されたcDNAに適用するアッセイは転写産物の3'側に位置しているかどうか確認してください。配列情報に信頼できること、該当するスプライスバリアントおよびSNPが考慮されていることを確認してください。

図11.1A.本アッセイの増幅プロットプロファイルは異常で、ベースラインから変動が顕著。

図11.1B.アッセイに含まれるプローブの配列を折り畳み回数予測ソフトに入力した。プローブが溶液中で安定した折り畳み構造をとれることは明白で、これが観察された問題を引き起こした可能性がある。
RNA/DNAテンプレートの質
テンプレートの質が分析性能に与える影響をSample Purification and Quality Assessmentに記載しました。テンプレートの質には、インヒビターの量、完全性、有無の評価が含まれます。RNAの品質が最適なRTプライミングプロトコル(逆転写を参照)に合致していることを確認し、できる限り最高品質のテンプレートを使うことが重要です。同様に、RT反応に加えるRNA量はプロトコルの範囲内でなければならず、多くの場合、すべての反応で同一でなければなりません。このガイドラインが該当しないのがReadyScript®です。本試薬とプロトコルを採用すると、cDNA濃度がRNA量に比例した線形性を示すからです。予想したCq よりも高いCqが得られたサンプルについてのトラブルシューティングでは、SPUDアッセイを実施するか、サンプルを1:5または1:10で段階希釈し、アッセイを繰り返してインヒビターを含むサンプルを同定します(図11.2)。


図11.2.テンプレートDNAの10倍段階希釈液の増幅。レプリケートの精度は高いが、ΔCqは一貫性がなく、希釈が進むにつれて減少する。また、データは、無テンプレートコントロール(NTC)で汚染またはプライマー二量体形成を示すポジティブシグナルが出ていることを示し、さらに105コピー未満の希釈液はNTCとデータが一致することを示している。
テンプレートの量も重要な考慮の対象です。PCRで使うテンプレートの量が多すぎる/少なすぎると、反応が上手くいかず、qPCR増幅プロットが通常と違って見えます。図11.3Aに、人工テンプレートオリゴの10倍段階希釈液を用いた反応を示しています。低希釈液は濃度が高すぎて反応を効率的に行えず、装置でベースラインデータを効率的に処理することもできないため(図11.3B)、増幅プロットが異常となり、データの信頼性が低下します。

図11.3.A)特異的プライマーとFAM標識プローブを用いて人工テンプレートの10倍段階希釈液を増幅。濃縮サンプルのCqが非常に低く、増幅プロットが等間隔でなく異常。B)上記増幅プロットの生データを示す。また、ターゲットが最高濃度の反応では、バックグラウンド蛍光も極めて高く、反応中の蛍光収率も極めて低かった。
PCRアッセイの計画
PCRサイクルの条件は、実験と試薬の両方に適していなければなりません(例として、「マスターミックス」参照)。検証を行わずに装置のデフォルト設定を採用することはお勧めできません。
サーマルサイクラーの不具合
装置の故障は知らないうちに発生するため、診断が困難な場合があります。高額な修理費を被らないよう、装置のオペレーター全員に十分なトレーニングを実施し、最初は監督者を配置してください。装置の故障で致命的な不具合が生じ、増幅や蛍光データが得られない場合もあれば、データが不正確になったりサンプルの処理が不均一になったりして、同一の生体サンプル同士の処理で人的な差異が生じてしまう場合もあります。コントロールサンプルとコントロールアッセイの併用は、不良診断に非常に有用です。装置の故障が疑われる場合、信頼性が高く、最適化された分析法をすべてのウェルに適用してください。この均一性チェックにより、装置の特定部位に起因する問題や、各測定・各装置の個別の問題が明らかになります。
診断ツールを使用するPCRトラブルシューティングの例
よく計画されたPCRの実行後のトラブルシューティングに使える診断ツールがいくつかあります。
- コントロールサンプルとアッセイ
- エンドポイントゲル/SYBR® Green I色素試薬
- 増幅プロット(レプリケートと増幅プロットプロファイルの確認)
- 検量線(グラジエント、R2)/希釈系列
- 融解/解離プロット(SYBR® Green I色素、Molecular ビーコン、Scorpions®プローブ)
- 生データ/多成分測定
コントロールサンプル/反応
コントロールの使用を強く推奨します。適切な一連のコントロールデータなしで、失敗したアッセイの不良診断をすることはほぼ不可能です。

図11.4.A)無希釈のテンプレートは増幅できないが、希釈すると増幅効率が改善した。B)qPCRミックスに0.3% BSAを添加すれば、無希釈のテンプレートからの増幅が可能となる。
分析が完全に失敗した場合、トラブルシューティングに利用できる情報がほぼないため、調査が困難です。分析失敗の多くは何らかの致命的エラーの結果であるため、まず実験の設定を確認してからPCRを繰り返してください。これで失敗した場合、実験の各要素に関する情報でトラブルシューティングのプロセスが決まります(図11-5)。

図11.5.PCRのトラブルシューティングの基本手順
qPCRアッセイが完全に失敗したときの最初のステップは、分析デザイン、オリゴ配列、およびオリゴ製造者提供のQCデータを確認することです。アッセイが失敗していた可能性があっても、qPCRの多成分/生データからさらに情報を得ることができます。図11.6Aに、6-FAM™またはHEX™(VIC®)で標識したプローブを含む2つのアッセイの生データプロットを示しています。どちらの測定法も増幅を示していますが、HEX™シグナルはFAMシグナルの約半分です。HEXは元来弱い色素なので、正常の結果です。アガロースゲルによる分析(図11.6B)では、両反応とも同程度の濃度の生成物が得られたことから、qPCRでのCq値は同程度であるという結果が裏付けられました。

図11.6.A)FAM標識プローブとHEX™標識プローブを含む二重染色アッセイの生データプロット。当然、FAMプローブのほうがより強い蛍光を発します。B)アガロースゲルにより各反応で同等量の生成物ができた、qPCRでのCq観察結果が確認できた。
生データの検査は、プローブが正しく標識され、反応に加えられたことを確認するのに有用な方法です。図11.7に、トリプレックスアッセイにおける3つのターゲットの増幅生データを示しています。各ターゲットに特異的なプローブを、FAM、HEX™、およびTAMRAで標識します。HEX™プローブおよびTAMRAプローブはバックグラウンドが低く、効率的な増幅を示しますが、FAMシグナルは常に一貫して高く、増幅の徴候がありません。これは、反応におけるプローブ濃度が高すぎること、あるいはシグナルの初回消光がないプローブの欠陥に一致する結果です。このような場合、プローブ濃度と分析デザインを検証し、プローブが適合した標識体と消光物質を有しているか検証し、必要であれば新しいプローブでテストしてください。
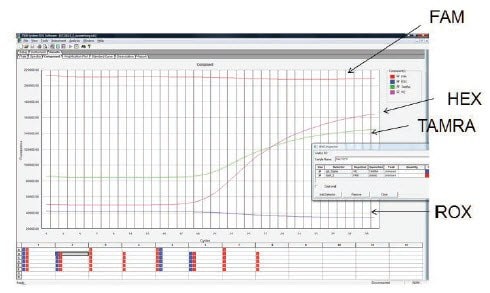
図11.7.トリプレックス反応により、FAM、HEX™、およびTAMRAで標識したプローブを用いて3つのターゲットを検出した。HEX™プローブおよびTAMRAプローブではターゲットから増幅できましたが、FAMプローブでは増幅できなかった。生データを検討したところ、バックグラウンド蛍光が非常に高く、反応中に一貫して差が認められなかった。これは、反応液中のプローブ濃度が高すぎる、あるいは消光が不十分なプローブの欠陥に一致する結果である。
初回の実験がプローブ検出に依存していた場合、ポジティブおよびネガティブコントロール(貴重なサンプルを除く)を含め、SYBR® Green I試薬を用いてアッセイを繰り返す必要があります。あるいは、失敗した反応の産物を臭化エチジウムで染色したアガロースゲルで調べることも可能です。汚染リスクを回避して初回の失敗を検証する再現実験ができるSYBR® Green I法を再現実験に採用することが望ましいです。SYBR® Green I実験でデータが得られた場合、初回のプローブの不具合は、技術的エラーまたはプローブの不具合のいずれかが原因であった可能性があります。測定ミスとプローブの不具合を識別するには、プローブ実験を再度行い、反応が再失敗した場合はプローブを交換してください。この方法は、データ不良の反応を調べる際に利用できます。図11.8に示す例では、プローブ反応が最適化されておらず、SYBR® Green Iを用いた反応と比較すると、プローブのシグナルが実験を反映していないことがわかります。このような場合、分析デザインを検証し、新しいプローブでテストを行ってください。

図11.8.qPCRプローブまたはSYBR® Green I色素のいずれかを含み、その他の条件は同一の反応を実行した。SYBR® Green I反応の感度は約11サイクル高く、エンドポイントの蛍光がはるかに強かった。これは、プローブの不具合またはプローブのデザインに問題があることを示している(出典:Professor Stephen Bustin、英国)。
プローブの標識の検証
生データまたは多成分プロットは、反応に適切な濃度のプローブが含まれているか、またプローブの標識と消光が適切かどうかを調べるための有用な診断ツールです。図11.9に、プローブ3つを用いた反応の多成分プロットを示しています。第1、第2のプローブからは増幅プロットが得られ、バックグラウンド蛍光が認められました。第3のプローブからデータは得られず、生データを検討した結果、バックグラウンド蛍光はプローブを含まない水ブランクコントロールと同等であることが判明しました。したがって、このデータは反応で蛍光が消失した結果を示しています。これは、プローブが含まれていなかったり、プローブが標識されていなかったという、設定時のエラーによるものと考えられます。

図11.9.同一のテンプレートサンプルから3つの遺伝子が検出された。2回の反応(1、2)では増幅が見られたが、第3の反応では増幅しなかった。多成分測定の結果、3回目の反応のバックグラウンド蛍光はシグナルが出ない水コントロールと同等であることが判明した。
DNase I分解を行えば、さらにプローブ標識の確認ができます。この際、プローブとプライマーの保存液が酵素に汚染されると不適切な結果が出るので、細心の注意を払って実施する必要があります。反応に含まれる量(例:300 nM)に相当する欠陥プローブ(図11.10A)を、DNase Iの存在下または非存在下でインキュベートします。これはリアルタイムで実施可能で(図11.10B)、蛍光強度を経時的に測定します。あるいは、最初と最後(10分後)の時点の測定値からでも十分な情報が得られます。このテストを行う場合、同じ蛍光色素で標識された同じクエンチャーを有し、正常に機能するプローブのデータと比較することが重要です(図11.10B)。

図11.10.A)FAMで標識した異なるプローブを用いて2つのテンプレートを検出した。第1プローブを用いたときは強い蛍光シグナルが検出されたが、第2プローブを用いたときのシグナルは大幅に低下した。B)リアルタイム装置にて、DNase Iの存在下、非存在下でコントロールプローブおよび試験プローブ(300 nM)をDNase Iバッファー中で37℃でインキュベートした。プローブ1を用いたときの蛍光強度はプローブ2を用いたときの約2倍であり、プローブ2の標識が不十分であることが示された。
増幅プロット
増幅プロットの形状および技術的レプリケートの再現データから、qPCR法の質に関する多くの情報が得られ、本来あるべき姿と全く違うという気づきが得られる可能性があります。図11.11Aの増幅プロットは定型から外れ、ノイズが非常に多く、正しい解釈は困難です。dR蛍光値をさらに検討したところ、エンドポイントの蛍光収率が400単位に過ぎないことが明らかになりました。これは、反応が不十分であることを示し、装置のソフトが増幅プロットを作成して自動スケーリングしたことを示しています。同様に、図11.11Bのデータは、プロファイリング開始時に明らかなFoxtail(減少曲線)を示し、ベースラインの測定後に再び増加しています。Foxtailの出現は2つの反応で一致していますが、一方の反応のほうがはるかに低いエンドポイントを有するため(図11.11C)、Foxtailが相対的に大きくなる可能性があります。
図11.11.A)機器ソフトウェアにより蛍光が弱い不良データをオートスケーリングした、ノイズの多い増幅プロット。B)エンドポイントdRが低い反応は、最初のFoxtailが顕著。C)高品質なアッセイに比例する場合、Foxtailは正常な影響と考えられる。
同様に、図11.12Aの増幅プロットは明らかに異常であり、提供時の状態で使用されていない恐れがあります。0 dR未満に低下する増幅プロット(図11.12A)は、ベースライン時の設定が不適切であることを示す典型的な指標です。この反応の生データを検討した結果(図11.12B)、実際の増幅プロットのプロファイルは正常であることがわかり、解析データは装置のソフトウェアの問題によることが確認できました。生データから適切なベースラインを推定し、ソフトに適用することが可能です。この場合、サイクル6~16が反応の最初の線形ベースライン相を示し、適用すると、増幅プロットが正常になりました(図11.12C)。

図11.12.A)増幅プロットは明らかに異常であり、プロファイルの一部がベースラインを下回った。B)生データプロットを検討した結果、反応データは予想通りであった。C)適切なサイクルに従って装置のベースラインを設定することにより、解析した増幅プロットのデータは正常なプロファイルに戻る。
増幅プロットプロファイルを解釈し、アッセイの品質および最適化に関する情報を得ることもできます。人工テンプレートオリゴの10倍段階希釈液を用いたqPCR反応を図11.13に示しています。各濃度をデュプリケートで測定しています。レプリケート間の再現性が悪く、データ間のサイクル差(ΔCq)が一定ではなく、10倍段階希釈の場合に予想される3.323サイクルではありません。これを検量線とみなして増幅プロットを検討した結果、本分析は標準を下回り、解析に使用できませんでした。理由については詳細な検討が必要ですが、分析のデザインが良くない(PCR/qPCR/dPCR分析デザインを参照)、分析条件が最適でない(分析の最適化・バリデーションを参照)、または不適切なピペット操作(アッセイの再実施)が原因かもしれません。

図11.13.cDNAサンプルを10倍段階希釈し、デュプリケートqPCRを用いて各希釈液で特異的テンプレートを検出した。再現性が低いため、ピペット操作またはアッセイの最適化に問題があることが示された。

図11.14.標準的qPCRでデータが急上昇し、非典型的なプロファイルを示す。

図11.15.蛍光プロットが減少またはフック状になる場合、考えられる原因は、相補的DNA鎖がプライマーおよび/またはプローブと競合し、テンプレートに結合するためと考えられる。Ctに影響がない限り、無視する。
解離/融解曲線
解離/融解曲線解析はqPCR後に実行されます。これはDNA結合色素(SYBR® Green Iなど)または非分解プローブ(Molecular ビーコンまたはScorpions®プローブなど)を併用し、単一産物が増幅されたことを確認するための解析ツールです。PCR増幅後、得られたアンプリコンを温度を上げながらインキュベートし、二本鎖状態と一本鎖状態の間でDNAが遷移する際の蛍光シグナルの変化を検出します。反応の結果、単一のアンプリコンができた場合、アンプリコンは均一に融解し、dF/dT(温度に対する蛍光の変化率)プロットのピークは1つです。融解曲線の検討は、コントロールデータを併合すると特に効果的です。図11.16Aに、実験で用いた試験サンプル、ポジティブコントロール、および無テンプレートコントロールのqPCR後の融解曲線を示しています。試験サンプルの融解曲線はポジティブコントロールと一致し、どちらもdF/dTのピークが1つあります。無テンプレートコントロールの融解曲線は、ピークの幅の広く、Tmが低いです。こうした結果は、ネガティブコントロールにプライマー二量体が存在することに合致します。このことは臭化エチジウムで染色したアガロースゲル(図11.16B)を用いて確認しました。図11.16Bもテンプレートが低濃度で存在するとプライマーの二量体が現れることを示しています。このため、ターゲット濃度が低いサンプルでターゲットが検出された場合、ターゲットの過大評価が起きます。したがって、この分析法は最適化、再設計の必要があります。これに対して、図11.16Cは、無テンプレートコントロールにおけるアンプリコンの融解曲線が、ポジティブコントロールおよびサンプルの融解曲線と同一であることを示しています。これは、実験の設定中にテンプレートを使用しないコントロールが汚染されたことの明確な印です。最後は、cDNAサンプル中に存在するgDNAからターゲットの増幅が認められた例です(図11.16D)。gDNA由来のアンプリコンはcDNA由来のものよりも長いため、Tmが高くなっています。

図11.16A.ポジティブコントロール、試験サンプル、およびNTCを増幅し、PCR後の融解曲線解析に供した。NTCには、プライマー二量体形成に一致する、低温で融解し、幅が広い融解ピークを持つ生成物が明らかに存在する。

図11.16B.上記希釈サンプルなどをゲル分解した結果、プライマー二量体が明らかに認められた。プライマー二量体の形成はテンプレート濃度に反比例する。

図11.16C.NTC反応液が汚染されたどうかを確認するために融解曲線解析を適用した例。
図11.16D.gDNAのPCRで生じたより大きなアンプリコンの同定。

図11.17.A)融解曲線、B)SYBR® Green I反応のアガロースゲル解析。融解曲線はTmの異なる産物を示唆しているが、ゲルイメージは単一のアンプリコンの存在を示している。このことは、ATまたはGCに富む領域あるいは反復配列を含む単一のアンプリコン配列によって、不規則な融解が起きていることを示す。
テンプレートの段階希釈/検量線
最終定量用の検量線の作成が実験デザインに含まれているか否かに関係なく、適切なテンプレートの段階希釈物を使った検出は、分析法のバリデーションやトラブルシューティングの際に非常に役立ちます。段階希釈物を使った検出により、定量のダイナミックレンジを求めることができます。図11.18Aは、低濃度のデータポイントが直線に乗らない検量線です。このようなデータパターンが起きる理由としては、低濃度のサンプルでプライマー二量体が形成される場合が多いです(図11.18B参照)。これは図11.2に示したデータから作成した検量線です。図11.18Cは、高濃度のサンプルが直線範囲から外れた検量線です。その理由は、テンプレートによる反応阻害またはベースラインの設定が不適切である場合が多いです。

図11.18.A)低濃度のターゲットのデータポイントが検量線に乗っていない。B)この図はプライマー二量体が生じた典型的な反応。この場合、低濃度のサンプルでCqの増加は認められない。

図11.18C.高濃度のテンプレートを含むサンプルのデータポイントが検量線に乗っていない。これは、高濃度のテンプレートによって阻害された反応、またはベースラインの不適切な設定による反応の典型。
希釈のダイナミックレンジにおける反応効率の測定にも、検量線を使用します。効率の算出に用いるすべてのポイントが線上に乗っているように注意してください。反応効率ができるだけ100%に近い反応になるようにし、明らかに効率が高いもの(>110%)や、低いもの(<85%)も検討してください。

図11.19A.テンプレートの核酸を10倍段階希釈した。増幅プロットの勾配が異常に小さく、ΔCqが期待値の3.3ではなく4サイクル。

図11.19B.ターゲットの量に対してCqをプロットした検量線の勾配を用いて反応効率を計算する。

図11.20A.テンプレートの核酸を10倍段階希釈した。増幅プロット間のΔCqが3.3ではなく1.5サイクルである。

図11.20B.ターゲットの量に対してCqをプロットした検量線の勾配を用いて反応効率を計算した結果、140%に近い値であった。

図11.20C融解曲線プロファイルを検討した結果、低濃度サンプル(黄色および青色)も、増幅したプライマー二量体からのシグナル(低Tmにピーク)を含むことがわかった。
トラブルシューティング:RT-PCRのケーススタディ
プローブアッセイの失敗
ヒトcDNAサンプル中のEIFB1を検出するが、増幅を示さないプローブアッセイを設計しました。初回反応は、ABi StepOne装置にて適合試薬を用いて行いました。200 nM~900 nMの濃度範囲でプライマーの最適化を試みましたが(図11.21)、改善が認められませんでした。アッセイのデザインを検証し、ターゲットに適していることを確認しました。in silicoでは、質の高いアッセイ法であると予測されました。新しいプライマーを合成し、別のオペレーターがSYBR® Green I試薬(異なる試薬)と装置(エッペンドルフRealplex)を使用し、初回で合成したプライマーとともに反応にかけました(図11.22)。このアプローチを採用するにあたって、主目的は問題の解決であり、副次目的は失敗の説明であることを念頭に置きました。この反応で、両バッチのプライマーから得られた増幅は同程度でした。この段階で、反応の問題はプローブにあると考えられたため、新しいプローブを合成し、2人目のオペレーターがRealplex装置でLuminoCt®試薬(初回反応と異なる試薬)を用いて両バッチを比較しました(図11.23)。両プローブから増幅データが得られましたが、新しいプローブは元のプローブよりも若干良いと考えられました。ただし、元のプローブは試験ラボから別の試験ラボに送られたことから、数日間室温の溶液中にあったことは考慮しなければなりません。この段階で、LuminoCt®試薬を用いて2番目のオペレーターがRealplex装置で操作すると、元のアッセイも再アッセイも機能することが明らかになりました。
よって、プローブ以外の初回の失敗の原因として以下が考えられました:
- オペレーター:実験は経験豊富な科学者が数回繰り返しており、理由として可能性が低いと考えられる。
- 装置:他のいくつかのアッセイで失敗が起きており、装置に問題があった可能性がある。
- 試薬:検証が最も容易な理由。第1オペレーターが、ABI StepOne装置で2つのオリゴバッチを用いて、LuminoCt®試薬を元の試薬と比較した。元の試薬では反応が失敗したが、LuminoCt®試薬では良好な増幅が得られた(図11.24)。

図11.21.EIFB1に対するプライマーを200~900 nMの濃度で試験した。どの条件下(ABi StepOne Plus装置でオリゴおよびABi試薬を変える)でも増幅が認められなかった。

図11.22.EIFB1に対するプライマーの2バッチ(初回の失敗バッチと新しいバッチ)をSYBR® Green I試薬を用いて比較した。(エッペンドルフRealplex装置でオリゴおよび試薬を変える)。どちらのプライマーセットでも増幅を裏付ける結果が得られた。

図11.23.EIFB1に対するプライマーおよびプローブの2バッチ(初回の失敗バッチと新しいバッチ)をLuminoCt®試薬を用いて比較した。(エッペンドルフRealplex装置でオリゴおよび試薬を変える)。どちらのオリゴセットでも増幅を裏付ける結果が得られた。

図11.24.EIFB1プライマー・プローブアッセイを2種類の試薬(元のABiまたはLuminoCt®試薬)で実施した。LuminoCt®試薬を用いた反応のみ、この分析法でデータが得られた。
反応効率が正しい値ではなく、変動する
標準的プライマーと標準的プローブアッセイを用いて、人工オリゴの段階希釈について試験を行いました。本アッセイは当初、別の装置で開発と最適化がなされましたが、別の試験ラボ・装置に移行したときに予期しない希釈効果は生じませんでした(図11.25A)。アッセイの全条件を新しい試験ラボに合わせて再最適化しましたが、データに変化はありませんでした。オペレーターは、同じ段階希釈液を用いて数時間以内にアッセイを繰り返した場合、希釈効果がより顕著であることを見出しました。トラブルシューティングの一環として、別のオペレーターが別の装置で分析を行ったところ、再度、予想した検量線が得られました。このことから、初回の問題は、オペレーターのミス、装置の故障、または実験手順でのいくつかの微妙な違いによることが示唆されました。両オペレーターとも経験豊富であり、別の実験で装置が良好に稼働していたため、他の微妙な違いについて検討しました。重要な手がかりは、サンプルを4℃で保存した後の同じ段階希釈液で得られたデータにばらつきが見られたことでした(図11.25A、図11.25B)。そこで、段階希釈液に用いた試験管を調べ、別の希釈液で再試験を行いました。段階希釈液を入れる反応チューブをエッペンドルフ(1.5mL)に変更した後、予測検量線を作成しました(図11.25C)。その結果、PCR法には分子生物学用のローリテンションプラスチック製品を選ぶことが大切で、このような分析法はプロトコルの微妙な違いにも感度が高いことがわかりました。

図11.25A.人工的テンプレートオリゴを10倍希釈し、特異的プローブアッセイにより検出した。増幅プロット間の差が一致しなかった。

図11.25B.人工的テンプレートオリゴを10倍希釈(図11.25Aで検出された希釈液)し、4℃で数時間放置した後、特異的プローブアッセイにより検出した。増幅プロット間の差は一致せず、希釈から試験までの時間が長くなるほど悪くなった。

図11.25C分子生物学グレードのチューブで人工的テンプレートオリゴを10倍希釈し、特異的プローブアッセイにより検出した。増幅プロット間の差が予想どおりに一致した。
要約-PCRトラブルシューティングチェックリスト
- サンプルの品質を確認する(劣化したサンプルでは間違った結果が出る)。
- RTプロトコルが分析法に適合していることを確認する(例えば、オリゴ(dT)プライマーを用いたRTは、配列の3'側配列1kbでqPCRを実施しなければならない)。
- 分析法の設計を確認する。
- すべてのコントロールを確認する。
- SYBR® Green I色素を用いてプライマーを確認/ゲル泳動する。
- ソフトウェアの設定(ベースライン、色素検出、標準物質の濃度)が正しいことを確認する。
- ROX濃度が装置に適している(マルチプレックスに干渉しない)ことを確認する。
- バックグラウンドの蛍光レベルを確認する。
- DNase Iアッセイによるプローブ標識の確認、またはプローブの再合成。
続きを確認するには、ログインするか、新規登録が必要です。
アカウントをお持ちではありませんか?