標的タンパク質分解
標的タンパク質分解は、疾患関連タンパク質を分解するために低分子を使って細胞内のタンパク質分解系をハイジャックするという新しい戦略です。細胞内のタンパク質含有量を減らすことで、ゲノム編集技術(CRISPR-Cas9など)と同様の表現型を得ることができますが、低分子を用いる利点も得られます。選択的プロテインノックダウンの影響を調べるための研究ツールとして関心を集めているだけでなく、占有率ベースの阻害にない利点があり、従来低分子では対処できなかった約80%のタンパク質を創薬可能(druggable)にする点で世界中の創薬研究者たちに急速に採用されています。これらのアプローチで使用される分子は、タンパク質分解誘導化合物(protein degrader)と呼ばれ、例えばタンパク質分解誘導キメラタンパク質(PROTAC®)やモレキュラーグルーなどがあります。
ヘテロ二官能性タンパク質分解誘導化合物は、一方の末端に標的に結合する分子弾頭(リガンド)を、他方の末端にE3ユビキチンリガーゼを標的とするリガンドを持ち、これらは真ん中にあるリンカーでつながれています(図1)。分解誘導化合物が添加されると、標的がE3リガーゼに集められます。E3リガーゼに接近した標的は、ポリユビキチン化され、それがプロテアソームによる分解の合図となります。1-3
*PROTAC®はArvinas Operations, Inc.,の登録商標であり、同社の許諾を得て使用しています。

図1.タンパク質分解誘導キメラタンパク質(PROTAC®)化合物による標的タンパク質分解
タンパク質分解誘導化合物合成の課題
タンパク質分解誘導化合物は、構造のわずかな変化により三元複合体形成やそれに続く分解が変わってしまうため、設計が困難です。4 PDB 5T3に基づく図2の3Dモデルでは、2つの異なるタンパク質(標的とE3リガーゼ)の結合を達成するための厳密な設計の重要性とタンパク質-タンパク質界面の確立を図解しています。計算科学は進歩していますが、タンパク質分解誘導化合物の設計はいまだに経験に大きく依存しています。研究者たちは分解誘導化合物のライブラリを作製して、モジュラーアプローチを用いてリガンド、リンカー、exit vectorをさまざまに組み合わせています。集中的な化学的取り組みが先行して行われているのです4,6。

図2.a) 標的リガンド、E3リガーゼリガンド、リンカー、exit vectorを強調表示した代表的なPROTAC® MZ1の化学構造。b) 分解誘導化合物の構造は三元複合体形成に影響を及ぼす。MZ1と共に図示6。 c) 経験に基づく設計では、個々の場合に応じて検討するライブラリの構築が必要。
ヘテロ二官能性タンパク質分解誘導化合物ライブラリの効率的合成
メルクのタンパク質分解誘導化合物ビルディングブロックは、タンパク質分解誘導化合物のヒット化合物発見を加速するための、1つの出発標的リガンドからヘテロ二官能性タンパク質分解誘導化合物スクリーニングライブラリを作製する最も簡単な方法です。タンパク質分解誘導化合物を構築するすべての成分を構成するこのビルディングブロックの集まりの中で、メルクのリガンド –リンカー複合体は、先行合成ステップを排除し、末端官能基に標的リガンドを結合させる化学反応だけが必要です(図3)。さらに、同じ末端基が選択される場合、50以上のリガンド –リンカー複合体を1つの出発標的リガンドと反応させると同時に、最初のスクリーニングライブラリを作製することができます。

図3.リガンド–リンカー複合体
リガンド –リンカー複合体の発見
メルクがご提供する一連のリガンド –リンカー複合体には、E3リガンド、exit vector、リンカー、末端基の戦略的組み合わせが含まれています。
E3リガーゼのリクルーターとリガンド:標的タンパク質分解のために研究されているE3リガーゼは増えている一方で、タンパク質分解誘導化合物の開発では、ほとんどの場合、少数のものしか使用されていません。7 メルクの複合体には、バリデーション済みE3リガーゼであるCRBN、VHL、IAP、およびRNF4のリガンドやさまざまなexit vectorが含まれています(図4)。
リンカー:アルキルリンカーとPEGリンカーは、幅広い疎水性、柔軟性、長さをサンプリングするのに最適なスターターです。また、お客様のライブラリで幅広いリンカー特性を得られるように、メルクでは多数の「ミックス」リンカー8 と「リジッド」リンカー9–11 を提供しています(図3)。
末端基:標的弾頭をつなぐためにさまざまな官能基を利用できます。メルクで最も多く提供しているのは末端がアミンのものです(図3)。
利点
- 合成時間の短縮:リガンド –リンカー複合体により、単一タンパク質分解誘導化合物の合成ならびにライブラリ構築用同時合成の時間が短縮されます。
- 分子デザイン:複合体内で関心度に基づいてE3リガンド、exit vector、リンカーをさまざまに並び替えることで、先行のコンビナトリアルライブラリの設計が簡単になります。
- 適合性:リンカーは標的リガンド上に存在する共通の官能基と結合します
- SAR:リガンド –リンカー複合体内に戦略的に組み込まれた成分変動から、情報に基づく最適化のためにSARを事前にある程度把握できます。
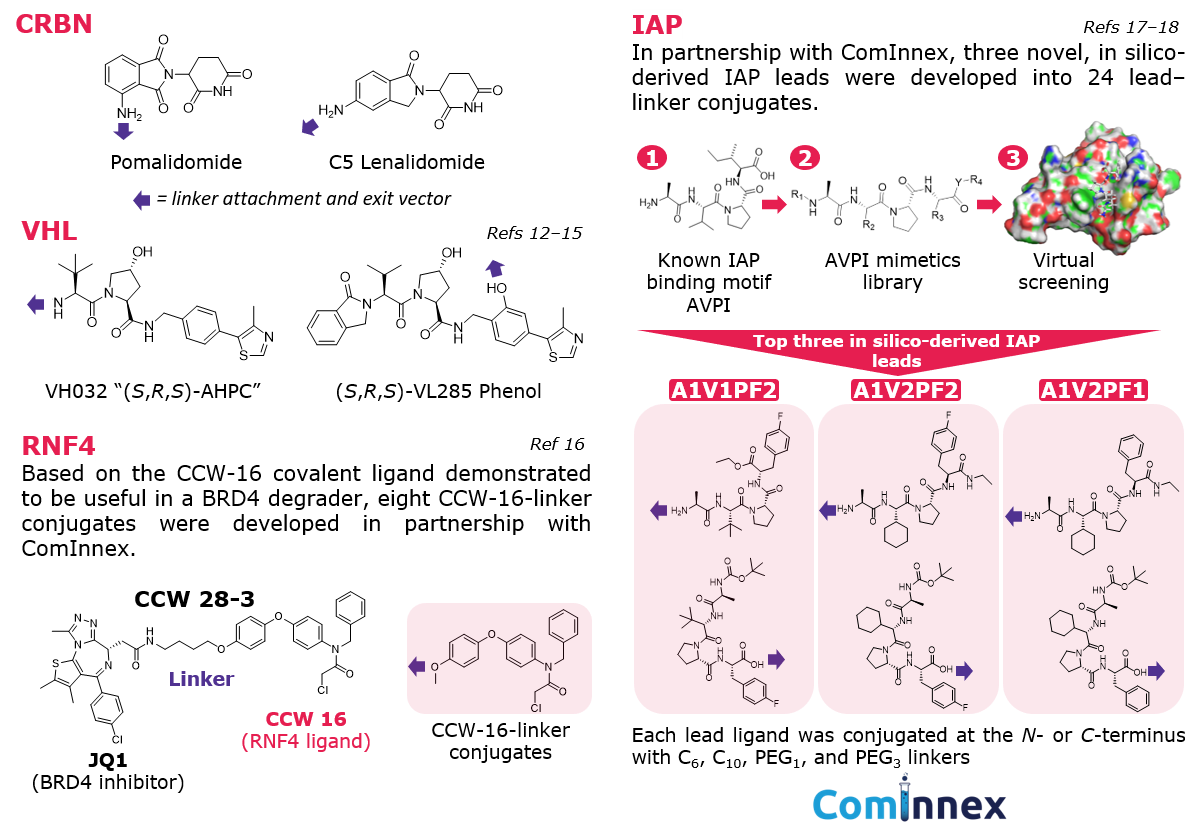
図4.複合体で使用されているE3リガーゼリガンド
お客様のニーズに基づくタンパク質分解誘導化合物ライブラリ構築のサポートをお任せください。
150を超えるリガンド–リンカー複合体、250を超えるヘテロ二官能性リンカー、20を超えるリガンド、そして関連プローブ化合物を含む全合成用製品の並び替え可能なリストや構造データファイルについては、担当のMerckテクニカルスペシャリストまたはSigmaAldrich.com/techserviceにお問い合わせください。
関連資料
- Degrader Building Blocks with IAP In Silico-Derived Ligands
- Accelerating IAP-Based Protein Degrader Discovery with Novel Ligand Library
ウェビナー
参考文献
続きを確認するには、ログインするか、新規登録が必要です。
アカウントをお持ちではありませんか?