Gestion des corps d'inclusion lors de l'expression de protéines recombinantes
Les protéines recombinantes sont habituellement exprimées dans l'espace intracellulaire, mais il est également possible de piloter leur expression pour qu'elles soient sécrétées dans l'espace périplasmique ou dans le milieu de culture. Si la sécrétion s'avère avantageuse en termes de repliement protéique, de solubilité et de formation de ponts disulfure, l'expression intracellulaire offre généralement un rendement beaucoup plus important.
Or, les protéines recombinantes qui s'accumulent à l'intérieur des cellules forment fréquemment des corps d'inclusion, agrégats insolubles de protéines incorrectement repliées dépourvues d'activité biologique. La protéine recombinante étant souvent le principal constituant des corps d'inclusion, la préparation de ces derniers peut être une étape de purification très importante. Toutefois, l'isolement des protéines contenues dans les corps d'inclusion pose fréquemment des problèmes de repliement et ne permet pas une restauration complète de l'activité biologique. Le tableau 10.1 récapitule les avantages et les inconvénients des produits recombinants exprimés sous forme de corps d'inclusion. Des corps d'inclusion se forment souvent lorsque des protéines eucaryotes sont exprimées chez des hôtes bactériens.
Plusieurs solutions sont envisageables lorsque la protéine est exprimée sous forme de corps d'inclusion : optimiser les conditions d'expression d'une forme soluble de la protéine, accepter la formation de corps d'inclusion mais élaborer des stratégies pour solubiliser et replier la protéine, essayer un autre hôte d'expression, ou modifier le plasmide recombinant. L'expression sous forme de corps d'inclusion permet d'exprimer des protéines qui sont toxiques pour la cellule hôte.
Optimisation de l'expression d'une forme soluble
On ne sait pas exactement pourquoi des corps d'inclusion se forment. On sait en revanche qu'une croissance plus lente aboutit généralement à l'expression d'une forme plus soluble, réduisant ainsi la tendance à la formation de corps d'inclusion.
Il est donc intéressant d'envisager quelques modifications simples des conditions de culture, destinées à réduire la vitesse de croissance et/ou le taux d'expression, afin de favoriser l'expression d'une forme soluble. L'inconvénient est que ces modifications risquent également de réduire le rendement global en protéine recombinante.
Il est possible de ralentir la vitesse de croissance en abaissant la température de croissance entre 20 °C et 30 °C.
Pour les protéines exprimées sous le contrôle d'un promoteur inductible, il est également possible de réduire leur taux d'expression en modifiant les conditions d'induction :
- induction à une plus faible densité cellulaire (A600 = 0,5)
- temps d'induction plus court
- induction avec une plus faible concentration d'agent inducteur (par ex. IPTG 0,1 mM)
Si cela se révèle insuffisant, des modifications de plus grande envergure peuvent être envisagées. Il peut s'agir de l'utilisation d'étiquettes de protéines de fusion, comme la glutathion-S-transférase (GST) et la protéine de liaison au maltose (MBP), qui semblent améliorer la solubilité (référence : Esposito, D. et Chatterjee, D. K. Enhancement of soluble protein expression through the use of fusion tags. Current Opinion Biotech, 2006, n o17, p. 353–358). Parmi les autres solutions envisageables figurent une coexpression avec des chaperonines ou d'autres composants de la machinerie de repliement, ainsi que l'utilisation d'un autre organisme hôte. La description exhaustive des techniques permettant de favoriser l'expression de protéines solubles sort du cadre du présent guide.
Solubilisation des corps d'inclusion
Si la modification des conditions de culture n'améliore pas significativement le rendement en protéines marquées solubles, des dénaturants courants tels que le chlorhydrate de guanidine (Gua-HCl) 4 à 6 M, l'urée 4 à 8 M, des détergents, un pH basique (> 9), des solvants organiques ou de la N-lauroylsarcosine peuvent être utilisés pour solubiliser les corps d'inclusion.
La réussite d'une purification par affinité en présence d'agents dénaturants dépendra de la nature de la protéine marquée. Il est important de tester le dénaturant choisi avec la protéine cible avant de l'introduire dans la stratégie de solubilisation.
Pour chaque dénaturant, la réussite de la solubilisation dépendra de la présence d'un agent réducteur, de sa concentration, de la durée de l'opération, de la température, de la force ionique et du rapport dénaturant/protéine. Le tableau 10.2 récapitule les conditions expérimentales de départ à respecter pour la solubilisation des corps d'inclusion. À ce stade, les protéines solubilisées sont souvent purifiées au moyen d'une technique de séparation compatible avec la présence du dénaturant. La purification et le repliement peuvent généralement être combinés en une même étape de purification, par exemple par repliement sur colonne chromatographique.
Plusieurs autres protocoles de solubilisation ont été publiés (par exemple, la base de données REFOLD). Parmi les solutions envisageables figurent l'utilisation de SDS (10 %), de N-lauroylsarcosine ou d'autres détergents, et l'emploi de pH extrêmes.
Repliement des protéines recombinantes solubilisées
Après solubilisation, les protéines doivent être correctement repliées pour retrouver leur fonction. Les agents dénaturants doivent toujours être éliminés pour permettre le repliement de la protéine et la formation des bonnes liaisons intramoléculaires. Lors du repliement, le pH, la présence de réactifs réducteurs (souvent un mélange des formes réduite et oxydée d'un réducteur faible, par ex. le glutathion), la vitesse d'élimination de l'agent dénaturant et la pureté de la protéine à replier font partie des paramètres critiques. Le tableau 10.3 compare les méthodes de repliement classiques à la purification par affinité après repliement sur colonne.
Le repliement nécessite généralement une optimisation poussée. Il convient de toujours envisager d'autres solutions (évoquées plus haut), par exemple l'optimisation des paramètres d'expression, la construction d'un nouveau plasmide ou l'utilisation d'un autre hôte d'expression.
Repliement sur colonne
La mise en œuvre d'une protéine portant une étiquette histidine permet d'utiliser une technique de purification après repliement sur colonne simple mais efficace, qui donne une protéine soluble présentant l'activité biologique recherchée. Le protocole illustré sur la figure 10.1 a été utilisé avec succès pour différentes protéines à étiquette histidine.

Figure 10.1 :Schéma général du protocole utilisé pour l'extraction, la solubilisation et le repliement de protéines à étiquette histidine (His6) produites sous forme de corps d'inclusion dans des cellules d'E. coli.
La forte liaison entre les protéines à étiquette histidine et les ions métalliques divalents immobilisés n'est pas affectée par des agents chaotropiques hautement concentrés (comme l'urée ou le chlorhydrate de guanidine). Les protéines à étiquette histidine (His6) peuvent donc être solubilisées par extraction chaotropique et fixées sur une colonne de Ni-Sepharose. L'élimination des protéines contaminantes et le repliement par échange en présence d'un tampon non dénaturant peuvent être réalisés avant l'élution de la protéine.
Une fois repliée, si une plus grande pureté est requise, la protéine peut être de nouveau purifiée par toute autre technique de chromatographie, comme n'importe quelle protéine native (voir chapitre 8, Purification des protéines marquées à la protéine A).
Analyse des protéines correctement repliées
Le tableau 10.4 récapitule différentes techniques utilisées pour déterminer si les protéines sont correctement repliées.
Application
Purification après repliement sur colonne d'une protéine à étiquette histidine insoluble à partir d'une culture d'E. coli de 100 ml au moyen d'une colonne HisTrap FF de 1 ml montée sur un système ÄKTAprime plus
Ce protocole utilise une colonne HisTrap FF de 1 ml, mais il peut également être réalisé avec une colonne HisTrap HP de 1 ml ou HisTrap FF Crude de 1 ml.
Préparation des tampons
Utiliser de l'eau et des produits chimiques ultrapurs et passer tous les tampons à travers un filtre de 0,45 µM avant emploi.
Autres tampons de liaison possibles : De l'imidazole 5–40 mM peut être inclus dans le tampon de liaison afin de limiter la liaison non spécifique des protéines ne portant pas d'étiquette histidine. La concentration de l'imidazole dépend de la protéine ; il convient de la réduire si la protéine d'intérêt est éluée ou si elle ne se lie pas à une concentration d'imidazole donnée.
Fractionnement, lavage et isolement des corps d'inclusion
- Remettre en suspension la pâte cellulaire issue des 100 ml de culture dans 4 ml de tampon de resuspension.
- Fractionner les cellules par sonication sur de la glace (par ex. 4 × 10 s).
- Centrifuger à haute vitesse pendant 10 minutes à 4 °C.
- Éliminer le surnageant et remettre le culot en suspension dans 3 ml de tampon d'isolement froid. Soniquer les cellules comme indiqué ci-dessus.
- Centrifuger à haute vitesse pendant 10 minutes à 4 °C.
- Répéter les étapes 4 et 5.
À ce stade, le culot peut être lavé une fois dans un tampon sans urée, puis placé au congélateur en vue d'un traitement ultérieur.
Solubilisation et préparation des échantillons
- Remettre le culot en suspension dans 5 ml de tampon de liaison.
- Placer sous agitation pendant 30 à 60 minutes à température ambiante.
- Centrifuger à haute vitesse pendant 15 minutes à 4 °C.
- Éliminer les particules résiduelles en passant l'échantillon à travers un filtre de 0,45 µM.
La concentration optimale de β-mercaptoéthanol (0–20 mM) doit être déterminée expérimentalement pour chaque protéine.
Si l'échantillon n'a pas été préparé conformément aux instructions précédentes, l'ajuster à la composition du tampon de liaison en le diluant dans le tampon de liaison ou en procédant à un échange de tampon sur colonne de dessalage (chapitre 11, Dessalage/échange de tampon et concentration), puis le passer à travers un filtre de 0,45 µM.
Préparation du système
Le choix d'un repliement et d'une élution en gradient linéaire nécessite l'utilisation d'un système de chromatographie.
Nous utilisons ici le système ÄKTAprime plus. Une fois le système préparé, les étapes restantes (voir Sélection de l'application type et lancement du procédé) s'exécutent automatiquement.
- Plonger chaque tubulure d'arrivée de la voie A (vanne 8 voies) dans les éluants conformément aux instructions précédentes, et la tubulure de la voie B (vanne 2 voies) dans le tampon d'élution.
- Plonger les trois tubulures de vidange marron dans les flacons de collecte de déchets.
- Monter la colonne entre la voie 1 de la vanne d'injection (vanne 7 voies) et la cellule à flux continu UV.
- Remplir le portoir du collecteur de fractions avec des tubes de 18 mm (au moins 40 tubes) et positionner la plaque blanche du bras de fractionnement contre le premier tube.
- Brancher une boucle d'injection suffisamment longue pour le volume de l'échantillon entre les voies 2 et 6 de la vanne d'injection. Remplir la boucle manuellement à l'aide d'une seringue.
Remarque : Si une boucle Superloop™ est nécessaire, prendre connaissance des informations complémentaires figurant dans la notice correspondante.
Sélection de l'application type et lancement du procédé
- Vérifier la communication avec le logiciel PrimeView. Le texte Controlled By: prime (Commandé par : prime) doit s'afficher dans le coin inférieur droit de l'écran.
- Utiliser les flèches et le bouton OK pour naviguer dans l'arborescence du menu jusqu'à trouver On-Column Refolding HisTrap (Repliement sur colonne HisTrap).

3. Indiquer le volume de l'échantillon et appuyer sur OK pour lancer l'application type.

Figure 10.2 :Gradient théorique dans une application type de repliement sur colonne HisTrap

Figure 10.3 :Repliement sur colonne d'une protéine portant une étiquette histidine.
Criblage des conditions de repliement par chromatographie d'affinité sur ions métalliques immobilisés
Les plaques à 96 puits MultiTrap sont utiles pour le criblage des conditions de repliement par chromatographie d'affinité sur ions métalliques immobilisés (IMAC) (tableau 10.5). Dans cet exemple, la protéine solubilisée et dépliée est répartie dans les puits d'une plaque His MultiTrap FF, avant d'être incubée dans différents tampons de repliement. Le degré de repliement est ensuite déterminé (figure 10.4). Les tampons ayant obtenu les meilleurs résultats lors d'expériences de criblage similaires peuvent être utilisés pour assurer la transposition d'échelle du repliement sur colonne HisTrap FF.
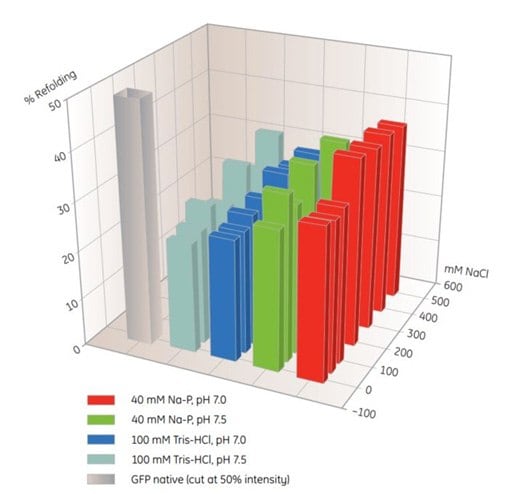
Figure 10.4 :Criblage par IMAC des conditions de repliement de la protéine fluorescente verte (GFP) portant une étiquette histidine au moyen d'une plaque His MultiTrap™ FF. Ce premier criblage portait sur les substances tampons, le pH et les concentrations en sels. Données aimablement fournies par J. Buchner, M. Haslbeck et T. Dashivets, Université technique de Munich (Allemagne).
Pour continuer à lire, veuillez vous connecter à votre compte ou en créer un.
Vous n'avez pas de compte ?