Kolonie-PCR
Die Kolonie-PCR ist eine einfache und schnelle Methode zur Überprüfung des korrekten Aufbaus eines klonierten DNA-Konstrukts. Traditionell war die Überprüfung der DNA-Insertion im Klonierungsvorgang zeitaufwändig und erfolgte im Anschluss an die Transformation von Bakterien mit einer mühsamen DNA-Aufreinigung und einem anschließenden Restriktionsenzym-Verdau. Die Kolonie-PCR ist eine direkte PCR-Methode, die sowohl die DNA-Isolierung als auch den Restriktionsverdau umgeht und eine schnelle, einfache und kostengünstige Lösung für das Screening klonierter Konstrukte bietet. Kolonie-PCR-Anwendungen sind auch mit dem Screening von Klonen kompatibel, die mit sequenz- und restriktionsunabhängigen Methoden erzeugt wurden.
REDExtract-N-Amp™ PCR ReadyMix™ und JumpStart™ REDTaq® PCR ReadyMix™ für Kolonie-PCR-Anwendungen
Geeignet für die Verwendung in Kolonie-PCR-Anwendungen bieten REDExtract-N-Amp™ PCR ReadyMix™ (Bestell- Nr. R4775) und JumpStart™ REDTaq® PCR ReadyMix™ (Bestell- Nr. P0982) beide Puffer, Polymerase, dNTP und Stabilisatoren als einzelne 2x-Lösung an. Sie sind so formuliert, dass sie eine direkte PCR ermöglichen und die Proben nach der PCR ohne zusätzliche Puffer oder Ladefarbstoffe auf ein Agarosegel geladen werden können. Der inerte Farbstoff migriert mit der gleichen Geschwindigkeit wie ein 125-Basenpaar-Fragment und bietet eine visuelle Bestätigung der DNA-Migration.
Leistungsmerkmale und Vorteile
- Ultimative Flexibilität: Eine DNA-Aufreinigung ist vor der Amplifikation nicht erforderlich. Geeignet für den Einsatz mit gängigen E. coli-Stämmen wie SIG10 5a, NovaBlue (K-12), BL21(DE3) und DH5α sowie mit S. pombe-Hefe. Die Probe kann auch erneut amplifiziert werden, wie z. B. bei der Nested (verschachtelten) PCR.
- Hohe Spezifität & Ausbeute: JumpStart™ Taq-DNA-Polymerase, ein durch Antikörper inaktiviertes Hot-Start-Enzym, wurde entwickelt, um eine unspezifische Amplifikation zu minimieren und gleichzeitig die Zielausbeute und Spezifität zu erhöhen. JumpStart™ Taq amplifiziert Ziele mit einer Länge bis zu 7 kb.
- Komfort: Die Hot-Start-Funktionen der JumpStart™ Produkte ermöglichen den Reaktionsaufbau bei Raumtemperatur, was bei der Arbeit mit mehreren Proben von Vorteil ist. REDExtract-N-Amp™ PCR ReadyMix™ und JumpStart™ REDTaq® PCR ReadyMix™ sind bei 4 °C oder -20 °C stabil.
- Inhibitor-Toleranz: JumpStart™ REDTaq® PCR ReadyMix™ verträgt nachweislich LB-Konzentrationen bis zu 20 % und LB-Agar oder Terrific Broth bis zu 10 %.
- Reproduzierbarkeit: Verringert das Risiko einer Kontamination durch mehrere Pipettierschritte und bietet eine gleichbleibende Leistung über mehrere Reaktionen.
- Gel-fertig: Es muss kein Ladepuffer oder Tracking-Farbstoff hinzugefügt werden. Das PCR-Produkt wird nach der Amplifikation direkt in ein Agarosegel geladen. Der rote Tracer migriert ungefähr mit der gleichen Geschwindigkeit wie ein 125-bp-Fragment (etwas schneller als Bromphenolblau).
Kolonie-PCR-Protokoll
Das Kolonie-PCR-Protokoll ist unkompliziert: Einfach eine kleine Menge einer Kolonie von einer Platte oder einer dichten bakteriellen Flüssigkultur in den PCR-Mastermix geben und mit dem Thermocycling fortfahren. Die Zelllyse erfolgt während der ersten Hochtemperatur-Inkubation. Die resultierenden PCR-Produkte können mit Gelelektrophorese oder anderen DNA-Nachweisverfahren analysiert werden. Diese zeitsparende Methode eignet sich hervorragend für das Screening weniger Kolonien oder für das Hochdurchsatz-Screening vieler Klone.
1. Den PCR-Mastermix vorbereiten (Größenordnung abhängig von der Anzahl der zu analysierenden Proben):
2. Den PCR-Mastermix (20 µl) in jedes PCR-Röhrchen oder jede Platte geben.
3. Mit einer sterilen Mikropipettenspitze oder einem sterilen Zahnstocher Zellen aus jeder Kolonie in ein PCR-Röhrchen überführen und kurz rühren, um sie im PCR-Mastermix zu resuspendieren. Die Mischung kann leicht trüb aussehen. Hinweis: Nicht zu viele Zellen auswählen. Überschüssige Zellen würden die PCR stören.
4. Die Ziel-DNA unter den folgenden Thermocycling-Bedingungen amplifizieren:
Hinweis: Der Schritt der Zelllyse ist länger als eine typische erste Denaturierung, damit die Bakterienwand effizient durchbrochen wird
Kolonie-PCR-Anwendungen mit Bakterien und Hefe
Die Kolonie-PCR wurde mit repräsentativen Bakterien- und Hefestämmen durchgeführt. E. coli-kompetente Zellen SIG10 5a (Bestell- Nr. CMC0001), NovaBlue (Bestell- Nr. 69825), BL21(DE3) (Bestell- Nr. 69450), und DH5α (Thermo Fisher Scientific) wurden mit pET28a(+) transformiert, das eine eingefügte Sequenz enthielt. SIG10 5a wurde darüber hinaus mit einem leeren pET28a(+)-Vektor transformiert. Die E. coli-Kolonien wurden mit REDExtract-N-Amp™ PCR ReadyMix™ und flankierenden Primern, die 1 kbp des Inserts amplifizieren, auf Inserts untersucht. Alle E. coli-Stämme waren mit der Kolonie-PCR kompatibel, wobei die Zielamplikons direkt aus den rohen Zell-Inputs ohne einen DNA-Extraktionsschritt erzeugt werden (Abbildung 1).

Abbildung 1Agarosegel der E. coli Kolonie-PCR-Produkte. Die Kolonie-PCR eines 1 kbp-Inserts wurde mit REDExtract-N-Amp™ PCR ReadyMix™ durchgeführt und auf einem 1 % Agarosegel analysiert. In allen transformierten Zellen, die pET28a(+) mit Inserts enthielten (Proben 3-10), wurden Produkte nachgewiesen, während in Zellen, die mit dem leeren Vektor pET28a(+) transformiert waren (Proben 1-2), kein Produkt amplifiziert wurde.
Die Hefe Schizosaccharomyces pombe wurde mit pESP-1 transformiert. An den entstandenen Kolonien und der Plasmidkontrolle wurde eine PCR durchgeführt. Die Hefekolonien wurden mit JumpStart™ REDTaq® PCR ReadyMix™ und flankierenden Primern, die 226 bp des Inserts amplifizieren, auf Inserts untersucht. Wie bei E. coli wurden die Zielsequenzen aus Hefeproben ohne DNA-Aufreinigung amplifiziert (Abbildung 2).

Abbildung 2Agarosegel der S. pombe Kolonie-PCR-Produkte. Die Kolonie-PCR eines 226 bp-Inserts wurde mit dem JumpStart™ REDTaq® PCR ReadyMix™ durchgeführt und auf einem 1 % Agarosegel analysiert. Die Produkte wurden in allen transformierten Zellen (Kolonie 1-3) und dem Plasmid-Template (Kontrolle) nachgewiesen.
Auswirkung des Wachstumsmediums auf die PCR-Ausbeute mit JumpStart™ REDTaq® PCR ReadyMix™ Reagenzien
Um die Auswirkungen der Kontamination von Wachstumsmedien auf die PCR zu quantifizieren, wurden die gängigen E. coli-Medien LB, LB-Agar und Terrific Broth (TB) mit JumpStart™ REDTaq® PCR ReadyMix™ Reagenzien dotiert. Die Amplifikation wurde mit pBR322 als Template, geeigneten Primern, α [32P] dCTP und unterschiedlichen Mengen (0 - 50 %) LB, LB-Agar und TB durchgeführt. Die PCR-Ausbeute wurde nach Ausfällung mit Trichloressigsäure auf Glasfiltern und anschließender Szintillationszählung bestimmt. Die Ausbeuten wurden auf 0 % zugesetzte Medien normalisiert. Die Reaktionen wurden in dreifacher Ausführung durchgeführt. Alle Medienzusätze wirkten sich auf die Produktausbeute aus, aber die PCR mit JumpStart™ REDTaq® PCR ReadyMix™ war auch dann noch robust, wenn bis zu 20 % der Reaktionsmischung aus LB und bis zu 10 % der Reaktionsmischung aus TB oder LB-Agar bestanden (Abbildung 3). Diese Werte liegen über dem, was eine typische Kolonie-PCR kontaminieren würde, und zeigen die hohe Inhibitortoleranz der JumpStart™ REDTaq® PCR ReadyMix™ Reagenzien.
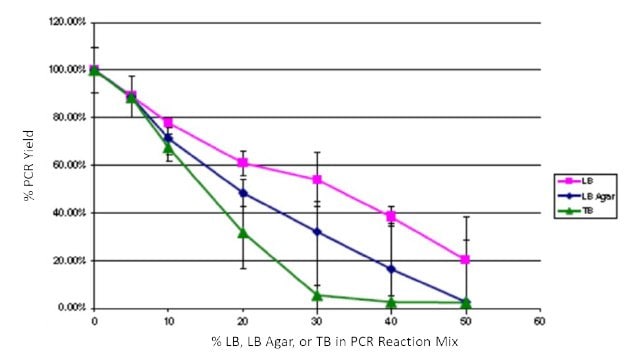
Abbildung 3Auswirkungen der Dotierung von LB, LB-Agar und TB in PCR ReadyMix™ Reagenzien. LB, rosa Quadrate; LB-Agar, blaue Rauten; TB, grüne Dreiecke.
Kolonie-PCR-Leistung mit JumpStartTM und REDTaq® ReadyMixTM Reagenzien
REDExtract-N-Amp™ PCR ReadyMix™, JumpStart™ REDTaq® PCR ReadyMix™ und andere JumpStart™ und REDTaq® ReadyMix™ Produkte wie R2523 und P1107 sind robuste Mastermischungen, die eine direkte PCR von Kolonien und Kulturen gängiger Klonierungs- und Expressionsstämme, einschließlich E. coli und S. pombe, ermöglichen und das schnelle Screening geklonter Konstrukte erleichtern. Sie weisen eine hohe Toleranz gegenüber gängigen PCR-Inhibitoren auf, darunter LB-Konzentrationen bis zu 20 % und LB-Agar oder Terrific Broth bis zu 10 %, was zu einer hohen Leistung bei der Kolonie-PCR führt. Die Mastermischungen können die Auswirkungen von Zelltrümmern überwinden und können direkt mit rohen Kolonien oder Suspensionen für die Amplifikation von Zielen mit einer Länge bis zu 7kb verwendet werden. Weitere ultraschnelle PCR-Master-Mischungen, darunter unser KOD One™ PCR Master Mix, sind ebenfalls erhältlich und für Kolonie-PCR-Anwendungen geeignet. Weitere PCR-Reagenzien und -Informationen finden Sie in unserem Informationszentrum zu PCR-Reagenzien und -Kits.
Um weiterzulesen, melden Sie sich bitte an oder erstellen ein Konto.
Sie haben kein Konto?